-
光合作用反应是地球上重要且高效的反应, 它由镶嵌在类囊体膜上的两个巨大色素蛋白质复合体-光系统II (photosystem II, PSII)和光系统I (photosystem I, PSI) 所驱动. 两个光系统的动态激发平衡依赖于其外围的捕光蛋白. 当环境光发生变化时, 为了维持光系统间的激发平衡, 光系统周围的捕光蛋白的数量不仅可被调节, 而且其功能也发生变化[1–9]. 捕光蛋白的功能异质性主要起源于其结构的异质性[10–19]. 蛋白质的结构变化可由多阶层的能量地形(energy landscape)理论描述. 在该理论中, 蛋白质的结构可考虑为被明确的能量势垒所分割的构象子态, 每个状态都对应着明确的原子坐标[20,21], 因此色素蛋白质复合体的运动可考虑为各个构象子态间的跃迁或转换, 运动频率取决于系统温度与时间. 生物学方法很难表征如此复杂的蛋白质动态. 低温下, 蛋白质运动的自由度受限, 因此冷冻单分子光谱技术能通过镶嵌在蛋白质骨架中的色素光谱来表征独立的构象子态[22]. 在自然光合作用研究领域中, 该技术在表征蛋白质动态, 色素的电子结构, 复合体的功能状态等方面发挥着重要作用[11–13,15,23–34].
另一方面, 围绕PSII和PSI所发生的主要生理事件不同, 例如光捕获、能量调节机制、光保护机制和电子转移等[2,5,6,35–53]. 由于缺少能在微观空间内明确区分两个光系统的方法, 因此独立探究这些事件变得困难. 冷冻光谱显微镜具有极高的光谱辨识度, 根据光谱特征可精确区分PSII和PSI[54], 使得在单个细胞内可视化两个光系统的空间分布成为可能[55,56]. 另外, 多种光谱手段与冷冻光学显微镜的结合扩展了多维度研究光合作用的可能性. 该技术的应用不仅促进了在微观尺度下探索光合作用的发展, 而且还建立了基于光谱手段追踪光合物质的重要技术框架, 该框架可用于探究光系统在发育过程中的细胞体内组装机制[57–61].
本文旨在综合地概述冷冻光谱显微镜的在光合作用领域中的发展与应用. 第2节简述了色素蛋白复合体的结构基础; 第3节详细描述了冷冻光谱显微镜的光学搭建与技术细节, 以及数据分析方法; 第4节和第5节分别介绍了单分子光谱与单细胞光谱解析的例子; 第6节阐述了对该领域的总结与展望.
-
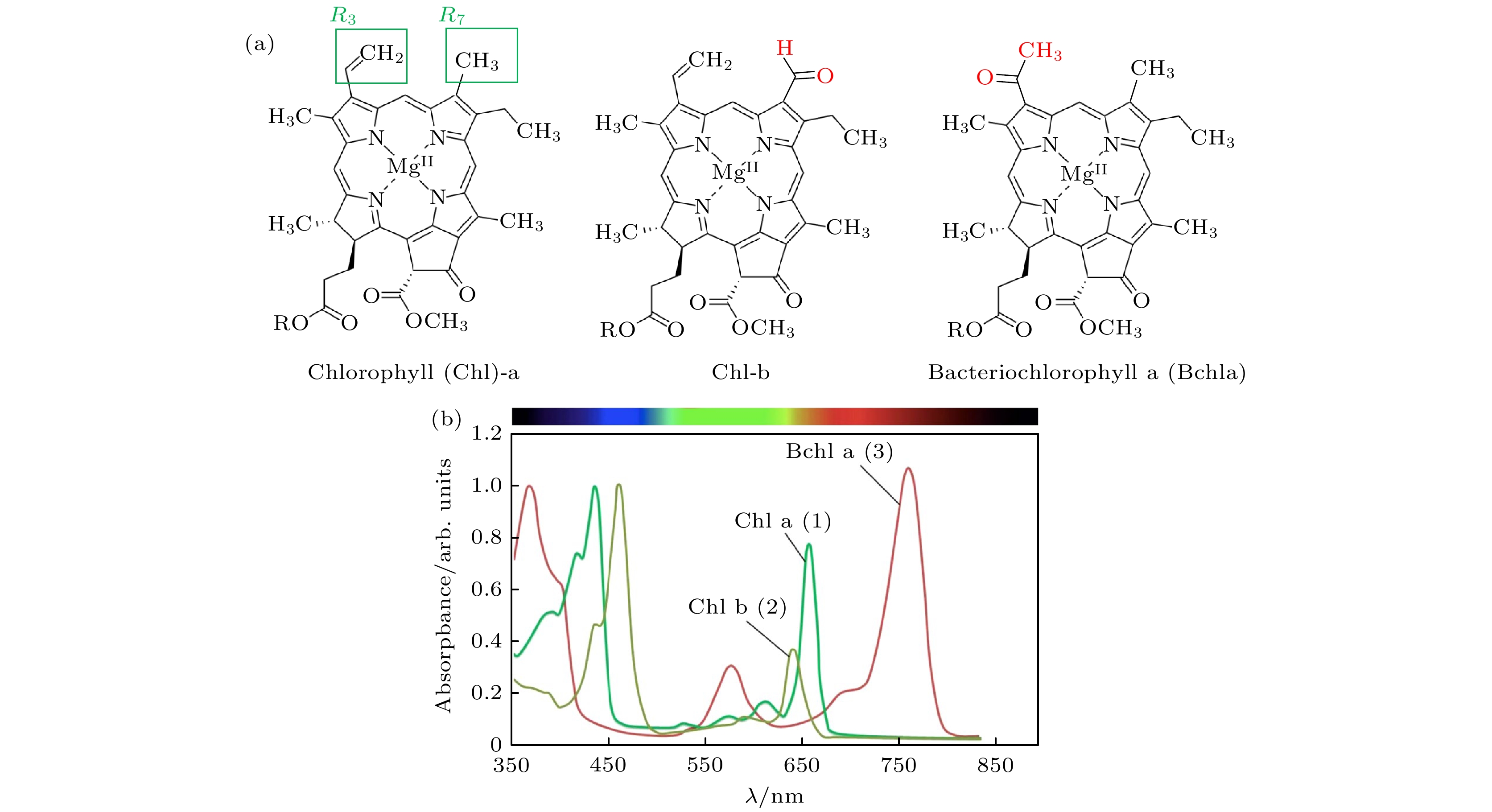
光合作用中高效的光电转化效率是由于色素分子强大的光捕获能力与分子间能量转移的多样性[62]. 叶绿素a (chlorophyll a, Chl-a)、叶绿素b (chlorophyll b, Chl-b)和细菌叶绿素(bacteriochlorophyll a, BChla)是光合生物中最基础的捕光色素分子. Chl-a的分子结构由一个连接了镁原子的卟啉环和一个疏水性的碳氢化合物长链所构成(图1(a)左)[63]. Chl-b和BChla的分子结构类似于Chl-a, 不同之处在于它们的卟啉环上R7和R3官能团的差异. Chl-a和Chl-b分子广泛存在于高等植物和真核藻类细胞中, 而BChla普遍存在于光合细菌中(图1(a)右). Chl-a溶液在大约430 nm (Soret带)和665 nm (Qy带)处有较强的吸收峰, 而Chl-b在这两个波段的吸收峰迁移至460 nm和645 nm (图1(b))[63]. 此外, 在Chl-a和Chl-b的Qy吸收带的短波长侧能够观察到一些强度较低的吸收峰, 它们被称为Qx带[62,64]. BChla的Soret带位于360 nm, 而Qx和Qy吸收带分别位于580 nm和772 nm[62,65]. 各色素分子在吸收光谱上的差异主要源于卟啉环上共轭电子体系的改变. 另一类重要的色素分子是可吸收黄绿光的类胡萝卜素(carotenoid, Car). 由于其极短的荧光寿命(< 10 ps)[66], 该类分子可充当一个理想的能量淬灭体, 许多研究表明该分子在光能量保护机制中可能起到重要作用[67].
-
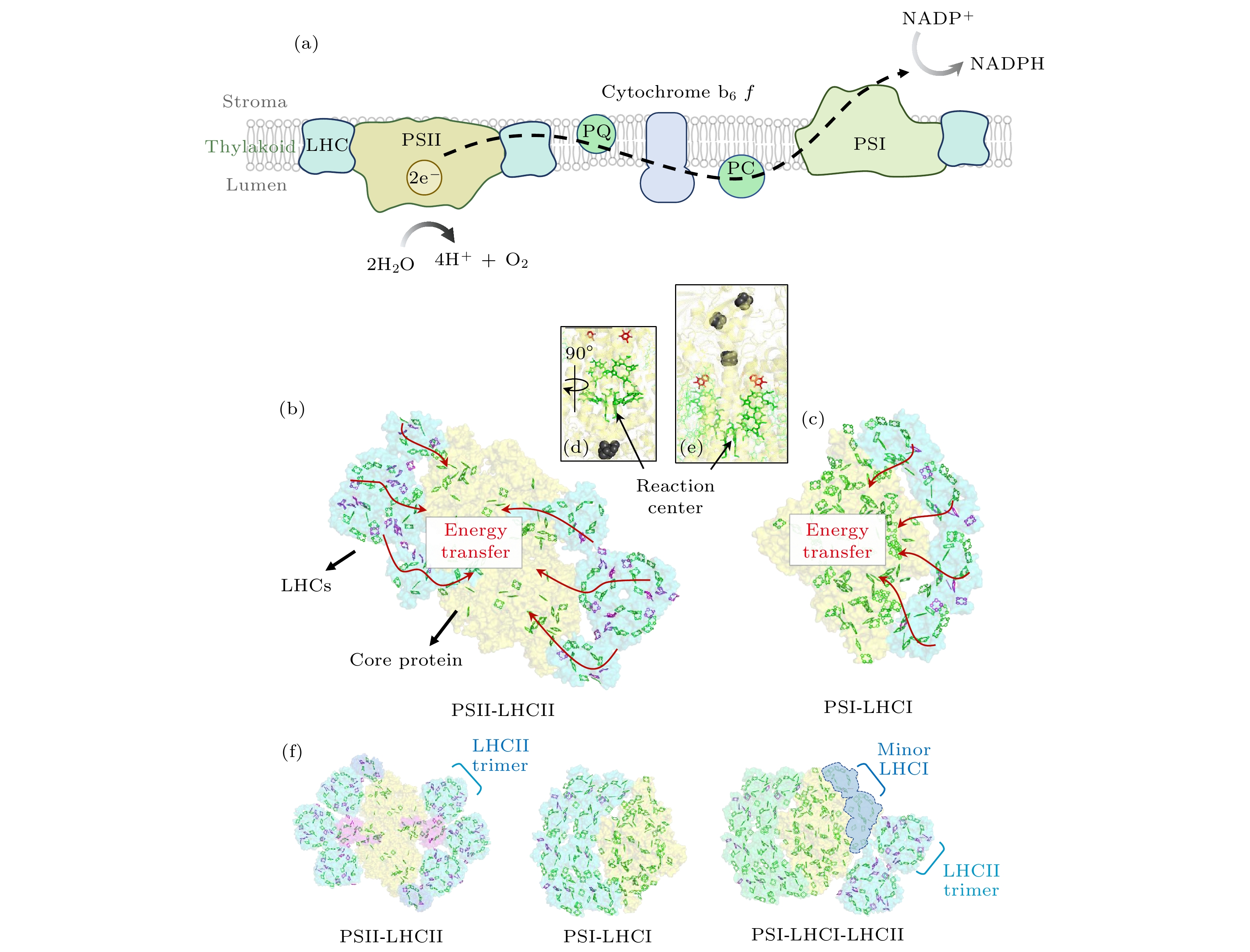
如图2(a)所示, 光合作用反应由类囊体膜中两大色素蛋白复合体的协同合作所驱动, 它们分别称为光系统II (PSII)和光系统I (PSI)[68]. 被光激发后的PSII可分解水并获取电子, 电子被一系列转运中间体运输至PSI并用以还原烟酰胺腺嘌呤二核苷酸磷酸(nicotinamide adenine dinucleotide phosphate, NADPH), 即辅酶II. NADPH是CO2固定反应中所需的重要物质. 图2(b)—(e)展示了植物中光系统的分子结构. 由该结构可知, 光系统由核心蛋白和外周捕光天线蛋白所构成. 天线蛋白结合了Chl-a和Chl-b, 但核心蛋白仅含Chl-a, 因此, 能量可快速地从含激发能较高的Chl-b的天线蛋白流向核心蛋白的反应中心分子, 这是执行电荷分离反应的场所(图2(d), (e)). PSII和PSI的反应中心分子分别导致了吸收光谱上680 nm和700 nm处的光致变化, 因此也被简称为P680和P700.
高效的光合作用反应依赖于两个光系统的动态激发平衡, 然而, 环境光的实时变化让两个光系统面临着被过度激发的风险. 为了应对环境的扰动, 光系统可通过改变其连接的捕光蛋白数量来调节激发水平. 例如在衣藻细胞Chlamydomonas reinhardtii中, 当PSII被过度激发时, PSI会结合更多的捕光蛋白以提高激发水平, 这涉及到被称为状态转换(state transition, ST)的能量调控机制. 衣藻细胞常被用于研究ST机制, 这是因为相对于植物细胞而言, 衣藻细胞中参与到ST机制的捕光天线蛋白LHCs (light-harvesting complexes)的数量更多[3,42]. 超级复合体PSI-LHCI-LHCII的成功发现是证实ST机制存在的一个有力证据[69] (图2(f)). 最近, 微观尺度下的ST机制已通过冷冻光谱显微镜被系统性地研究(参见5.2节).
-
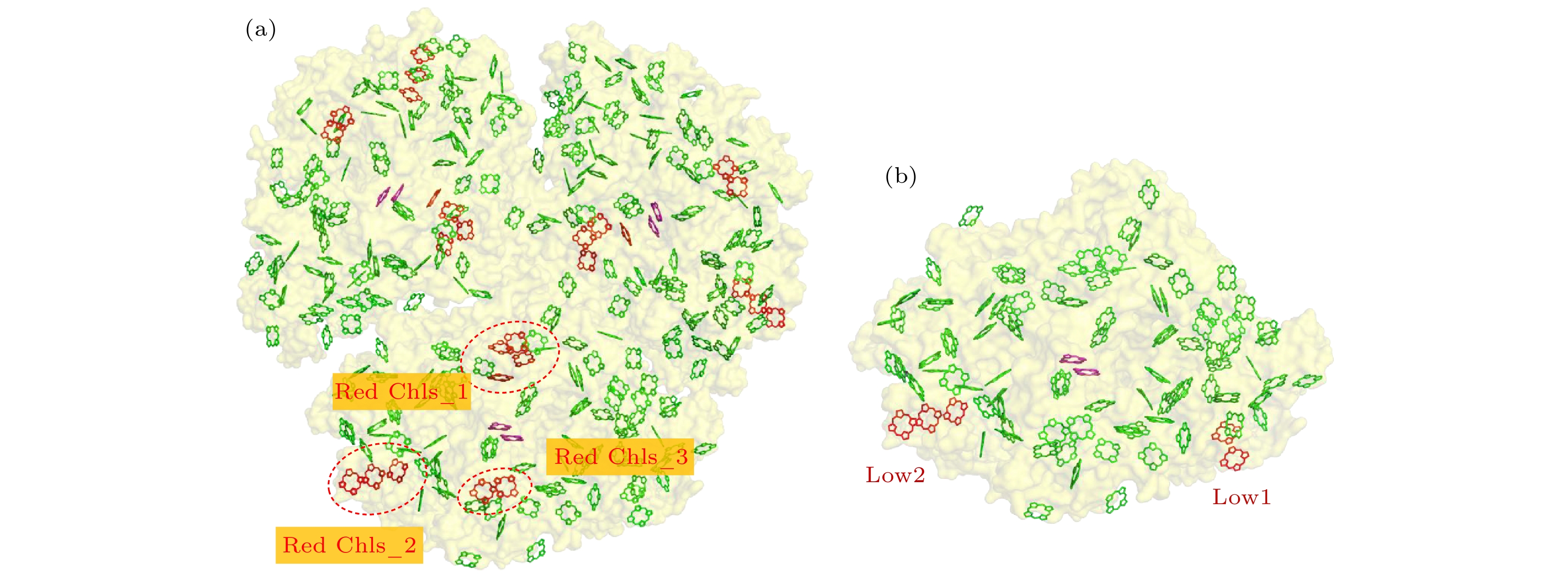
图3(a)展示了耐热型蓝细菌Thermosynechococcus (T.) elongatus的PSI三聚体的晶体结构[70], 该结构中的PSI单体含有约96 个Chl-a分子(不含Chl-b), 除了反应中心P700以外的所有分子被统称为天线系统. PSI的天线系统中存在少量具有低激发能的Chl, 它们的吸收光谱会发生较大红移, 因此被称为红叶绿素(red chlorophyll, Red Chl). PSI的红移荧光被认为是源自于点位能量(site energy)发生变化的Chl多聚体. 室温下, Red Chl组成的分子团与P700间发生的uphill能量转移(uphill energy transfer)严重限制了PSI的荧光发射. Uphill能量转移机制描述了激发能量从激发能较低的分子被向上传递给激发能较高的分子的过程, 其发生的概率取决于系统温度. Uphill能量转移是PSI的一个重要光学特征. 低温下, uphill能量转移会受到抑制, 这让PSI中Red Chls的荧光增强, 因此PSI的荧光更容易被探测到. 蛋白质的结构动态可能通过两种方式来影响Chl的点位能量: i) 改变多聚体中单个Chl的点位能量; ii) 直接改变整个多聚体的点位能量. 对于前者, 影响Chl所在位置的多个因素须被考虑, 例如电荷分布, 与蛋白质残基的结合点位, 分子本身结构的变形等; 而后者主要被多聚体中Chl分子间耦合强度的变化所影响[12,71–73]. 然而, 天线系统中Red Chls的归属问题存在长期争议[11,25,26,31,74–81], 早期许多关于PSI的单分子光谱实验都是围绕着这个问题来开展的.
基于最初的蛋白质结构[70], 3个可能的Red Chls被提出(图3(a)). Red Chls_1和Red Chls_3分别由两个和一个Chl二聚体组成. Red Chls_2由一个Chl三聚体组成, 并且3个色素分子几乎彼此平行且呈阶梯状排布, 该结构容易导致发射光谱出现明显的红移. 因此, Red Chl_2是最受关注的候选者. 最近, Çoruh等[81]成功解析了单体化PSI的结构. 单体化PSI与三聚体中的PSI单体相比, 缺少了13个分布在蛋白质外围的天线分子, 其中Red Chls_2结构中最外端的一个分子是缺失的. 单体和三聚体的稳态发射光谱表明, 单体化PSI的发射峰出现明显的蓝移, 表明了红发射状态的减少. 这进一步证明了Red Chls_2是其中一个红发射源的合适候选者.
另一方面, Kato等[82]也解析了来自蓝藻Gloeobacter violaceus PCC 7421的PSI结构, 该结构不含Red Chls. 通过将该结构与之前报道的含有Red Chl的PSI结构进行比较后[70,83], 证实了来自于Chl二聚体和三聚体的两个红发射荧光仅出现在含有Red Chl的PSI中. 稳态光谱的测试结果表示, 该Chl二聚体和三聚体分别在大约700—725 nm和725—752 nm波长区域有较强发射. 这两个Red Chls被命名为Low1和Low2. Low2与之前提议的Red Chl_2一致, 但Low1是之前没有被预测过的新候选者(图3(b)). 需要注意的是, 由于Low1和Low2的光谱分布较宽, 这导致它们之间存在光谱重叠. 因此, 天线系统的能量传递有利于两个发射池中的哪一个仍然难以确定. 此外, 组合了光谱烧孔和定点基因突变技术的实验确认了Red Chls的吸收状态, 例如: T. elongatus的PSI主要有C708, C715和C719, Synechocystis sp. PCC6803的PSI有C706和C714等(C后面的数字代表吸收峰的波长)[31,80]. 然而, 阐明各个吸收状态所对应的分子数量和位置仍然需要被持续地研究.
-
通常, 在冷冻光谱显微镜中, 液氮或液氦可作为冷冻剂将样品冷却至约77 K或1.2—4.0 K. 处于冷冻状态的生物样品可承受高强度, 高频激光的扫描, 这抑制了在光谱测量中可能会产生的荧光漂白, 为长时间观察样品荧光的变化提供了保障. 在自然光合作用研究中, 低温测量至少具有以下几个优势: 第一, 可以抑制红移(或低激发能)天线分子与反应中心间的uphill能量转移, 从而提高素色分子的荧光量子收率. 对于含红移较大色素的光合蛋白的荧光探测来说降低体系温度尤为重要, 例如PSI (参见4.4.2节). 第二, 低温下产氧型光合生物的荧光光谱中PSII和PSI的特征发射成分变得明显, 它们分别在682—695 nm和710—760 nm (取决于生物种类)的波长区域有容易被识别的发射峰(参见4.1节). 第三, 低温可以冻结细胞的生理状态. 因此, 冷冻光谱显微镜可在细胞发育过程中探测光合蛋白质的中间组装体, 并发掘新的色素蛋白质组分(参见5.3节). 第四, 基于荧光去激发原理的一种超分辨成像技术被提议可与共聚焦光谱显微镜相结合, 用以发展超分辨的光谱成像技术[84]. 然而, 由于光合生物中含多种光谱重叠的物质, 导致各物质的荧光激发与发射难以被统一调控, 因此严重地限制了该超分辨方案在光合作用研究中的开展. 低温可改变分子的激发方式[85], 因此冷冻光谱显微镜有潜力促进光合作用研究中超分辨显微光谱学的开展[68] (参见第6节).
-
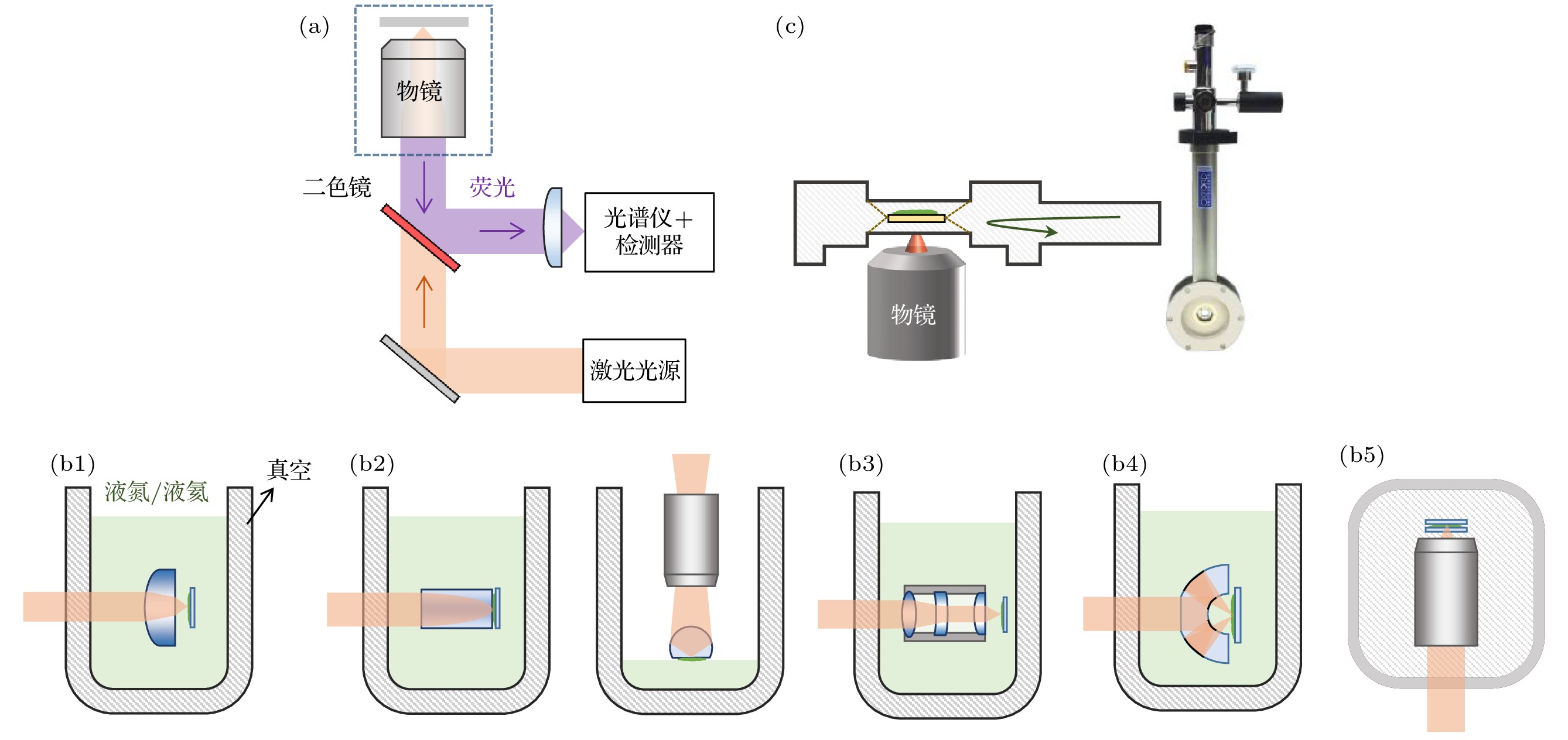
在冷冻显微镜系统中, 样品与用于聚光的光学组件(商业化物镜或组装的透镜等)间必须保持真空环境, 这是为了保护光学组件不被低温破坏和避免样品表面形成水雾层; 因此, 样品与聚光组件通常被设计在一个真空腔里(图4(a)). 目前已发展出各种类型的自制冷冻光学方案如图4(b)所示[68]. 图4(b1)为浸入冷冻剂中的单组件透镜[86]; 图4(b2)左图为折射率梯度聚光类型[87]; 图4(b3)为可安装在真空腔内的改良版商用物镜[88]; 图4(b4)为由熔融石英制作的反射物镜[88,89]等. 在这些方法中, 折射率梯度型冷冻系统是最初应用于叶绿体低温成像的方案[90]. 另外, 少数几家公司提供与显微镜架相兼容的商用低温恒温器, 如Oxford Instruments, Linkam Scientific Instruments等[91–95] (图4(a), (c)). 虽然冷冻显微镜测试可通过直接使用商业化设备来实现, 但该方法仅允许使用长工作距离的物镜, 这使得数值孔径(numerical aperture, NA)被限制在一个较小的数值.
在低温聚光系统的自制方案中, 单组件透镜的方案是较容易实施的(图4(b1), (b4)), 但该方法不能修正色差. Fujiyoshi等[89]扩展了基于单组件Cassegrain反射镜的光学性能, 他们调整光线的入射和出射表面至几乎相互垂直的状态, 这显著性地减少了色差. 他们的最新版本已将镜物的NA提升至0.93[96]. 2000年, Yoshimoto[97]发展了一种将整个承载了物镜的扫描台放置于绝热真空腔内的方案 (图4(c5)); 这种设计是为了缩短物镜与样品间的距离, 以获得更高得NA. 2014年, Shibata等[98]以相似的概念开发了一个冷冻显微镜系统, 该系统使用一个与真空相兼容的物镜 (NA = 0.9), 可在80 K下对光合细胞进行高分辨的发射光谱成像. 需注意地是, 他们设计的真空腔搭配了两个可从仓外调控样品位置的水平操控杆, 该方法极大地拓宽了可测量的范围. 最近, 他们将同样的方案实施在一个高速激发光谱显微镜上[34].
研究者们还考虑通过低温下的液体浸没法来提高NA. Metzger等[99]首次使用1-丙醇作为低温浸没液, 在160 K下成功使用了NA为1.25的物镜进行成像. 与使用NA = 0.85的干物镜所测到的结果相比, 该方法可将空间分辨率提高1.3倍, 但此方法很难适用于观察嵌入玻璃化水中的生物样品. 最近, Wang等[100]证明, 通过固体浸没效应可提高冷冻显微镜的分辨率和光收集效率(图4(b2)右). 他们使用了一种由二氧化锆(折射率约为2.17)制成的小型超半球形固体浸没透镜, 将样品贴在该透镜下方, 然后使其直接与冷冻剂接触, 并在该特殊透镜上方设置了一个干物镜(NA = 0.55)来收集荧光, 该系统让有效NA被提高至2.17. 他们基于随机光学重构显微镜(超分辨成像技术的一种)在液氮温度下实现了大肠杆菌细胞的双色成像, 并显著地提高分辨率至12 nm. 但遗憾的是, 由于固体浸没型物镜存在无法补偿色差的缺点, 因此目前很难实现高光谱成像.
在冷冻光谱显微镜中, 冷却时间缓慢(约几十分钟)可能会导致结晶水的形成, 这将破坏细胞内部的精细结构. 最近, 一些研究小组在缩短冷却时间方面取得进展. Huebinger等[101]将生物样品附着在人造金刚石片下表面, 然后使其背面(人造金刚石片上表面)直接暴露在液氮/液氦混合物的强烈流动中. 由于该材料具有很好的导热性, 因此可在几秒内完成样品冷却步骤. Wang等[100]使用类似的方法, 他们通过将附着在固体浸没透镜上的样品直接浸入冷冻剂中以实现快速冷却. Yamanaka等[102]设计了一个特殊的显微镜平台, 样品和物镜之间使用氮气填充, 然后在样品上方直接淋入液氮使其快速冷却. 以上3种方法在样品与聚光组件之间均未做高真空处理, 因此样品中可能会出现水雾层. 水雾层所造成的光散射会严重地影响依赖于激发光形状的光谱的精确测量, 例如吸收光谱和激发光谱.
-
吸收光谱是最基本的光谱信号, 可探测分子的电子基态. 当已知分子的吸光系数时, 吸收光谱的强度可直接估计分子的绝对浓度(表1). 目前, 单像素的吸收光谱显微探测已经被实现, 这是通过计算样品所在区域和空白区域对探测波段波长的吸收强度变化来完成的[103]. 由于吸收光谱显微镜本质上是测量吸光度的差值, 因此主要用于研究单细胞光合生物, 单个色素蛋白分子的吸收光谱探测目前还尚未实现. 另一方面, 荧光的激发光谱是一种荧光强度取决于激发波长的光谱, 可提供等价于吸收光谱的信息. 许多工作使用单分子激发光谱来研究单个色素蛋白的电子结构[13,24,104]和能量传递路径等问题[16,34]. 最近的研究证实了激发光谱显微镜对光合细胞中的天线分子非常敏感, 可用于研究与天线分子相关的能量调节机制[48,49,105].
荧光发射光谱是最早用于低温显微镜的信号[23] (表1). 低温下, 光合细胞的发射光谱出现分离的PSII和PSI特征荧光带[55,56]. 在使用标准荧光物质作为参照的情况下, 荧光发射带的强度变化还可用于表征能量淬灭[106]. 这里想要强调, 室温下区分PSII和PSI的方法也被Hasegawa等[103]提出, 该方案通过用近红外波长的连续激光对光合细胞进行反斯塔克激发来实现. 发射光谱也常用作于单分子光谱探测的信号来源, 研究与色素蛋白质的结构动态相关的问题[17,29,107]. 荧光寿命光谱可检测发射分子的激发态动力学和定量物质的量子收率, 这对表征淬灭机制十分重要[5,108,109]. 最近, 低温荧光寿命显微镜已被实现, 它被用于研究衣藻细胞体内淬灭态的LHCII[56].
相对于Chl的显著性自发荧光而言, Car色素易发生分子内部的能量转换并容易被淬灭, 导致它几乎不发荧光. Car的拉曼散射信号能够被检测, 并可通过特征性的分子振动模式来区分具体的分子种类, 但缺点是信号强度极低[110]. 在实际的实验中, Car的自发拉曼信号会被Chl的自发荧光信号所覆盖, 这严重限制了拉曼信号在光合作用中的使用[111]. 一些报道提出了可提高拉曼信号对比度的方法, 例如使用长波长的激发光源来尽量避免Chl的激发[112], 或让自发荧光快速漂白后再探测拉曼信号[113], 以及通过共振效应增强拉曼信号等[110,114]. 目前, 室温拉曼光谱显微镜已被应用于光合细胞测试. 低温拉曼光谱显微镜的研发最近也似乎取得一些进展[115]. 单分子拉曼光谱虽然已通过针尖增强效应来实现[116], 但色素蛋白复合体的拉曼探测也还存在着巨大的挑战(表1).
需要注意的是, 在进行显微光谱探测时, 光谱的精确测量受到色素重吸收, 激发光散射和反射效应等因素的影响, 影响程度取决于探测的样品中的空间位置[48,117,118]. 吸收光谱和激发光谱对激发光的光谱散射非常敏感, 严重的散射可导致它们发生很大的光谱畸变. 而发射光谱和荧光寿命则主要受Chl的重吸收效应的影响, 尤其是对于短波长区域. 拉曼光谱虽然对激发光散射和反射效果很敏感, 但可以通过改变激发波长来转移探测的拉曼散射至近红外波段, 在那里这些影响因素将变得微弱. 以上提到的3个因素在单细胞的显微镜光谱实验中尤为明显.
-
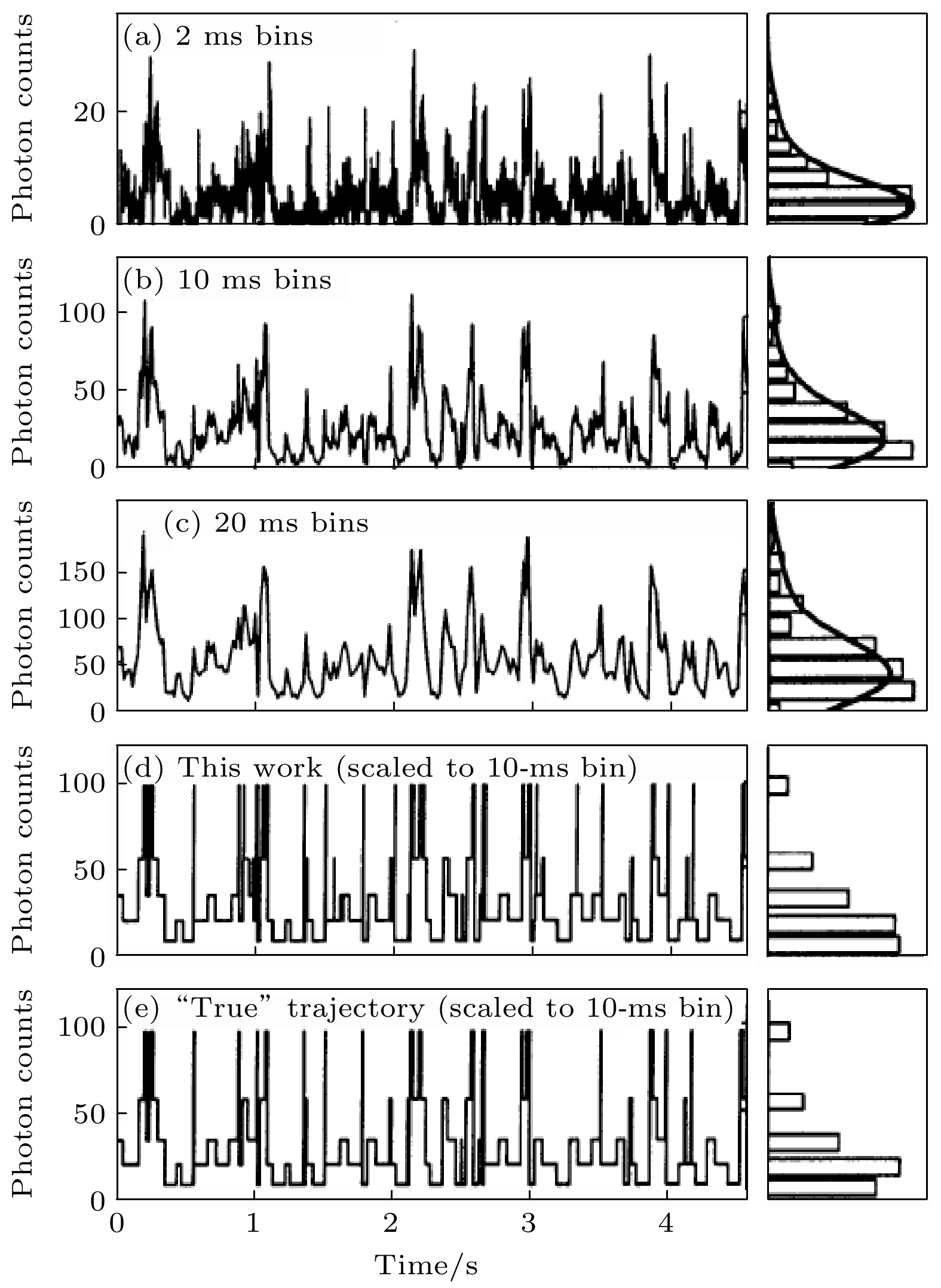
单分子的两个常用物理参量: 荧光强度和寿命, 能够被基于单光子计数 (time-correlated single photon counting, TCSPC) 的方法直接测量(见4.3.4节). 在计算荧光强度和寿命时, 通常需要对所有单光子进行数值统计, 从而得到光子的直方图分布(称为荧光衰减曲线), 光子数量可作为荧光强度参数. 然后通过直接对荧光衰减曲线进行拟合而得到荧光寿命参数. 然而, 由于单分子的信噪比较低, 因此使用全体光子统计法难以精确地分离背景噪声与单分子信号. 另一种分析方法是分段式地光子统计. 在该方法中, 设置一定的时间间隔(或单位时间), 然后对各个时间间隔内的所有光子进行数值统计, 从而得到多个荧光衰减曲线. 因此, 从多个荧光衰减曲线中能够获得多个荧光强度和寿命. 广义似然比检验的变换点分析法(change points analysis)可快速计算一段时序数据中的变换点(图5), 它能够高效地分析和精炼物理参量[119]. 变换点分析法可用于分析时序观测中从多个荧光衰减曲线中得到的强度和寿命的变化. 这些变换点一旦被确定, 一段时序数据上的分子状态将被确定. 结合了变换点分析的单光子计数测试已被证明能够精准地反映单分子的真实特征.
目前, 许多捕光蛋白的光物理性质已被该方法直接分析, 包括LH2 (light harvesting complex 2)[120], LHCII (light-harvesting complex II)[107,121]和APC (allophycocyanin)[122]. 另外, 该方法常用于表征的捕光蛋白的结构异质性, 这对于理解能量调控机理十分关键, 例如: LHCSR1 (light-harvesting complex stress-related 1). LHCSR1是一种需通过紫外线诱导的色素蛋白分子, 它在非光化学淬灭(nonphotochemical quenching, NPQ)机制中扮演重要的角色[123,124]. NPQ是一种避免光合生物受到过度激发的光保护机制, 它将多余能量以热的形式耗散掉[125]. 目前, 虽然对于NPQ的分子机制尚未达成共识, 但能够确定地是该机制可被两个因子所触发: pH值[126]和Car的脱环氧作用[127,128]. 2017年, Kondo等[107]使用单分子光谱首次测量了在不同pH值和Car的脱环氧作用下苔藓植物的LHCSR1. 他们的变换点分析显示, 荧光强度与寿命在时序观测中出现了至少两种明确的状态: 高强度/长寿命和低强度/短寿命, 即非淬灭状态与淬灭状态. 这些数据发现了LHCSR1蛋白中与pH值和Car的分子结构变化高度相关的能量淬灭, 并且证明了LHCSR1自身有能力通过结构的变化来调控淬灭程度, 他们的结论为体内NPQ机制的建立提供了一个分子模型. 原则上, 一段时序观测上的任意参量的变化都能通过该方法进行分析.
-
两种最常用的聚类分析法是K均值(K-means)聚类与高斯混合模型(Gaussian mixture model, GMM)聚类. 前者是通过预测质心, 然后通过欧氏距离计算质心与每个数据间的值, 再将数据分类进入唯一一个组的硬聚类方法; 而后者是一种软聚类方法, 在GMM聚类法中, 所有数据被假设来自于由多变量高斯成分构成的联合概率密度函数. 当二维高斯函数被假设时, 联合概率密度函数为
式中,
$ \xi $ 是具有两个分量的向量; w表示每个高斯组分所占的比例, 满足$ {\displaystyle\sum }_{i=1}^{k}{w}_{i}=1 $ ; µ代表均值;$ \boldsymbol{\Sigma } $ 代表协方差矩阵. 通过GMM拟合所测数据, 联合概率密度函数可估算出每个数据被分入各高斯组分的概率. 无论是对于哪一种聚类法, 都可以使用贝叶斯或Akaike信息准则来选择一个最佳的分组数量.最近, GMM聚类法被Squires等[30]用来分析单个光合蛋白的物理参量. 他们使用单光子光谱法同时获取了从螺旋藻Spirulina platensis中提炼的藻蓝蛋白组装体C-PC (C-phycocyanin)的多个物理量, 包括发射光谱、荧光强度、寿命、荧光偏振强度, 每种物理参量通过GMM聚类法细分为多个组分. C-PC是一种三色素类型的复合体, 各物理参量的分类能够简化对C-PC中单个色素的光物理动态的直接表征. 取决于数据的维度, 聚类分析法不仅可被应用于分类物理参量, 还可分类光谱和二维光谱图像.
-
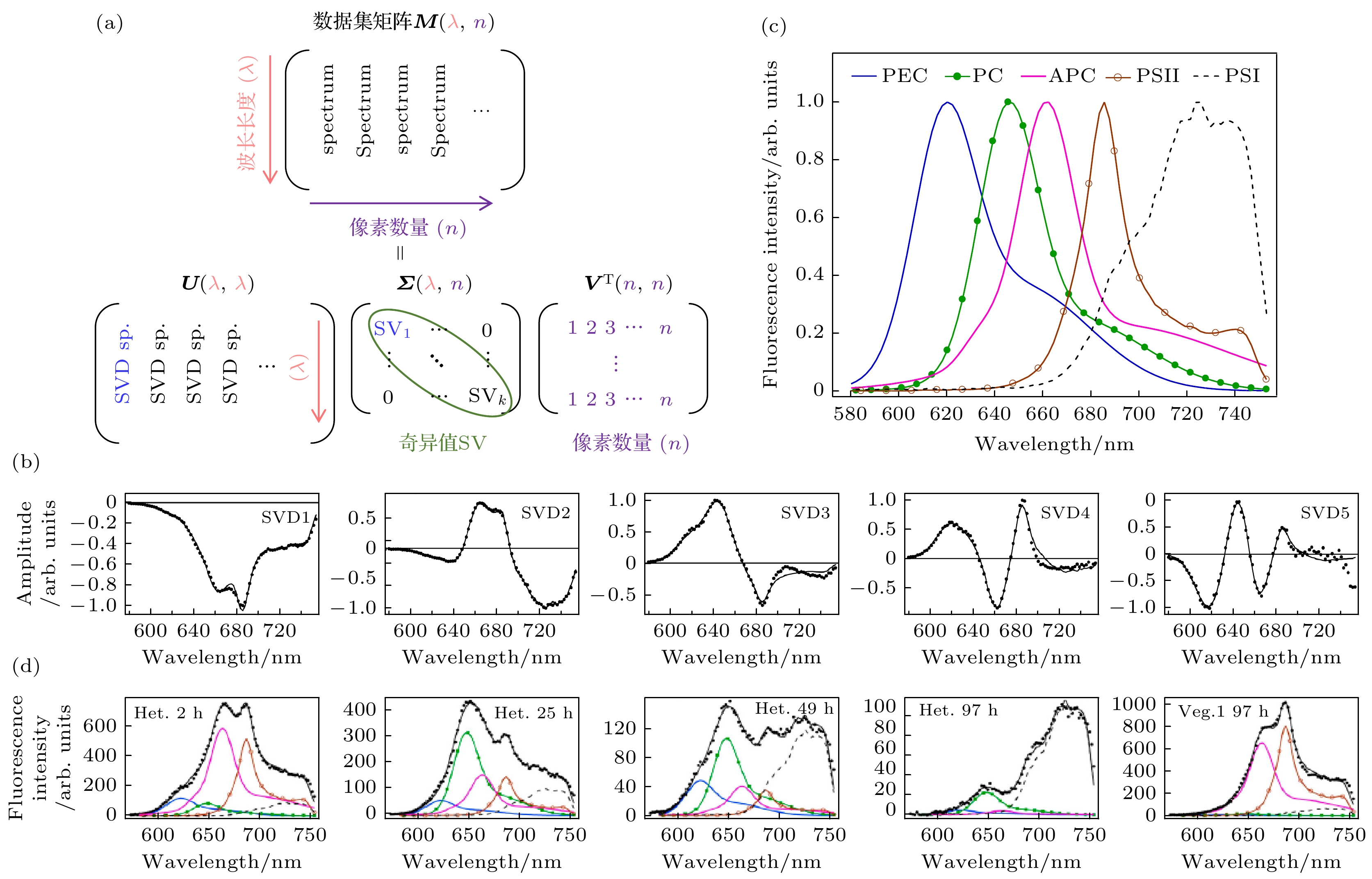
在单细胞光谱探测实验中, 光合细胞内各像素上所含的光谱是多种光合物质的光谱的和. 然而, 虽然纯化过的光合物质的光谱能在体外被独立地测量, 但各成分对单细胞光谱的真实贡献是无法被准确估计的. 因此, 提取各物质的成分光谱对定量该光合物质在微观区域的变化非常重要. 奇异值分解(singular value decomposition, SVD)法是一种分解矩阵的方法, 该方法有助于提取可用于描述当前所有光谱数据的成分光谱. 如图6(a)所示, 所测量到的大量光谱可被收纳在一个矩阵数据集M中, 通过SVD法可将该数据集分解为3个矩阵U,
${\boldsymbol{ \varSigma}} $ 和VT的乘积, U和$ {\boldsymbol{\varSigma}} $ 分别收纳了SVD谱和奇异值(singular value, SV). SVD谱是一个与所测光谱等长的数值谱, 有正负振幅但没有物理意义. 在M矩阵中的任何光谱都可通过SVD谱的线性组合来近似. 为了再现这些SVD谱, 非负的物理性光谱是需要提前准备的. 最后, 通过最小化SVD谱与物理性光谱间的差异来修正物理性光谱(成分光谱).2013年, Kumazaki等[129]在显微镜光谱测试中使用该方法分析了链状蓝细菌 (anabaena variabilis)多细胞生物中大量的荧光发射光谱. 该蓝细菌生物含有两种特征细胞: 营养细胞(vegetative cell)和异型细胞(heterocyst cell), 后者在低氮环境下容易形成. 他们分析了来自两种细胞的1625个发射光谱, 并获得具有最大SVs的5个SVD谱(图6(b)). 他们使用从细胞中分离出来的光系统以其捕光蛋白的发射光谱作为最初的成分光谱, 然后通过最小化初始成分光谱与SVD谱间的差值来精制用以定量所有光谱的成分光谱(图6(c)). 成分光谱的相对强度变化可用于追踪特征细胞在缺氮处理后分化过程中的生理状态(图6(d)). 另外, 在光合成分的发射峰波长已知的条件下, 初始成分光谱可直接通过高斯函数来获取[61]. 最近, 该方法也应用于发育过程中光系统的早期组装体的表征(见5.3节)和多细胞光合生物中光合物质的空间性定位分析等[130].
-
低温下的分子吸收谱线会变得尖锐, 这使得通过窄带激光来选择性地激发单个分子成为可能. 最初, 冷冻显微光谱技术的发展是起源于对浓稠相中单分子光谱探测的需求[131,132]. 在光合作用研究中, 一个早期的单分子光谱探测到了单个捕光蛋白的荧光强度和寿命, 该研究开启了借助单分子荧光参数来表征其结构变化的先例[23]. 后续大量的单分子实验在该领域内被展开, 这些实验证明, 光合蛋白的结构异质性与能量异质性和功能异质性直接相关[17,29].
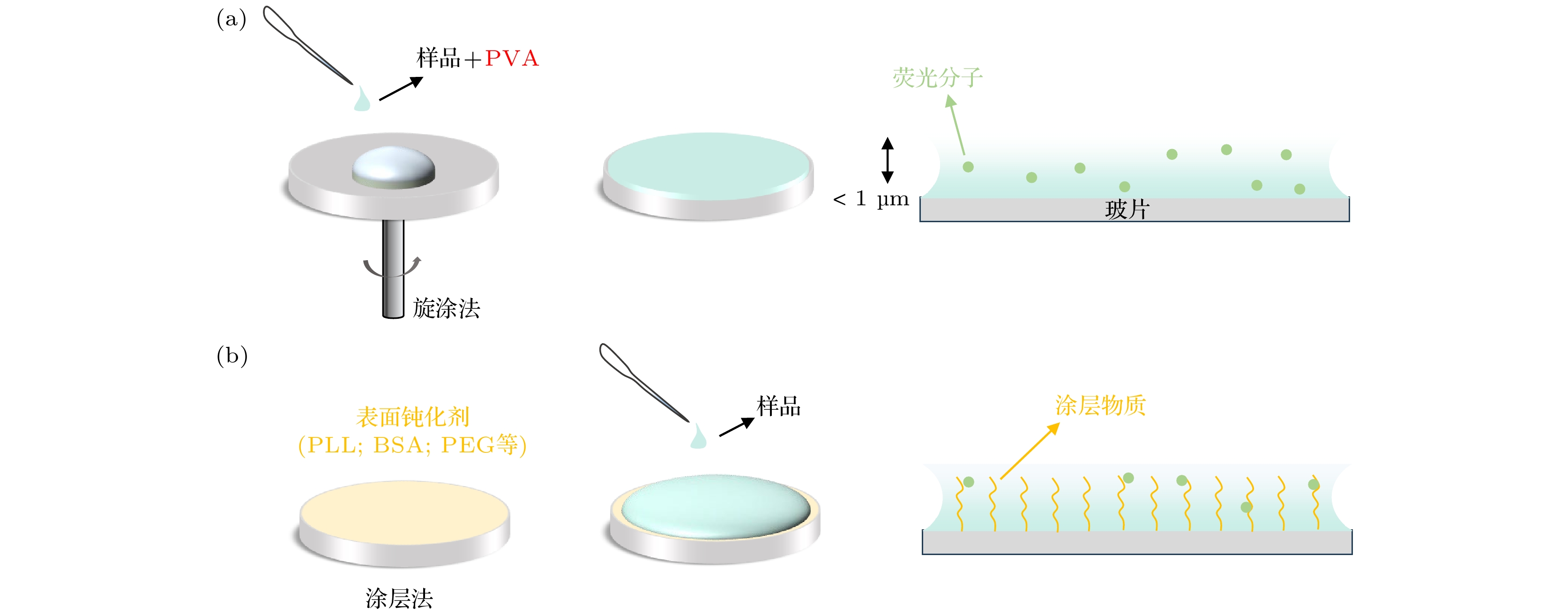
在单分子光谱实验中, 样品的分子浓度通常需被稀释至低于10 pmol/L. 当低浓度的分子样品被打散在基板(一般为石英载玻片)上后, 通过使用共聚焦显微镜系统能实现对接近光学衍射极限大小的亮点进行空间性的选择. 目前, 主流的单分子制样方法有两种. 第1种是通过将低浓度样品与聚合物相混合, 然后通过快速旋涂的方法来制备(图7(a)). PVA (Poly (vinyl alcohol))因其黏度高, 易成膜等特点常被用作于单分子的旋涂制样[16,17,71,72,98]. 在旋涂后, 样品会形成一层厚度约几百纳米的膜[133], 这与光谱显微镜的光轴分辨率接近, 因此, 单分子光谱的探测会变得高效. 该方法因其操作简单、制样快等优点, 是目前最常用的制备方法. 然而, PVA所引入的聚合物-蛋白质相互作用对单分子的结构有一定的影响, 在讨论光谱特征时该因素应该被考虑(参见4.2节). 第2种制备方法是涂层法(图7(b)). 在该方案中, 使用一些生物兼容分子预先涂在基板上(钝化处理), 例如PLL (poly-L-lysine ), BSA (bovine serum albumin), PEG (polyethylene glycol), 该步骤是为了阻止目标分子与基板的非特异性结合. 然后将稀释的样品滴在基板上, 自由运动的分子可被静电相互作用或其他分子作用力牢牢“钩”住[24,134,135]. 其他一些方案也相继被提出, 例如使用蛋白质His-tag标签与Ni-NTA涂层的相互作用来固定单分子[9,107], 或通过外加电场实现对溶液相中单分子的布朗运动进行抑制[136] 等.
为了确认所探测到的信号是否来自于单个分子, 需通过发射荧光强度的二次相关函数
$ {G}^{\left(2\right)}\left(\tau \right) $ ((2)式)来进一步验证[137,138]. 在这个实验中, 荧光图像上接近光学分辨率的荧光点被测量, 在荧光发射的检测端放置一个50/50的分波器, 通过分波器后的光子被两个独立的检测器所测量. 两个检测器以可变的延迟时间$ \tau $ 记录到达的荧光光子:$ {G}^{\left(2\right)}\left(\tau \right) $ 函数表示为两个探测器处光子间时间差$ \tau $ 的直方图, 它可以描述同一时间在两个独立的检测器处观察到同一个光子的概率. 如果荧光光子是来自于单个发射体(单分子),$ {G}^{\left(2\right)}\left(0\right) $ 处的强度应接近于零. 对于一个严格的单分子光谱探测来说, 强度二次相关实验是必须被预先执行的. -
2011年, Hussels和Brecht[72]讨论了在PVA存在或者不存在的情况下, 单个PSI发射光谱的异质性问题. 原则上, 所有单分子光谱的平均光谱应与溶液的稳态光谱一致, 但他们发现, 相对于非PVA条件, PVA条件下的单个PSI的发射光谱的峰位分布更广, 这表示通过PVA旋涂法制备的单分子表现出更多非完整结构的异质性. 聚合物–蛋白质相互作用可能通过改变水–蛋白质界面或直接与蛋白质接触来改变蛋白质构象[139]. 他们还发现PVA膜中单分子的红发射峰受到较强的限制, 导致了平均光谱蓝移了约7 nm[72]. 同样的蓝移现象, 被Zhang等[34]再次观察到. 他们还对比了PVA对PSI单体和三聚体的影响, 结果表明在PVA条件下三聚体的发射光谱发生明显蓝移但单体受此影响不显著. 因为PSI的发射光谱仅来源于几个由Chl多聚体组成的Red Chl, 因此, 当PVA存在时, 聚合物–蛋白质相互作用可能通过以下两种方式影响PSI的发射峰: 改变色素多聚体中某个分子的点位能量, 或改变色素分子间的耦合强度. 这两个因素都被考虑为与蛋白质的结构动态相关.
Zhang等[34]首次讨论了PVA对单分子激发光谱的影响. 首先, 他们观察到单个PSI的激发光谱峰存在较大的异质性(10—15 nm). 然后, 他们发现在PVA条件下PSI三聚体的平均激发光谱比稳态光谱更窄, 这是由于单个PSI的激发光谱的峰位异质性受限所导致的. 与发射光谱不同的是, 激发光谱能被PSI中所有Chls所贡献, 所有分子的点位能量是完全随机变化且独立的, 因此, 色素的点位能量变化不被考虑为激发光谱的峰位异质性的主要分子起源. 他们考虑激发光谱的峰位异质性应考虑为单个色素蛋白的分子取向和能量传递路径的异质性. 虽然目前不能定量哪一个因素的贡献更大, 但可以推测是PVA通过某种方式影响了以上异质性, 从而改变了单分子的激发光谱. 此外, 他们建议三聚体的激发光谱可能对分子取向更为敏感. 分子取向的异质性可能通过对单分子进行时程观察来规避.
以上报道可以确定, PVA薄膜中色素蛋白复合体不具备与天然复合体一样的完整结构. 因此, 通过测量PVA薄膜中的单分子光谱而推导的结论应当考虑蛋白质本身存在结构失序的问题. 目前, 单分子光谱实验中聚合物-蛋白质相互作用的影响在国际上尚未受到重视, 这可能是由于缺少一个全面且综合的对比实验.
-
荧光光谱可分为3类: 荧光寿命、发射光谱和激发光谱. 在光学显微镜系统中, 用于探测荧光的检测器一般分为单元素检测器和二维阵列检测器两大类, 例如APD (avalanche photodiodes)系列和CCD (charge-coupled device)系列等. 前者可通过与TCSPC系统相连接直接检测荧光寿命, 后者可采用与光谱仪组合的配置快速地检测发射光谱. 使用搭配了点激光的共聚焦显微镜执行光栅扫描可获取单个像素中的光谱.
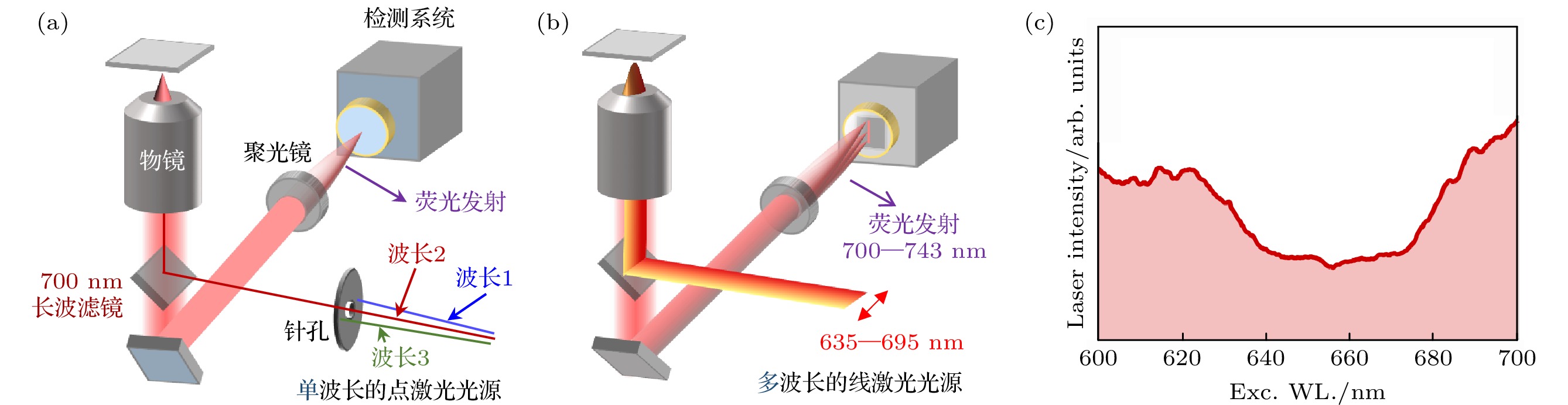
与荧光寿命和发射光谱不同的是, 激发光谱的获取一般需要进行激发波长扫描. 通常的做法是, 先使用单波长激发获得荧光图像(图像预扫描), 然后选择性地对图像上的亮点进行激发波长扫描以获得荧光强度与激发波长的函数(图8(a)). 该做法的弊端是因为波长扫描非常耗时, 所以不可避免地将带来显微镜失焦的问题, 这是导致激发光谱获取效率低的主要原因. 最近, 搭配了线激光的光谱显微镜系统变得逐渐流行[68], 该系统可将光谱获取效率提高几十至100倍. 2020年, Jana和Shibata[105]开发了基于多色线激光的激发光谱光谱显微镜. 在他们的系统中, 通过使用三棱镜将一束白光分解为包含了多个波长的多色激光, 该激光在空间上呈线状分布并具有一定长度和宽度. 当在对激发光谱进行测量时, 样品中的各个像素会被线激光光源的长轴(长度方向)和短轴(宽度方向)依次扫描, 从而快速获得多个激发波长下的荧光强度. 线激光的短轴能够过滤失焦信号, 提供了共聚焦的功能. 因此, 如此配置也被称为半共聚焦光学系统(图8(b)). 他们的显微镜系统可记录实时变化的激光光谱, 用于校正激发光谱(图8(c)). 该高速激发光谱显微镜无需执行图像预扫描, 成功地实现了激发光谱的高通量获取(图8(b)), 已被使用于专门研究色素蛋白体系中与天线系统相关的问题[34,49,140].
-
在极低的温度下(< 4 K), 在单个色素的发射光谱中能观察到明显的零声子线 (zore-phonon line, ZPL)和声子带(phonon wing, PW) 结构. ZPL出现在波长上的高能量端且表现为极窄的谱线, 其反映了纯粹的电子跃迁过程. 而PW位于低能量端, 呈现为一个很宽的光谱形状, 它反映的是电子基态的声子激发模型, 又或者是蛋白质的低频振动模型(色素的周围环境). 如(3)式所示, ZPL和PW之间的强度分布可用黄-里斯因子S表示, 该因子表示电子跃迁过程中声子发射的平均数量, 能粗略地估计色素蛋白质复合体中的电子-声子耦合强度:
另一个重要的现象是光谱扩散, 称为spectral diffusion或spectral jump[141]. 光谱扩散被定义为色素因吸收频率发生变化其光物理性质或分子环境被改变, 从而导致光谱峰位波长发生位移(约几纳米). 当体系温度升高时, 更快的蛋白质运动会迅速调制色素分子的点位能量, 快速的光谱扩散让色素蛋白质的ZPL变宽, 因此ZPL出现的概率变低[12]. 并且, 色素分子周围的环境发生变化(如氨基酸残基结构等)时, 也会影响ZPL出现的概率.
大量的低温单分子光谱实验聚焦于LH2和PSI的ZPL探测. ZPL的变化能反映蛋白质在能量地形中的构象子态或某个子态的振动自由度. 在LH2中, BChla分子的特殊排布导致其可发生高度离域激发, 因此可以通过观测激发光谱上的ZPL来研究其激子态结构(参见4.4.1节). 在PSI中, 发射光谱的ZPL也被用来研究Red Chls的发射状态. 通过对发射光谱的偏振检测能确认各个发射状态间的角度, 不仅能确认它们之间发射是否发生发生直接能量转移还可通过蛋白质的原子结构来预测Red Chls的归属(参见4.4.2节).
-
荧光闪烁(fluorescence blinking)是单分子的一个非常重要且有趣的现象, 它被定义为荧光随时间而发生间歇性的点亮或熄灭(或荧光强度的增强和减弱)[10]. 2014年, Shibata等[98]首次捕捉到了PSI的荧光闪烁现象. 虽然目前并没有完全阐明PSI荧光闪烁的潜在机理, 但似乎能够通过天线系统中传能路径变化来解释[32,34,142]. 这里假设流向Red Chls或反应中心的传能分支比的波动会导致荧光发生间歇性变化: 在荧光点亮期间, 激发能量优先流向Red Chls; 在荧光熄灭期间, 激发能量传递偏向于流入反应中心, 此处的荧光通过一个淬灭体P700+ (反应中心的氧化态)被消耗[32]. 在PSI的单分子光谱实验中, 由于其荧光量子收率很低, 高强度激光的使用让反应中心P700几乎不可避免地被光氧化为P700+, 即使加入还原剂, 该成分也仍然存在[32]. 以上所建议到的荧光闪烁机理成立的一个前提条件是天线系统内的激发能量传导路径发生波动. 2009年, Brecht等[142]在1.4 K下观察到了单个PSI中不同红发射波长间的反相关变化. 最近, Zhang等[34]使用一种新的冷冻单分子光谱技术在80 K下观测到了不同Red Chls中激发光谱的差异, 且激发光谱随时间而发生显著变化(参见4.4.2节). 以上两个结果是天线系统内传能路径存在波动的有力证据.
-
单分子的荧光强度和寿命可以通过使用单光子计数法来同时探测. 通常, 强度和寿命分别使用每单位时间内的发射光子数量和通过拟合荧光衰减曲线
$ F\left(t\right) $ (光子集合)而得到的时间参数$ \tau $ 来表示:式中, IRF (instrument response function)代表仪器响应函数, 参数
$ \tau $ 可通过单组分指数函数与IRF的卷积计算来求解. 在单分子的时程观测中, 这两个参数能被变换点分析快速处理(参见3.4.1节). 该测试常被用于分析捕光蛋白的功能性状态. 需要注意地是, 荧光强度取决于发射分子的浓度和寿命. 在使用强度表征状态易发生变化的光合物质时, 必须考虑以上两个因素对强度的影响, 尤其是对于非单分子探测[55,56].另一方面, 每个单光子的延迟时间都包含了荧光寿命的信息, 但时间参数
$ \tau $ 并不能将每个光子的信息完全利用. 这里介绍一种升级的分析手法, 即二维荧光寿命相关光谱(2-dimensional fluorescence-lifetime correlation spectroscopy, 2D-FLCS). 该方法是一种基于分析逐个光子而开发的, 主要具有以下4个优势: 第一, 不需要进行任何硬件升级, 仅需对数据处理进行改良; 第二, 可利用荧光寿命信息来分析蛋白质的结构动力学; 第三, 因为2D-FLCS的时间分辨率仅取决于光子的检测频率, 而不是光子集合的曝光时间(几十毫秒), 因此可提高时间分辨率至微秒尺度; 第四, 可表征同时发生在多个时间尺度上的多个动态过程. 2D-FLCS最初是由Ishii和Tahara[143,144]基于荧光相关光谱的思路在2013年所开发, 在他们的最新版本中, 通过双色脉冲交替激发的方式已实现非荧光共振能量转移成分的识别[145]. 2019年, Kondo等[15]首次使用该方法研究光合蛋白, 他们定量地分析了在NPQ触发条件(低pH值)下LHCs的构象子态的动态平衡. 他们在单个LHCs中观测到两个明确的状态: 捕光态(高强度/长寿命)和淬灭态(低强度/短寿命), 它们对应于明确的构象子态. 单分子2D-FLCS揭示了LHCs的两个状态涉及多个时间成分, 它们具有不同的转化速率. 通过合理使用模型函数的相关性分析, 可以计算处于两个状态的各成分的自由能. 因此, 构象子态间的跨越可通过各态间的自由能差异被定量描述. 当感知pH变化的残基被敲除时, 在LHCs中几乎观测不到各态间依赖于pH值的自由能差异. 这证明在pH敏感的残基部位的变化可调控LHCs的局部构象灵活度, 对LHCs光物理动态的表征为分析体内NPQ机制提供了分子模型. -
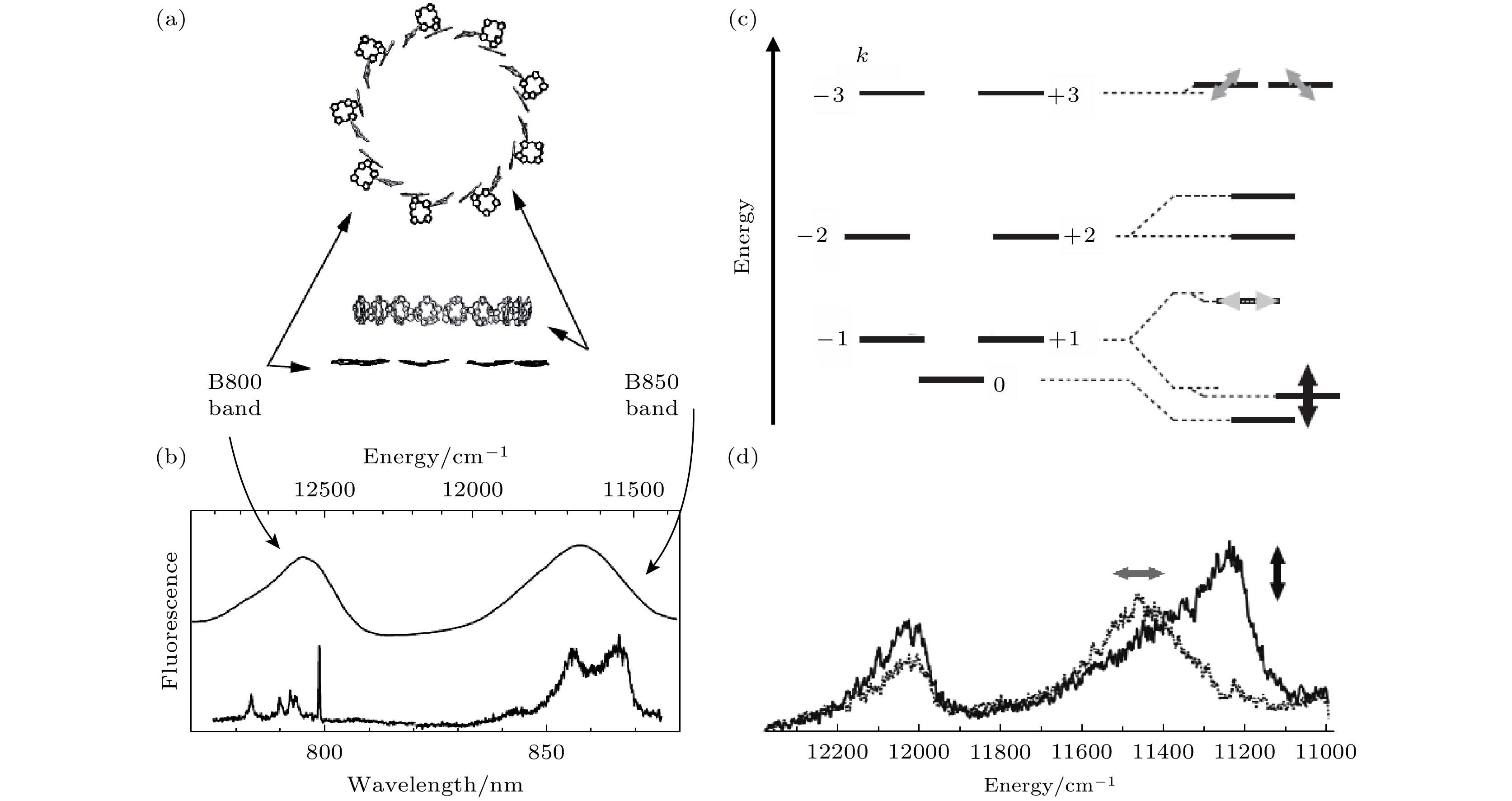
在紫细菌的光捕获蛋白LH2中, 色素排布的独特性吸引了许多学者对其电子跃迁和能量调控的兴趣[13,24,27,28,104,146,147]. 早期的许多冷冻单分子光谱实验围绕着研究LH2的电子结构来开展[27]. 从Rhodopseudomonas acidophila和Rhodobacter sphaeroides菌株中提取的LH2是最常用的模板. LH2复合体结合了27个BChla, 这些色素可细分为呈圆环型排布的两个组: 它们分别由分子间距约2 nm的9个BChla和分子间距小于2 nm的18个BChla构成(图9(a)). 溶液相中的两组色素在800 nm和 850 nm波长附近有最大吸收峰, 因此它们分别被命名为B800和B850 (图9(b)).
在1.5 K温度下, 在LH2的单分子激发光谱中可在800 nm附近观测到4—5个极窄的谱线; 而在850 nm区域, 能观测到经典的双峰分布(图9(b)). 该观测表示B800的光激发可通过局部的单个色素激发模型来描述, 而B850的激发模式应考虑Frenkel激子模型[148]. 理论上, B800的激发光谱应出现9个窄线, 它们严格地对应于9个BChla色素分子. 然而, 由于各个色素的快速光谱扩散导致在观测时间内相邻的两个分子被共同激发(局部离域), 因此无法被完全捕获B800中各个BChla色素的电子状态. 最近, Otomo等[33]完成了B800的中红外响应光谱的探测. 他们的结果表示, B800峰在低温激发光谱上的光谱扩散速率与中红外光的照射强度呈线性相关. 因为C=O的伸缩振动带的中红外吸收光谱对蛋白质二级结构十分敏感, 因此该实验可提供一种通过电子结构特征来研究单个复合体的振动的技术路线.
B850中色素间的耦合强度是B800的5倍左右[104]. 在B850中, 无论先激发哪一个色素, 分子间的强耦合都会让光激发以激子的形式扩散在整个B850环上. B850的激发可由18个激子态来描述, 其中一个名为k = ±1的次低能级激子态是光子吸收所允许的状态(图9(c)). k = ±1是两个能量简并且偶极子跃迁正交的激子态, 单分子偏振激发实验确认了两个状态的正交性质(图9(d)). LH2中B850的双峰间距可反映两个激子态的能级分裂, 低温单分子激发光谱的非时程观察表示, 在各个构象子态中能级分裂呈异质性分布, 并受单分子所处环境的影响[27,104]. 激发光谱的时程观察表明, 激子的能级分裂程度受到蛋白质动力学的强烈影响, 以至于处于光学跃迁禁止的激子态k = 0变得部分可观测(图9(d)). 然而, 因为k = 0与k = ±1激子态在激发光谱上相近并伴随着光谱扩散, 这使得LH2的最低激子态的归属变得模糊[27]. 为了观测最低激子态, Kunz等[147]在同一个LH2复合体上实施激发光谱与发射光谱的探测, 通过分析激发光谱的长波长峰与发射光谱的短波长峰的相关性, 成功地归属了LH2的最低激子态 (k = 0). 更多地, 他们在1.2 K的温度下在单个LH2中观察到了一个低能量发射峰, 它比通常所观察到的k = ±1和k = 0发射峰红移了约20 nm, 该发射状态被归属为自捕获激子态[149]. 得益于LH2的电子结构的显著光谱特征, 光谱的变化也可用于表征单个色素蛋白复合体中电子-声子耦合的波动[150]. 另一方面, LH2由于其高度离域激发的特征, 也常作为研究生物中量子相干态传能的模板[151]. 在单分子超快光谱探测的实验中[152], 各个LH2复合体的相干时间呈现出大的异质性, 这暗示了蛋白质结构与量子传能间可能存在相关性. 然而, 目前对于蛋白质的结构动力学以何种方式参与, 影响和调控量子传能效率等问题仍在被持续地挑战.
-
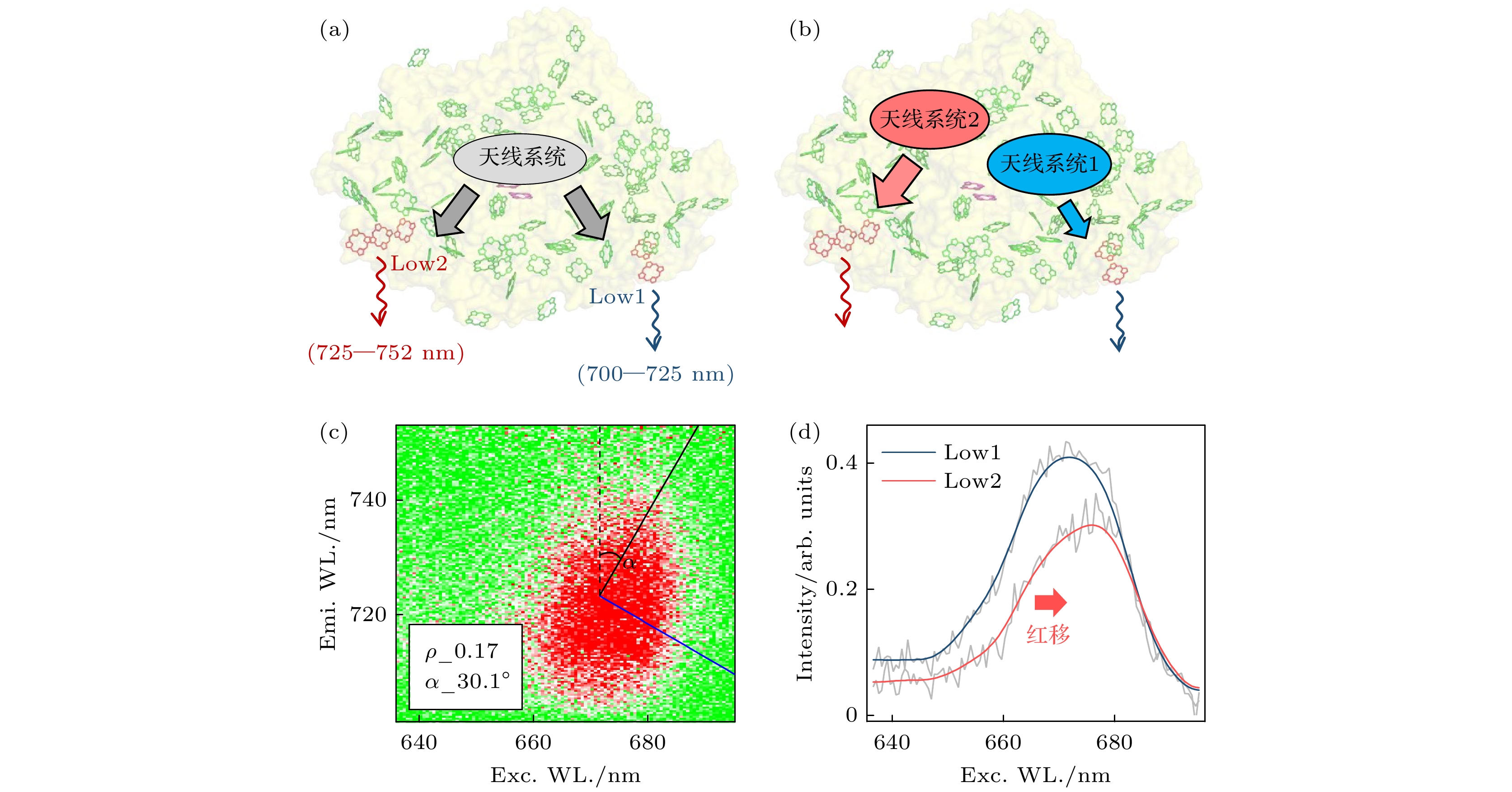
由于从T. elongatus中纯化的PSI具有较高的稳定性, 因此常作为研究Red Chl的生物模板. 最初的两个Red Chls-C708和C719是通过低温稳态吸收光谱和选择性激发的荧光光谱来确定的[153], 第3个Red Chl-C715后续被光谱烧孔实验所发现[76]. Jelezko等[11]是PSI单分子光谱实验的先驱, 2000年他们首次在4 K温度下测试了PSI的激发光谱和发射光谱. 他们的结果表示单个PSI的荧光强度依赖于温度, 这是uphill能量转移的特征. 其次, C719所发射的荧光在730 nm处有最大发射峰, 但很难观测到ZPL, 这可能是由于一个快速的光谱扩散所导致的. 后来, Brecht等[12]发现, 当样品溶剂被换成重水时, PSI的光谱扩散明显变慢并且观测到了更多的ZPL, 这强烈地证明光谱扩散受到质子运动的影响. 低温下, 在单个PSI中所观测到的光谱扩散表明色素分子的点位能量固有地受到当地蛋白质残基的构象运动影响. 他们也测量到了PSI中C715/C719较大的S因子(S ≥ 5), 这反映了Red Chls与环境基质的强耦合[71]. 在强耦合的情况下, Red Chls的电子基态与激发态所对应的原子坐标发生较大的位移. 一个较大的S因子能够通过分子激发态的电荷转移 (charge-transfer, CT) 混合来解释[11,154]. CT混合可产生一个较大的永久电偶极矩, 这导致光激发后色素分子的周围环境发生大的变形. 在710 nm, 717—725 nm和>725 nm三个波长区间内最容易检测到3个Red Chls的ZPL. 基于荧光偏振检测的低温单分子光谱实验表明, PSI的3个Red Chls起源于不同的分子团. Brecht等[26]还发现C715经历了最快的光谱扩散, 其次是C719, 而C708的光谱扩散最慢. 这表明了所属于C708的Red Chls处在一个最不易受到环境影响的位置, 而C715则相反.
发射池Red Chls间的能量转移也常被研究, 有研究报道C715和C719之间发生一个快速的直接能量转移 (≤ 5 ps)[77]. 但是, Skandary等[78]通过单分子偏振实验表明C708和C719的跃迁偶极矩间的角度接近90°, 这至少证明C708与C719之间不能发生直接的能量转移. C715与C719的发射光谱在频率上相近, 在快速光谱扩散存在的形况下难以被单独区分, 所以目前缺乏C715→C719能量转移的有力证据. 因此, 虽然由于3个Red Chls的发射光谱有明显的重叠, 但它们之间的能量转移难以用纯粹的downhill的形式来描述. Schlodder等[155]也报道了C719的荧光可能被反应中心淬灭, 并且该淬灭过程受到P700的氧化还原态的影响. 此外, 通过低温单分子光谱, 一个发射峰为745 nm的Red Chl被报道, 但缺少关于这个新发射池的后续研究[25].
-
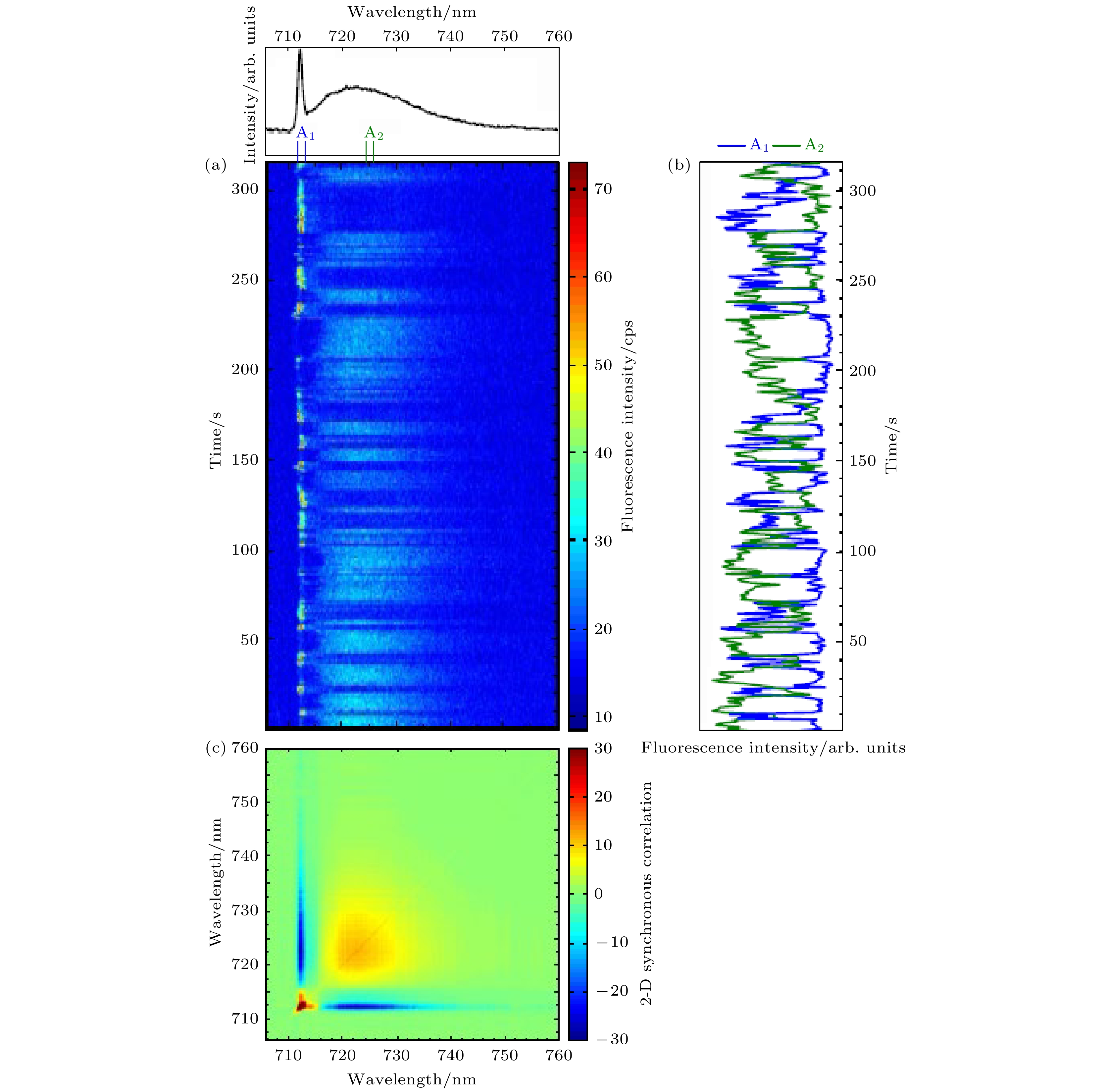
在光合蛋白中, 捕光天线系统内的能量传递路径问题是备受关注的重点之一. 由于PSI含有多个不同的发射池, 因此也常作为研究传能路径的模板. 2009年, Brecht等[142]使用单分子二维自相关光谱观察了1.4 K温度下PSI中C708和C719荧光发射的相关性. 二维自相关光谱是一种在两个波长维度
$ {\lambda }_{1}={\lambda }_{2} $ 处完全对称的光谱(图10), 可由时程观察得到的二维数据集$ y({\lambda }_{j}, {t}_{i}) $ 中产生. 如(5)式所示:式中, 平均光谱
$ {\overline{y}}_{\left({\lambda }_{j}\right)} $ 被定义为$ {\overline{y}}_{({\lambda }_{j})} = \dfrac{1}{n}{\displaystyle\sum }_{i=1}^{n}{y}_{({\lambda }_{j}, {t}_{i})} $ . 在二维自相关光谱中, 两个不同波长位置1和2处的荧光强度在时间分辨过程中的相关程度可由一个相关性系数r定量估计. 在该温度下, PSI的二维自相关光谱表示, C708和C719的发射光谱展示出相当大的反相关性 ($ r (1, 2)=-0.8 $ ). 强反相关性被解释为: 一个快速的蛋白质动力学导致天线系统随机地将能量传递给C708或C719. 这是激发能量传递路径被改变的有利证据.最近, Zhang等[34]使用冷冻单分子激发发射光谱法(single-molecule excitation-emission spectroscopy, SMEES)在80 K下直接观测了PSI的天线系统. 这里, 他们聚焦于Kato等[82]最近所提出的Low1 (700—725 nm)和Low2 (725—752 nm)发射池. Low1和Low2中的传能路径可由两个模型描述: 随机波动能量转移 (图11(a))和分支能量转移(图11(b)). 前者可通过Brecht等[142]所观察到的结果进行证实, 但后者的证明需要对天线分子进行有效探测. SMEES是基于一个他们开发的高速激发光谱显微镜系统来实施的[48,105], 该方法实现了低温下单分子PSI的激发光谱与发射光谱的同时获取, 让PSI中天线分子与终端发射分子(Red Chls) 的信息得以同时被描述. 使用该方法Zhang等[34]获得了PSI的二维激发发射矩阵 (two-dimensional excitation-emission matrix, 2D-EEM)图(图11(c)), 该数据可重建不同荧光发射区域的激发光谱 (图11(d)). 他们通过2D-EEM的倾斜角和相关性系数来定量单个PSI中的激发-发射相关性 (图11(c)). 他们的观测得出两个重要结论: i) 他们测量到倾斜角和相关性系数都较大的2D-EEM (图11(c)), 即一个较强的激发-发射相关性被揭示. 当正倾斜角足够大时, 能观测到Low2的激发光谱发生红移, 反之亦然(图11(d)). 激发光谱是由全体色素分子所贡献的, 因此Low1和Low2的激发光谱的差异反映了它们由不同的天线系统供能, 即证实了分支能量转移模型的存在(图11(b)). 更重要的是, 在测量了大量单分子后他们发现激发-发射相关性呈较大的异质性分布, 这揭示了分支能量转移模型强烈地依赖于蛋白质构象的特征; ii) 通过对Low1和Low2进行时程观察后发现, 它们的激发光谱发生显著变化, 这是传能路径发生波动的直接证据. 他们建议蛋白质结构波动是影响传能路径发生波动的主要原因, 这与之前的报道一致[142]. 他们的结果也表示, 2D-EEM 中的激发-发射相关性可被Low1和Low2的相对荧光强度所解释, 因此SMEES的观测结果可能提供一个基于传能路径变化的模型来解释PSI中的荧光闪烁现象.
低温单分子激发光谱也被用于研究光合细菌hRC (heliobacteria)的传能路径, hRC含有一个同型二聚体结构的反应中心, Red Chls分别分布在两个子蛋白中. 通过对电子传递辅因子的激发光谱观测, Kondo等[16]在6 K温度下发现了hRC中的两个子蛋白间可发生直接的能量转移, 且能量转移的对称性被受到蛋白质结构异质性的影响. 他们也观测到hRC中红发射状态的激发峰在长时间(几十分钟)内发生几纳米的光谱扩散. 这个现象被解释为: 发射分子的周围环境的波动调控了能量转移, 因此可反应向不同Red Chls供能的激发能量路径的切换.
-
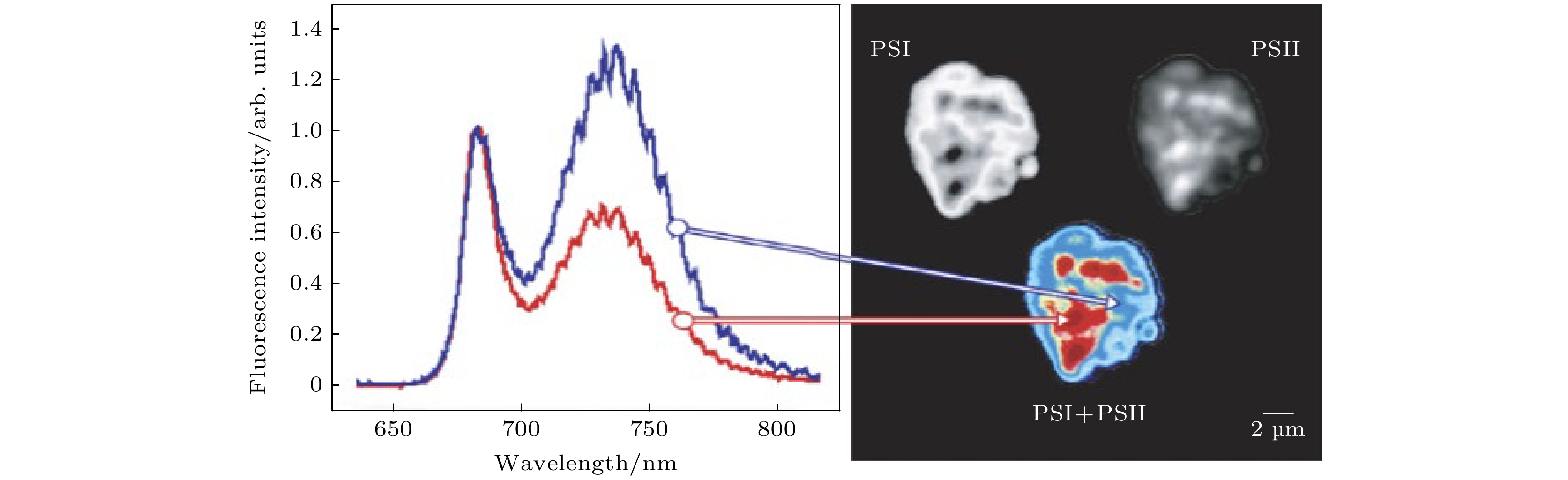
Vàcha等[90]首次成功使用基于折射率梯度物镜的冷冻显微镜在植物叶绿体内粗略地可视化了PSII和PSI的体内分布. 他们通过固体浸没效应将有效数值孔径(numerical aperture, NA)提高到0.9. 在这个实验中, 叶绿体溶液和盖玻片通过表面张力附着在透镜的输出表面上, 然后直接将其侵入低温器中的液氮里并固定. 分别在680 nm和730 nm附近通过两次连续扫描检测到PSII和PSI的发射信号. 在获得的双色图像中, 观察到几个具有较强PSII荧光信号的圆形区域, 被归属为PSII富集(PSII-rich)的主要区域-基粒堆(类囊体的层叠区). 基粒堆的估计直径为0.8 μm, 略大于电子显微镜种所观察到的大小. 这种较大的尺寸可能是由于轻微的失焦或邻近基粒堆的重叠. 基粒堆间的连接处结构-基质片层也能被表征, 这是PSI主要富集(PSI-rich)的区域. 该观察还表明, 提高冷冻显微镜中物镜的NA可获取关于每个光合成分在叶绿体体内的异质性分布的信息[90](参见5.2节). 该研究在2007年被进一步扩展, Vàcha等[91]使用了从热带植物Aglaonema simplex中分离的叶绿体, 该叶绿体的体积较大且结构更易区分. 他们升级了现有显微镜的配置, 在检测端使用连接了成像多色光谱仪的CCD相机, 可直接检测覆盖了650—800 nm波长范围的发射光谱. 他们在细胞内的局部区域获取了具有明确PSII和PSI特征的发射光谱(图12). 这些早期研究确定了使用低温显微光谱学区分细胞体内PSII和PSI所在的微观区域是有效的. 当在微观区域中分离PSII和PSI成为可能后, 围绕着PSII和PSI的光调控生理事件能够被系统研究, 例如ST机制(参见5.2节).
-
ST描述一种通过捕光蛋白LHCII的运动来快速调控PSII和PSI间相对激发水平的机制. 衣藻细胞Chlamydomonas reinhardtii是常被用作与研究ST机制的模板生物. 在经典的ST模型中, PSII通常与LHCII相连接的(称为状态1); 但当PSII被过度激发时, LHCII发生磷酸化并与PSII解离, 然后会与PSI相结合以提高PSI的激发水平(称为状态2) (图2(f)). 一些研究推测, 从状态1转变到状态2时, 并不是所有解离的LHCII都与PSI结合, 可能存在少量的自由LHCII (不与两个光系统连接). 体外的时间分辨荧光实验表明自由LHCII表现出淬灭态[108,109,156], 但体内证据是缺乏的. 自由LHCII的体内表征对于完善ST机制的模型非常关键.
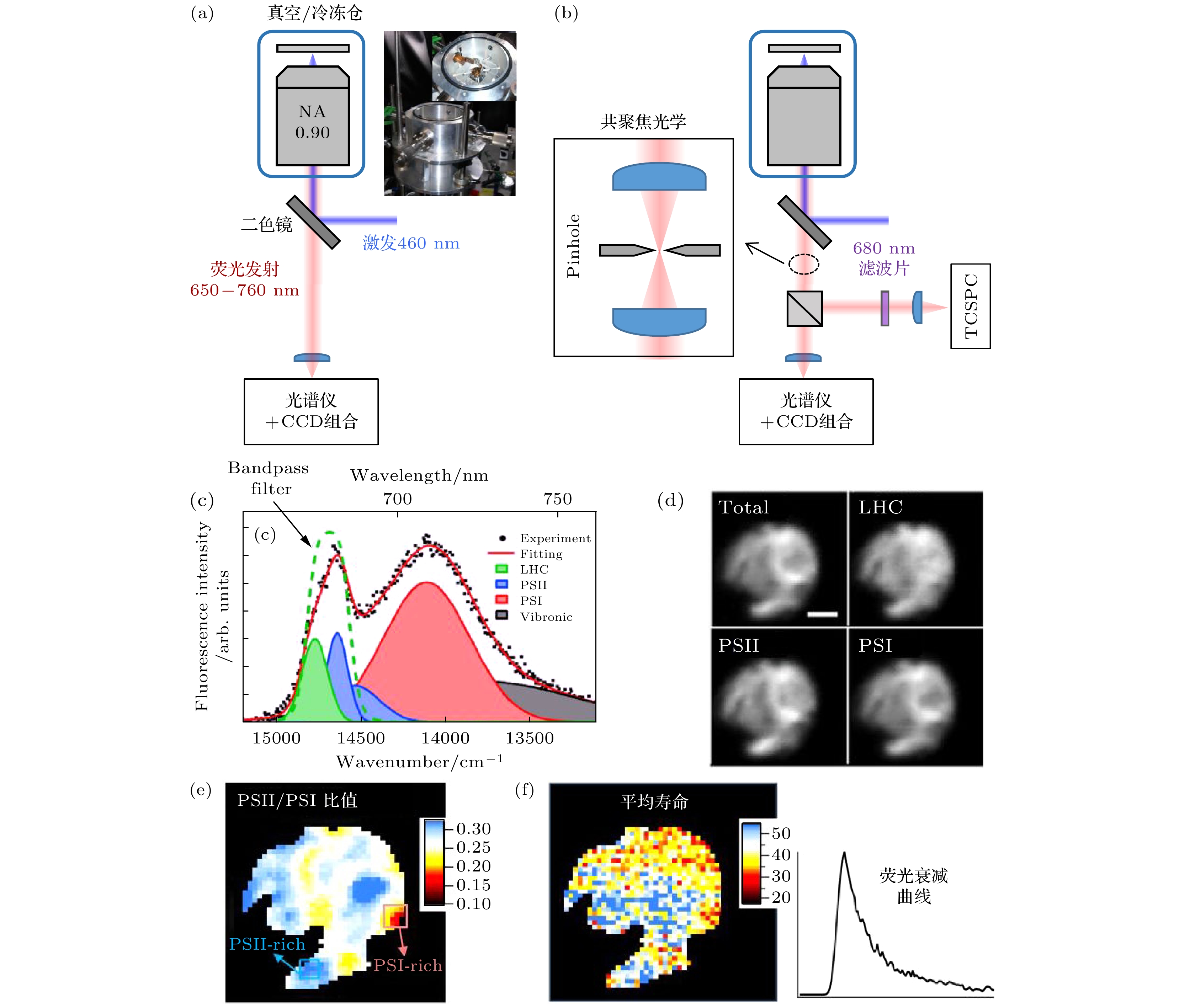
2018年, Fujita等[55]使用自主开发的冷冻光谱显微镜(图13(a))对衣藻细胞进行成像, 以研究LHCII在ST机制发生后在细胞体内的重新分布. 他们的光学系统可大范围选择观测面积的冷冻光谱显微镜, 搭配NA为0.9的真空兼容物镜, 可提供约350 nm的空间分辨率(图13(a)). 最初, 他们在80 K的温度下完成了衣藻细胞的单像素发射光谱的获取. 通过对每个像素的低温发射光谱进行高斯拟合, 将光谱分解为主要归属于LHCII (680 nm)、PSII (686 nm/692 nm)和PSI (712 nm/730 nm)的成分. 他们首先可视化了LHCII, PSII和PSI成分在细胞内的分布, 然后计算了LHCII与每个光系统在空间上的共定位程度. 虽然理论上状态1和状态2细胞中的共定位分析可表征LHCII的移动, 但实际上对LHCII的定量评估存在一些技术限制. 这是因为荧光强度不仅取决于物质的浓度而且还与其量子收率成正比, 光谱成分的幅度并不能直接反映其局部分子浓度. 因此, 要定量分析移动的LHCII, 需对其荧光动力学进行测量.
2022年, Fujita等[56]更新了他们的冷冻光谱显微镜, 即在荧光检测端加入了TCSPC系统. 升级的光学装置可同时获取同一个像素上的全波长发射光谱和荧光衰减曲线, 该系统被简称为Cryo-FLIM (cryogenic fluorescence lifetime imaging microscope) (图13(b)). 需要注意的是, 为保证Cryo-FLIM中荧光光谱检测端和衰减曲线检测端的信号是来自于同一测量位置, 两端的信号必须通过一个共聚焦光学(图13(b)). 在这个实验中, 他们修正了之前对PSI的归属, 并且探测了680 nm荧光的寿命. 因为680 nm的荧光主要反映了LHCII的贡献(图13(c)), 因此cryo-FLIM能评估细胞内LHCII的功能状态. 首先, 使用与之前同样的方法, 他们可视化了细胞内各光合成分(图13(d)). 根据光谱振幅, 他们计算了细胞内的PSII-rich和PSI-rich区域(图13(e)). 然后, 重建了对应于同一细胞的荧光寿命图, 其中每一个像素中的荧光衰减曲线都能够被独立分析(图13(f)). 在使用多成分函数全局拟合了所有细胞的发射光谱和荧光寿命后, 他们在PSI-rich区域中发现了一个极快的寿命成分(约3 ps), 该荧光寿命成分仅在状态2细胞中存在. 而且, 发射光谱的分析结果表示, 该快速衰减成分对应着一个高的荧光强度, 因此这可能是淬灭态自由LHCII富集的证据[56]. 一个体外的时间分辨荧光实验支持了这一结果[157]. 总之, 这些研究成果不仅为ST机制的完整模型的建立提供了证据, 还证明了低温显微光谱学研究可以用于可视化荧光成分在细胞内的异质性分布, 并同时表征其功能[56].
-
阐明两个光系统在活体细胞体内的组装机制是光合作用研究中被长期挑战的课题之一. 一些研究提出了光系统组装的逐步模型[58–60,158–160]. 在组装过程中, 光系统的荧光发射光谱表现出显著差异, 因此可通过荧光成分来表征和追踪早期组装体. 最初对光系统组装体的光谱表征要追溯到1957年[161], 许多后续的研究报道了光合细胞中处于不同发育状态下的荧光成分[94,162–167]. 然而, 在之前的测试中, 大多数的荧光成分会被集体样品所平均化. 在单细胞体内, 组装体中间态与荧光成分之间的联系尚不清楚.
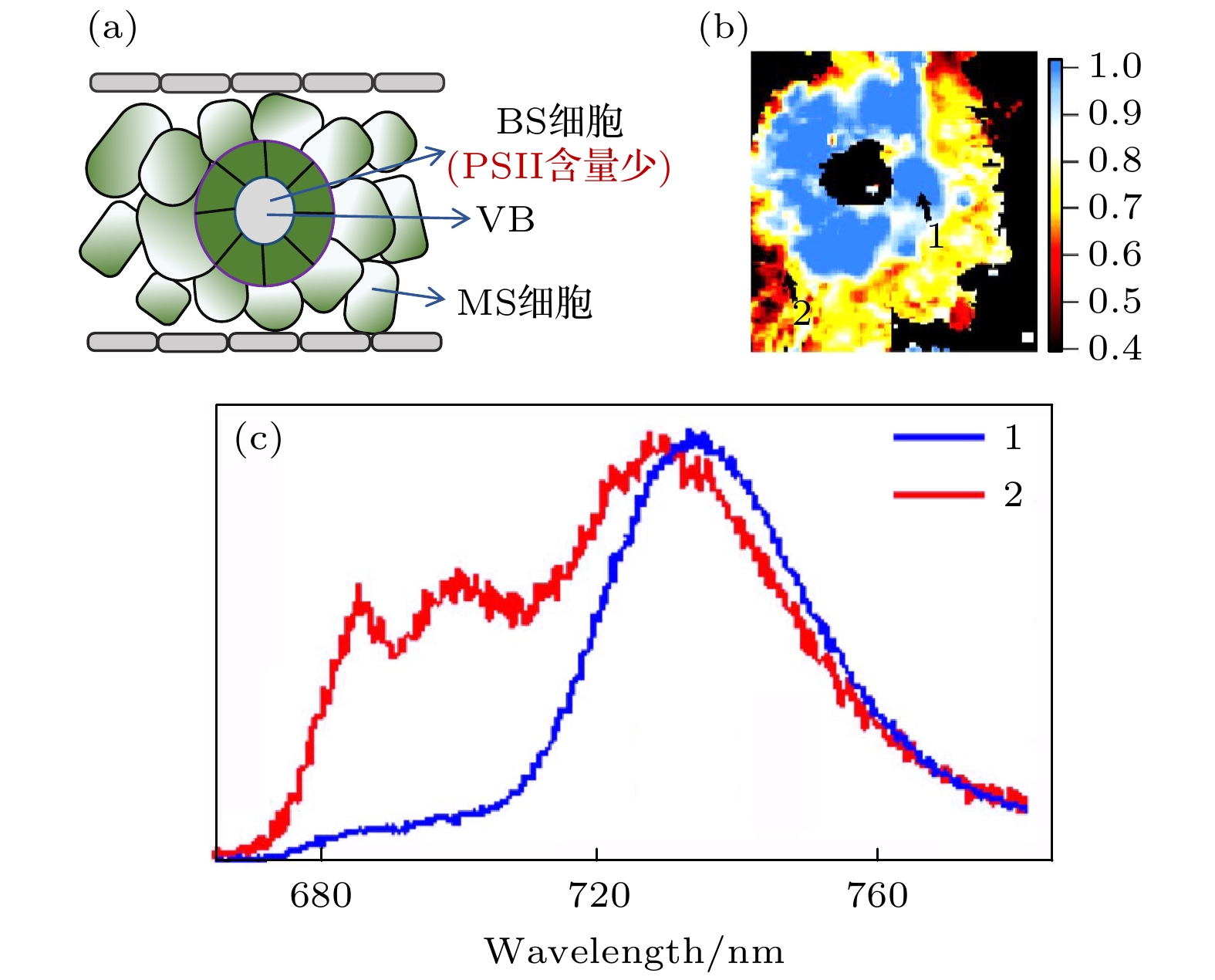
冷冻光谱显微镜的应用将该领域的研究体系推向单个细胞[61,94]. 通常, 黄化植物叶片在光照下的绿化过程让组装体变得可观测. 黄化叶片是植物幼苗在黑暗条件下发芽时形成的, 在这些叶片中, Chla色素的合成受到阻碍, 因此光系统的组装不能被启动. 光照会促发Chla合成, 从而促进了光系统的组装过程. 2013年, Shibata等[94]首次使用了冷冻光谱显微镜探究了玉米叶片的绿化过程. 玉米叶片含有两种特征类型的细胞: 束鞘细胞(bundle sheath, BS)和叶肉细胞 (mesophyll, MS). 与后者相比, 前者中PSII的含量显著减小(图14(a)). 冷冻显微镜测量的结果表示, BS细胞中荧光光谱主要来自于PSI的贡献, 而在MS细胞的发射光谱中PSII的贡献增大(图14(b), (c)). 因此, 在该生物体系中可选择性地观察BS细胞中PSI的发育过程, 也可在MS细胞中监测PSI和PSII的发育过程. 在早期的低温显微镜观察中, 由于仪器的限制, 荧光图像只能够通过3个波长通道进行检测. 他们在80 K温度下, 在绿化了1 h的BS细胞中成功捕捉到了与PSI组装体的中间态相关的679 nm荧光成分. 当BS细胞被绿化3 h后, 735 nm荧光成分变强, 他们归属该成分为PSI-LHCI或者自由的LHCI. 该研究证明了玉米叶片的荧光发射具有很强的细胞依赖性.
2019年, 他们使用了一个新的低温显微镜, 该系统可测量荧光图像上的每个像素中的完整发射光谱(图13(a)). 所有像素中的发射光谱通过奇异值分解法被进行综合性的分析, 由此得到与荧光物质相关的成分光谱(species-associated spectrum, SAS). 在成熟的叶片中, 他们可视化了可归属于PSII和PSI的SAS在细胞内的空间位置, 这与先前报道一致. 更多的是, 在经历了不同时长的绿化叶片切片中, 他们检测到荧光峰分别在677 nm (SAS677), 683 nm (SAS683), 685 nm (SAS685)和687 nm (SAS687)的4种成分. SAS677归属于Chla色素, 它是由PChld (protochlorophyllide)分子在一种还原酶的催化下通过光诱导而转化的直接产物. 该成分的归属是基于其光谱峰位置与之前报告的与PChld还原酶结合的Chla的相似性[168]. 另外, SAS677在连续2 h光照期间有逐渐减少的趋势, 这也支持了这一归属. SAS685在2 h的绿化期间也表现出逐渐减少, 表明其与两个光系统的早期中间体相关. 相反的是, SAS683和SAS687成分的含量在连续光照下逐渐增大. SAS683最先在MS细胞中, 因此可被归属于PSII的早期组装体. SAS687可被归属于PSI的早期组装体, 因为在2 h绿化后SAS687在BS细胞中增加; 其次, SAS687在740 nm波长处有一个宽的发射带, 这与成熟的PSI中红叶绿素的发射波长相似. 一个早期的研究支持了这一结论, 他们在8 h光照后的玉米叶片中分离的BS细胞中检测到了一个相似的荧光成分(发射峰为686 nm)[162]. 综上, 冷冻光谱显微技术表征了细胞体内特征微观区域的光系统发育过程, 这里所发现的SAS687和SAS683成分被认为是PSI和PSII早期组装体的候选者.
-
本文主要介绍了以冷冻光谱显微镜为硬件平台所开展的单分子光谱与单细胞光谱技术, 以及通过该技术在自然光合作用领域所取得的重要进展. 在该领域中, 仍有一些关键性的技术板块是有待突破的, 概述如下.
第一, 目前光学显微镜的空间分辨率无法完全解析光合成分的异质性分布和类囊体膜的动态, 因此提高分辨率是该领域需要长期努力的课题. 近年来, 一系列超分辨成像技术被不断发展, 包括单分子定位显微镜, 三维结构照明显微镜和受激发射损耗显微镜(stimulated emission depletion, STED)[169,170]. 从原理上看, STED技术是结合在共聚焦光谱显微镜上的最佳选择. 在STED方法中, 两束激光用于照射样品, 其中激发光将基态电子激发到激发态, 然后激发态电子在被空间调制过的STED光照射后返回基态(激发态删除), 这使得样品荧光点的点扩散函数变小. 为了尽量避免电子的二次激发, 通常选择远红光波长作为STED光. 然而, 由于光合成分具有多种不同的光谱形式, 使得用以删除各种成分的激发态所需要的合适波长不同, 因此将STED技术应用于光合作用研究是极具挑战性的工作. 冷冻光谱显微镜是嫁接STED技术的一个可能方案, 因为低温下电子基态的振动激发态减少, 这使STED光可能引发的不必要激发被限制. 另一方面, STED的空间分辨率仅依赖于光强度, 低温允许样品承受高强度光的连续照射, 因此可大大提高空间分辨率. 最近, 基于STED技术的冷冻显微成像似乎取得一些进展, 但光谱分解尚未实现[101]. 超分辨光谱显微技术可升级单分子和单细胞光谱的探测尺度, 能为光合作用研究带来新的突破, 尽管该技术的实现仍面临着许多挑战和困难.
第二, 另一个有待突破的技术是高速三维光谱显微成像. 三维光谱信息在评估细胞生物或厚组织中具有空间依赖性的荧光发射和重吸收效应等方面非常关键. 常规的三维光谱成像是通过光学切片来实现的, 但这里至少有两个限制: 首先, 该方法耗时过长, 这导致产生的随机热漂移难以被校正, 因此难以实现完整活细胞的三维光谱重建. 其次, 物体表层对激光强度的衰减和激光的波前变形使得物体深部区域的清晰三维图像变得难以获取. 最近, Kozawa等[171]使用一束经空间调制的 “光针” 作为激发光源, 通过波前工程调制的方案实现了高速三维动态成像. 虽然该方案尚未实现高光谱分解, 但为高速三维光谱显微镜的实现提供了一种可能性.
第三, 在光合作用研究中, 目前在微观区域内的时间分辨光谱探测是不足的. 虽然已在显微镜中实现单像素发射光谱与荧光寿命的同时获取, 但是该方法只能检测单波长的荧光寿命, 这让体内光合物质间的超快动力学分析受限[56]. 条纹照相机检测器能够提供一个全波长范围的时间分辨荧光测试, 是用作于评估色素蛋白中能量转移的常用工具[172]. 共聚焦显微镜与条纹照相机的结合有可能给该领域带来突破[173–175]. 然而, 因为条纹照相机的硬件限制, 数据采集所需的曝光时间过长(几十至几百秒), 这可能导致细胞荧光的快速漂白, 并极大地限制了显微镜的光栅扫描. 冷冻光学显微镜有潜力克服以上困难, 因为低温环境可延长样品的观测时间. 该方法的成功实施能让空间/波长/时间分辨的三维探测变为可能, 不仅可评估细胞体内超快荧光动力学的异质性, 还有可能促进单分子超快光谱的探测.
第四, 目前, 对天然单个色素蛋白复合体的探测是受到限制的, 因此能直接在天然膜上探测单分子的方案是被期待的. 最近, Yoneda和 Meredith等[176,177]开发了类囊体/人工膜杂化技术. 该方法将一种光聚合物粘连在基板上, 然后在指定区域使用紫外光照射使其聚合, 非聚合区域将被移除. 他们在约几十微米的非聚合区域成功地实现了天然类囊体膜的导入. 虽然对于导入的类囊体膜是否能执行完整的光合作用反应还有待验证, 但该方法对于使用冷冻光谱显微镜直接探膜上单分子的发展来说是有潜力的.
第五, 虽然单分子的各项荧光参数可固有地被考虑为反应其结构变化, 但由于色素蛋白分子的复杂性, 单分子光谱无法直接关联结构变化与功能异质性. 最近, 一些研究通过升级冷冻电镜算法解析了同种蛋白质的多种结构态, 并在关联这些结构态与单分子光谱上做出了尝试, 但仍充满了困难[80,178]. 因此, 能同时测量单分子动态和光谱的技术是期待被开展的. 高速原子力显微镜(atomic force microscope, AFM)具有高时空分辨率, 是能直接观测单分子动态的有力手段[179]. 随着桌面型高速AFM设备的发展, 光学显微镜与高速AFM的结合已被实现[180], 这为将来开发单分子光谱探测与高速AFM的联用技术奠定了基础.
近年来, 随着我国对冷冻电镜设备的大力投入, 使得结构生物学方向得以高速发展, 让我们在光合蛋白的结构解析的领域跻身国际一流梯队. 冷冻光谱显微镜是电镜技术的一种重要互补手段, 但在国内该技术几乎没有被开展. 冷冻显微光谱学在探究光合生物中光捕获与能量调控方面发挥着重要作用, 该领域值得被重视和更多关注.
感谢日本东北大学叶深教授、日本东北大学Yutaka Shibata教授、北京大学席鹏教授、中国科学院物理所陈海龙研究员、东华大学葛爱民研究员、南昌大学邵盛熙教授、宁波大学冷轩博士以及美国麻省理工学院王迪昊博士对本文的仔细阅读和给予的宝贵建议.
冷冻光学显微镜技术在光合作用研究中的发展与应用
Development and application of cryogenic optical microscopy in photosynthesis research
-
摘要: 在植物中, 光合作用的高效光电转化效率归因于色素蛋白复合体中所建立的强大捕光网络与灵活的能量转移机制. 对色素蛋白复合体结构与功能的解析是光合作用研究中的重要方向, 对人工光合作用研究和能源可持续发展战略具有指导意义. 随着冷冻电子显微镜的快速发展, 大量复合体的精密结构得以解析. 冷冻光学(光谱)显微镜是冷冻电子显微镜的重要互补技术, 发展至今已有约35年的历史. 该方法通过光谱特征可精确识别多种色素蛋白复合体, 而低温成像不仅有效地抑制了单个复合体或细胞样品的光损伤, 还限制了复合体中色素间的uphill能量转移, 从而提高荧光量子收率. 冷冻光学显微镜不仅成为表征单个蛋白质的结构动态与捕光功能的有力工具; 还为可视化和定量复杂的光合成分在细胞体内的空间分布提供了可能性. 因此, 该技术的应用极大地发展了在微观尺度下分析色素蛋白结构与功能的研究领域, 这对于光合作用研究体系的推进具有重大意义. 本文从单分子光谱与单细胞光谱技术两方面总结了冷冻光谱显微镜技术在自然光合作用中的主要应用与取得的成果, 其中包括探究色素蛋白复合体的捕光功能与蛋白质动态的关联, 复合体中能量异质性的表征, 在细胞体内可视化光合蛋白的能量调控机制等.Abstract: Efficient photosynthesis reaction is attributed to the flexible energy regulation of two important pigment-protein complexes, i.e. photosystem II (PSII) and photosystem I (PSI). Cryogenic spectral microscopy provides information about the spatial distribution and physiological functional states of photosynthetic components in photosynthetic organisms. Under low temperatures, the uphill energy transfer between pigments is efficiently suppressed so that the temperature-dependent PSI can be well analyzed. Therefore, a cryogenic spectral microscope allows us to discuss the physiological events surrounding PSII and PSI in the independent microscopic zones. This technique can be used to complement the insufficiencies of cryogenic electron microscopy and atomic force microscopy in analyzing the photophysics and photochemistry of photosynthetic species. Historically, cryogenic optical microscopes originated from the desire for single-molecule spectroscopy detection. So far, the combination of optical microscopies and various spectroscopic techniques has expanded the possibility of studying photosynthesis from multiple perspectives. In this paper, the important and recent progress of cryogenic spectral microscopy in the field of natural photosynthesis research is reviewed from two aspects: single-molecule spectroscopy and single-cell spectroscopy, and the advantages of this technique in clarifying the correlation between structure variability and function of pigment-protein complexes, as well as the physiological responses of photosynthetic organisms to variable environments, are also illustrated.
-
-
图 2 (a) 光合作用中电子转移的示意图[68], LHC表示捕光蛋白, PQ (plastoquinone)和PC (plastocyanin)分别代表两个主要的电子载体. (b), (c) 表示豌豆中PSII-LHCII (PDB code: 6 YP7)和PSI-LHCI (PDB code: 4 XK8)复合体的结构, Chl-a和Chl-b色素分子分别用绿色和紫色表示, 红色箭头表示假设的能量转移路径. (d), (e) PSII和PSI各自的反应中心结构. (f) 绿藻细胞Chlamydomonas中两个主要的光系统冷冻电镜结构, PSII-LHCII (PDB code: 6 KAF)和PSI-LHCI (PDB code: 6 JO6), 在PSII-LHCII中, 蓝绿色代表LHCII三聚体, 而深蓝色和紫色分别代表两个较小的LHCs (称为CP26和CP29), PSI-LHCI-LHCII (PDB code: 7DZ7)超级复合体除了连接少许的LHCI以外, 还额外结合了两个LHCII三聚体
Figure 2. (a) Electron flow of photosynthesis[68], LHC (light-harvesting complex), PQ (plastoquinone), PC (plastocyanin). (b), (c) Structures of PSII-LHCII (PDB code: 6 YP7) and PSI-LHCI (PDB code: 4 XK8) in the high plant. Core protein and light-harvesting protein were shown as yellow and blue-green, respectively. Chl-as and Chl-bs were indicated as green and purple, respectively. The red lines with arrows were the imaginary energy transfer pathway. (d), (e) Molecular structure of the reaction center of two PSs. (f) The Cryo-EM structures of PSII-LHCII (PDB code: 6 KAF) and PSI-LHCI (PDB code: 6 JO6) of Chlamydomonas cell. The PSI-LHCI-LHCII (PDB code: 7DZ7) supercomplex was easily formed upon state transition.
图 3 (a) T. elongatus中PSI三聚体晶体结构[70], 绿色表示Chl-a分子, 紫色表示反应中心P700, 红色分子代表着被预测的Red Chls; (b) PSI三聚体中的单体模型, Low2和Low1是Kato等[82]预测的Red Chls
Figure 3. (a) An X-ray crystal structure (PDB code: 1 JB0) of trimetric PSI isolated from a T. elongatus, the Chl-a was indicated in green, the reaction center was indicated in purple, the initial predicted red Chls candidates were depicted in red and marked by ellipses, they were renamed as Red Chls_1-3; (b) Low1 and Low2 were the possible Red Chls which were predicted by Kato et al.[82].
图 4 (a) 共聚焦荧光显微镜的简易示意图; (b) 自制冷冻光学方案[68], 绿色背景表示冷冻剂, 被斜线填充的空间表示真空环境; (c) 商业化冷冻光学方案, 在商业化的低温恒温器中, 样品被固定在真空环境里, 流动的冷冻剂(带箭头的线)通过一个导热性好的材料与样品架相连接
Figure 4. (a) Schematic of the confocal fluorescence microscope; (b) some home-built optical systems of cryogenic microscopes[68], the green background represents the refrigerant, and the space filled with diagonal lines represents the vacuum environment; (c) a cryogenic microscope equipped with a commercial cryostat, the sample is fixed in a vacuum environment, and a flowing cryogen (arrowed line) is connected to the sample holder through a material with good thermal conductivity.
图 5 变换点分析的模拟[119] (a)—(c) 使用不同时间间隔展示的单参数时程测量; (d) 使用变换点函数展示的数据; (e) 模拟的单参数时程测量数据, 该数据具有5个明确的状态, 可用于图(a)—(d)的模拟
Figure 5. Change-point analysis of single-parameter[119]: (a)–(c) Time-course single parameter trace with different intervals; (d) time-course single parameter trace used change-point analysis; (e) the simulated time-course single parameter trace with five states, this data was used for the simulations of panels (a)–(d).
图 6 (a) 奇异值矩阵分解的示意图, M是一个放置了大量光谱的数据集, U是放置SVD谱的正交矩阵, W和
$ {\boldsymbol{\varSigma}} $ 分别是放置奇异值SVλ的对角矩阵和一个转置矩阵; 在实际分析中, 一般选择前面较大的数值即可; (b)—(d) 一个通过奇异值矩阵分解分析的单细胞光谱显微测量例子[130]Figure 6. (a) Schematic form of SVD analysis of many spectra, M is a dataset with a large number of spectra, U is the orthogonal matrix with SVD spectra, W and
$ {\boldsymbol{\varSigma}} $ are the diagonal matrix with singular value SVλ and an transpose matrix, respectively; in practical analysis, the larger value in front is generally selected; (b)–(d) an example of SVD analysis in single-cell spectroscopy[130].图 7 (a) 旋涂制备法的流程图, 通过该方法制备的薄膜的厚度小于1 μm; (b) 涂层法的流程图, 其中常使用的表面钝化剂有PLL (Poly-L-lysine), BSA (bovine serum albumin)和PEG (polyethylene glycol)
Figure 7. (a) Processes of the spin-coat method preparation, by which the thickness of the film prepared is less than 1 μm; (b) the coating treatment method. The commonly used surface passivants include PLL (Poly-L-lysine), BSA (bovine serum albumin), and PEG (polyethylene glycol).
图 8 (a) 常规的激发光谱显微镜, 该系统通过单波长的激光扫描来获取激发光谱; (b) 基于线激光搭建的高速激发光谱显微镜[105], 在测量激发光谱时候, 该系统无需进行波长扫描; (c) 表示记录的激光光谱, 该光谱被用于激发光谱的校正
Figure 8. (a) Traditional excitation spectral microscope, acquisition of excitation spectra is completed by performing a wavelength scan; (b) a high-speed excitation spectral microscope[105], this system enables the rapid acquisition of excitation spectra without wavelength-scan; (c) the recorded spectrum of the line laser, this spectrum is necessary for the calibration of the excitation spectrum.
图 9 (a) LH2复合体中BChla分子的空间结构[27]; (b) LH2溶液的激发光谱和一个1.5 K的单分子激发光谱[27]; (c) B850的部分激子能级示意图. 其中, 左侧和右侧分别表示B850环上C9旋转对称存在或缺失的能级分布, 灰色和黑色双箭头线表示两个偏振正交的激子态; (d) 单个LH2的低温激发光谱; 灰色和黑色分别对应图(c)中的两个激子态[27]
Figure 9. (a) Arrangement of pigments of LH2 complex[27]; (b) the fluorescence excitation spectrum of LH2 bulk sample and a 1.5 K single-molecule excitation spectrum[27]; (c) the schematic of the B850 excitons with lower energy levels, the left and right sides represent the energy distribution of the part B850 excitons in the presence and absence of ninefold rotation symmetry; (d) the single-molecule spectra of the LH2 complex are taken at 1.5 K, the grey and black spectra may correspond to two orthogonal polarized excitations shown in panel (c)[27].
图 10 (a) 1.4 K 温度下对PSI单分子的发射光谱进行的时程观察, 顶部表示一个平均光谱; (b)两个波长区间的强度的时程变化; (c) 基于图(a)计算的二维自相关光谱图[142]
Figure 10. (a) A time-course observation of single PSI (T. elongatus) emission spectra, the top panel indicated an average emission spectrum; (b) the intensity changes in the two specific wavelength regions; (c) a 2-dimensional synchronous correlation spectroscopy calculated from panel (a)[142].
图 11 (a) 随机波动能量传递模型; (b) 分支能量传递模型; (c) 表示一个PSI三聚体的二维激发发射矩阵, 黑色和蓝色的实线是被计算的特征向量, 黑色的特征向量被用于计算倾斜角度α, ρ是激发-发射波长的相关性系数; (d) 表示两个从图(c)中重建的激发光谱, 蓝色和红色谱线分别代表发射波长覆盖在700—725 nm (Low1)和725—752 nm (Low2)区间的激发光谱[34]
Figure 11. (a) Model of random excitation energy transfer (EET) pathway of PSI; (b) the model of branched EET pathway of PSI; (c) a two-dimensional excitation-emission matrix of a selected PSI trimer 80 K, the black and blue solid lines respectively show the eigenvectors in which the black vector is used to calculate the inclination angle α, ρ indicates the correlation coefficient of excitation-emission wavelengths; (d) the two excitation spectra were reconstructed by integration over the red (700–725 nm, blue) and far-red (725–752 nm, red) emission wavelength regions[34].
图 12 叶绿体的冷冻显微镜观察, 在该叶绿体内的局部区域, 该观察获取了能很好甄别PSII和PSI特征的低温发射光谱[91]
Figure 12. Low-temperature observation of a chloroplast with a larger size, this study completed the spectral imaging of fluorescence emission and in some local regions obtained the spectra that can well identify PSII and PSI features[91].
图 13 (a) Shibata等[98]开发的冷冻光谱显微镜的示意图, 插图表示冷冻恒温仓的实拍图(由Yuki Fujita博士提供). (b) 该显微镜的最新版本的示意图, 该系统实现了单个像素上荧光光谱和寿命的同时获取[56]. (c) Chlamydomonas细胞图像上单像素中的低温发射光谱, 发射光谱能被5个高斯函数很好地拟合, 根据高斯峰位归属了LHCII, PSII和PSI的成分. 绿色点线表示680 nm滤波片的透射光谱[56]. (d) 各个光合成分在细胞内的分布图[56]. (e) PSII和PSI的比值图[56]. (f) 平均寿命图[56], 右侧为该细胞中一个像素上的荧光衰减曲线
Figure 13. (a) A cryogenic spectral microscope developed by Yutaka Shibata et al.[98], the illustration shows a photograph of the home-built cryostat that was provided by Dr. Yuki Fujita. (b) The latest version of this optical system, the present system enables the simultaneous acquisition of emission spectrum and lifetime on the same pixel[56]. (c) An emission spectrum selected from a pixel on a Chlamydomonas cell, the emission spectrum can be well fitted to the sum of five Gaussian functions. Based on the peak position of individual Gaussian curves, they assigned these Gaussian curves to LHCII, PSII, and PSI, respectively. The green dotted line indicates the transmission spectrum of a 680 nm bandpass filter[56]. (d) They visualized these photosynthetic components within a cell in terms of a method[56]. (e) The relative intensity ratio map of PSII and PSI[56]. (f) The average lifetime map of this cell, the right side shows a fluorescence curve selected from this cell[56].
图 14 (a) C4植物叶片横截面的示意图, 围绕着维管束(vascular bundle, VB)的细胞(bundle sheath, BS)中PSII的含量很低, 在远离VB的叶肉细胞(mesophyll, MS) 中, PSII的含量增大; (b) PSI的比值图; (c) 从PSI比值图中选出的两个代表性发射光谱, BS细胞中的光谱1展示了PSI的主要贡献, 相对于光谱1, MS细胞的光谱2中PSII的贡献显著性增大[61]
Figure 14. (a) Schematic of a leaf cross-section from a C4 plant, the bundle sheath (BS) cells with small PSII/PSI ratios are near the vascular bundle (VB), the mesophyll (MS) cells with large PSII/PSI ratios are far away from VB; (b) ratio map of PSI component; (c) low-temperature emission spectra were selected from two intracellular regions[61].
表 1 光谱信号的种类及其功能, 圆环和叉符号分别代表已实现和未实现的项目; 三角符号代表已实现但尚未使用在光合作用研究中的项目
Table 1. Type of spectral source used in the optical microscope, the circular and cross symbols represent completed and uncompleted projects, respectively; the triangle symbol represents projects that have been realized but have not yet been used in photosynthesis research.
信号源 主要功能 单细胞检测 单分子探测 低温 吸收光谱 叶绿素分子的电子基态, 绝对分子浓度的评估 ◎ × × 荧光激发光谱 捕光天线蛋白的探测, 能量传递路径的定性分析 ◎ ◎ ◎ 荧光发射光谱 两个光系统的表征, 荧光淬灭的表征 ◎ ◎ ◎ 荧光寿命 发射分子的激发态动力学表征, 量子收率的测量 ◎ ◎ ◎ 自发拉曼光谱 检测弱荧光或非荧光分子, 例如胡萝卜素等 ◎ Δ Δ -
[1] Butler W, Kitajima M 1975 Biochim. Biophys. Acta 396 72 doi: 10.1016/0005-2728(75)90190-5 [2] Wendler J, Holzwarth A R 1987 Biophys. J. 52 717 doi: 10.1016/S0006-3495(87)83266-6 [3] Delosme R, Olive J, Wollman F A 1996 Biochim. Biophys. Acta 1273 150 doi: 10.1016/0005-2728(95)00143-3 [4] Koblížek M, Komenda J, Masojídek J 1998 Photosynthesis: Mechanisms and Effects (Springer) pp213–216 [5] Iwai M, Yokono M, Inada N, Minagawa J 2010 Proc. Natl. Acad. Sci. U. S. A. 107 2337 doi: 10.1073/pnas.0908808107 [6] Minagawa J 2011 Biochim. Biophys. Acta 1807 897 doi: 10.1016/j.bbabio.2010.11.005 [7] Tian L J, Dinc E, Croce R 2015 J. Phys. Chem. Lett. 6 2339 doi: 10.1021/acs.jpclett.5b00944 [8] Bressan M, Dall'Osto L, Bargigia I, Alcocer M J, Viola D, Cerullo G, D'Andrea C, Bassi R, Ballottari M 2016 Nat. Plants 2 16131 doi: 10.1038/nplants.2016.131 [9] Dinc E, Tian L, Roy L M, Roth R, Goodenough U, Croce R 2016 Proc. Natl. Acad. Sci. U. S. A. 113 7673 doi: 10.1073/pnas.1605380113 [10] Dickson R M, Cubitt A B, Tsien R Y, Moerner W E 1997 Nature 388 355 doi: 10.1038/41048 [11] Jelezko F, Tietz C, Gerken U, Wrachtrup J, Bittl R 2000 J. Phys. Chem. B 104 8093 doi: 10.1021/jp001332t [12] Brecht M, Studier H, Radics V, Nieder J B, Bittl R 2008 J. Am. Chem. Soc. 130 17487 doi: 10.1021/ja806216p [13] Kunz R, Timpmann K, Southall J, Cogdell R J, Kohler J, Freiberg A 2013 J. Phys. Chem. B 117 12020 doi: 10.1021/jp4073697 [14] Schorner M, Beyer S R, Southall J, Cogdell R J, Kohler J 2015 J. Phys. Chem. B 119 13964 doi: 10.1021/acs.jpcb.5b07494 [15] Kondo T, Gordon J B, Pinnola A, Dall’Osto L, Bassi R, Schlau-Cohen G S 2019 Proc. Natl. Acad. Sci. U. S. A. 116 11247 doi: 10.1073/pnas.1821207116 [16] Kondo T, Mutoh R, Arai S, Kurisu G, Oh-Oka H, Fujiyoshi S, Matsushita M 2022 J. Chem. Phys. 156 105102 doi: 10.1063/5.0077290 [17] Kondo T, Shibata Y 2022 Biophys. Physicobiol. 19 e190013 doi: 10.2142/biophysico.bppb-v19.0013 [18] Li H, Wang Y G, Ye M P, Li S S, Li D Y, Ren H S, Wang M H, Du L C, Li H, Veglia G, G J L, Weng Y X 2020 Sci. China Chem. 63 1121 doi: 10.1007/s11426-020-9771-2 [19] Ruan M X, Li H, Zhang Y, Zhao R Q, Zhang J, Wang Y J, Gao J L, Wang Z, Wang Y X, Sun D P, Ding W, Weng Y X 2023 Nat. Plants 9 1547 doi: 10.1038/s41477-023-01500-2 [20] Elber R, Karplus M 1987 Science 235 318 doi: 10.1126/science.3798113 [21] Frauenfelder H, Sligar S G, Wolynes P G 1991 Science 254 1598 doi: 10.1126/science.1749933 [22] Hofmann C, Aartsma T J, Michel H, Köhler J 2003 Proc. Natl. Acad. Sci. U. S. A. 100 15534 doi: 10.1073/pnas.2533896100 [23] Bopp M A, Jia Y W, Li L Q, Cogdell R J, Hochstrasser R M 1997 Proc. Natl. Acad. Sci. U. S. A. 94 10630 doi: 10.1073/pnas.94.20.10630 [24] Rutkauskas D, Novoderezkhin V, Cogdell R J, van Grondelle R 2004 Biochemistry 43 4431 doi: 10.1021/bi0497648 [25] Elli A F, Jelezko F, Tietz C, Studier H, Brecht M, Bittl R, Wrachtrup J 2006 Biochemistry 45 1454 doi: 10.1021/bi0521700 [26] Brecht M, Studier H, Elli A F, Jelezko F, Bittl R 2007 Biochemistry 46 799 doi: 10.1021/bi061975k [27] Matsushita M 2008 Seibutsu Butsuri 48 035 doi: 10.2142/biophys.48.035 [28] Brotosudarmo T H, Kunz R, Bohm P, Gardiner A T, Moulisova V, Cogdell R J, Kohler J 2009 Biophys. J. 97 1491 doi: 10.1016/j.bpj.2009.06.034 [29] Kondo T, Chen W J, Schlau-Cohen G S 2017 Chem. Rev. 117 860 doi: 10.1021/acs.chemrev.6b00195 [30] Squires A H, Wang Q, Moerner W 2017 Biophys. J. 112 471a [31] Hatazaki S, Sharma D K, Hirata S, Nose K, Iyoda T, Kölsch A, Lokstein H, Vacha M 2018 J. Phys. Chem. Lett. 9 6669 doi: 10.1021/acs.jpclett.8b03064 [32] Jana S, Du T, Nagao R, Noguchi T, Shibata Y 2019 Biochim. Biophys. Acta 1860 30 doi: 10.1016/j.bbabio.2018.11.002 [33] Otomo K, Dewa T, Matsushita M, Fujiyoshi S 2023 J. Phys. Chem. B 127 4959 doi: 10.1021/acs.jpcb.3c00284 [34] Zhang X J, Taniguchi R, Nagao R, Tomo T, Noguchi T, Ye S, Shibata Y 2024 J. Phys. Chem. B 128 2664 doi: 10.1021/acs.jpcb.3c07789 [35] Müller P, Li X P, Niyogi K K 2001 Plant Physiol. 125 1558 doi: 10.1104/pp.125.4.1558 [36] Wollman F A 2001 EMBO J 20 3623 doi: 10.1093/emboj/20.14.3623 [37] Kanazawa A, Kramer D M 2002 Proc. Natl. Acad. Sci. U. S. A. 99 12789 doi: 10.1073/pnas.182427499 [38] Rozak P, Seiser R, Wacholtz W, Wise R 2002 Plant Cell Environ. 25 421 doi: 10.1046/j.0016-8025.2001.00823.x [39] Bellafiore S, Barneche F, Peltier G, Rochaix J D 2005 Nature 433 892 doi: 10.1038/nature03286 [40] Wientjes E, van Amerongen H, Croce R 2013 Biochim. Biophys. Acta 1827 420 doi: 10.1016/j.bbabio.2012.12.009 [41] Ünlü C, Drop B, Croce R, van Amerongen H 2014 Proc. Natl. Acad. Sci. U. S. A. 111 3460 doi: 10.1073/pnas.1319164111 [42] Kim E, Ahn T K, Kumazaki S 2015 Plant Cell Physiol. 56 759 doi: 10.1093/pcp/pcv004 [43] Nawrocki W J, Santabarbara S, Mosebach L, Wollman F A, Rappaport F 2016 Nat. Plants 2 16031 doi: 10.1038/nplants.2016.31 [44] de la Cruz Valbuena G, VA Camargo F, Borrego-Varillas R, Perozeni F, D’Andrea C, Ballottari M, Cerullo G 2019 J. Phys. Chem. Lett. 10 2500 doi: 10.1021/acs.jpclett.9b01184 [45] Calzadilla P I, Kirilovsky D 2020 Photochem. Photobiol. Sci. 19 585 doi: 10.1039/c9pp00451c [46] Huang Z H, Shen L L, Wang W D, Mao Z Y, Yi X H, Kuang T Y, Shen J R, Zhang X, Han G Y 2021 Nat. Commun. 12 1100 doi: 10.1038/s41467-021-21362-6 [47] Huokko T, Ni T, Dykes G F, Simpson D M, Brownridge P, Conradi F D, Beynon R J, Nixon P J, Mullineaux C W, Zhang P 2021 Nat. Commun. 12 3475 doi: 10.1038/s41467-021-23680-1 [48] Zhang X J, Fujita Y, Tokutsu R, Minagawa J, Ye S, Shibata Y 2021 Plant Cell Physiol. 62 872 doi: 10.1093/pcp/pcab047 [49] Zhang X J, Fujita Y, Kaneda N, Tokutsu R, Ye S, Minagawa J, Shibata Y 2022 Proc. Natl. Acad. Sci. U. S. A. 119 e2122032119 doi: 10.1073/pnas.2122032119 [50] Hamel P, Olive J, Pierre Y, Wollman F A, de Vitry C 2000 J. Biol. Chem. 275 17072 doi: 10.1074/jbc.M001468200 [51] Finazzi G, Rappaport F, Furia A, Fleischmann M, Rochaix J D, Zito F, Forti G 2002 EMBO Rep. 3 280 doi: 10.1093/embo-reports/kvf047 [52] Munekage Y, Hojo M, Meurer J, Endo T, Tasaka M, Shikanai T 2002 Cell 110 361 doi: 10.1016/S0092-8674(02)00867-X [53] DalCorso G, Pesaresi P, Masiero S, Aseeva E, Schünemann D, Finazzi G, Joliot P, Barbato R, Leister D 2008 Cell 132 273 doi: 10.1016/j.cell.2007.12.028 [54] Krause G, Briantais J M, Vernotte C 1983 Biochim. Biophys. Acta 723 169 doi: 10.1016/0005-2728(83)90116-0 [55] Fujita Y, Ito W, Washiyama K, Shibata Y 2018 J. Photochem. Photobiol. B: Biol. 185 111 doi: 10.1016/j.jphotobiol.2018.05.029 [56] Fujita Y, Zhang X J, Mohamed A, Ye S, Shibata Y 2022 J. Photochem. Photobiol. B: Biol. 236 112584 doi: 10.1016/j.jphotobiol.2022.112584 [57] Uniacke J, Zerges W 2007 Plant Cell 19 3640 doi: 10.1105/tpc.107.054882 [58] Nixon P J, Michoux F, Yu J, Boehm M, Komenda J 2010 Ann. Bot. 106 1 doi: 10.1093/aob/mcq059 [59] Nickelsen J, Rengstl B 2013 Annu. Rev. Plant Biol. 64 609 doi: 10.1146/annurev-arplant-050312-120124 [60] Nellaepalli S, Ozawa S I, Kuroda H, Takahashi Y 2018 Nat. Commun. 9 2439 doi: 10.1038/s41467-018-04823-3 [61] Chiba T, Shibata Y 2019 Biochim. Biophys. Acta 1860 148090 doi: 10.1016/j.bbabio.2019.148090 [62] Croce R, Van Amerongen H 2014 Nat. Chem. Biol. 10 492 doi: 10.1038/nchembio.1555 [63] Suvorov N, Pogorilyy V, Diachkova E, Vasil’ev Y, Mironov A, Grin M 2021 Int. J. Mol. Sci. 22 6392 doi: 10.3390/ijms22126392 [64] Oviedo M B, Sánchez C G 2011 J. Phys. Chem. A 115 12280 doi: 10.1021/jp203826q [65] Fuchs B M, Spring S, Teeling H, Quast C, Wulf J, Schattenhofer M, Yan S, Ferriera S, Johnson J, Glöckner F O 2007 Proc. Natl. Acad. Sci. U. S. A. 104 2891 doi: 10.1073/pnas.0608046104 [66] Wasielewski M R, Kispert L D 1986 Chem. Phys. Lett. 128 238 doi: 10.1016/0009-2614(86)80332-3 [67] Son M, Hart S M, Schlau-Cohen G S 2021 Trends Chem. 3 733 doi: 10.1016/j.trechm.2021.05.008 [68] Zhang X J, Shibata Y, Kumazaki S 2023 J. Photochem. Photobiol. C. 56 100616 doi: 10.1016/j.jphotochemrev.2023.100616 [69] Suga M, Ozawa S I, Yoshida-Motomura K, Akita F, Miyazaki N, Takahashi Y 2019 Nat. Plants 5 626 doi: 10.1038/s41477-019-0438-4 [70] Jordan P, Fromme P, Witt H T, Klukas O, Saenger W, Krauß N 2001 Nature 411 909 doi: 10.1038/35082000 [71] Brecht M 2009 Mol. Phys. 107 1955 doi: 10.1080/00268970902859777 [72] Hussels M, Brecht M 2011 Biochemistry 50 3628 doi: 10.1021/bi2000615 [73] Köhler J, Cogdell R J 2019 Metabolism, Structure and Function of Plant Tetrapyrroles: Control Mechanisms of Chlorophyll Biosynthesis and Analysis of Chlorophyll-Binding Proteins pp1–31 [74] Hayes J, Matsuzaki S, Rätsep M, Small G 2000 J. Phys. Chem. B 104 5625 doi: 10.1021/jp000447u [75] Rätsep M, Johnson T, Chitnis P, Small G 2000 J. Phys. Chem. B 104 836 doi: 10.1021/jp9929418 [76] Zazubovich V, Matsuzaki S, Johnson T, Hayes J, Chitnis P, Small G 2002 Chem. Phys. 275 47 doi: 10.1016/S0301-0104(01)00535-3 [77] Riley K J, Reinot T, Jankowiak R, Fromme P, Zazubovich V 2007 J. Phys. Chem. B 111 286 doi: 10.1021/jp062664m [78] Skandary S, Konrad A, Hussels M, Meixner A J, Brecht M 2015 J. Phys. Chem. B 119 13888 doi: 10.1021/acs.jpcb.5b04483 [79] Kato K, Shinoda T, Nagao R, Akimoto S, Suzuki T, Dohmae N, Chen M, Allakhverdiev S I, Shen J R, Akita F 2020 Nat. Commun. 11 238 doi: 10.1038/s41467-019-13898-5 [80] Toporik H, Khmelnitskiy A, Dobson Z, Riddle R, Williams D, Lin S, Jankowiak R, Mazor Y 2020 Nat. Commun. 11 5279 doi: 10.1038/s41467-020-18884-w [81] Çoruh O, Frank A, Tanaka H, Kawamoto A, El-Mohsnawy E, Kato T, Namba K, Gerle C, Nowaczyk M M, Kurisu G 2021 Commun. Biol. 4 304 doi: 10.1038/s42003-021-01808-9 [82] Kato K, Hamaguchi T, Nagao R, Kawakami K, Ueno Y, Suzuki T, Uchida H, Murakami A, Nakajima Y, Yokono M 2022 Elife 11 e73990 doi: 10.7554/eLife.73990 [83] Akita F, Nagao R, Kato K, Nakajima Y, Yokono M, Ueno Y, Suzuki T, Dohmae N, Shen J R, Akimoto S 2020 Commu. Biol. 3 232 doi: 10.1038/s42003-020-0949-6 [84] Klar T A, Jakobs S, Dyba M, Egner A, Hell S W 2000 Proc. Natl. Acad. Sci. U. S. A. 97 8206 doi: 10.1073/pnas.97.15.8206 [85] Giske A 2007 Ph. D. Dissertation (Heidelberg: Heidelberg University [86] Ambrose W P, Basche T, Moerner W E 1991 J. Chem. Phys. 95 7150 doi: 10.1063/1.461392 [87] Vacha M, Yokoyama H, Tokizaki T, Furuki M, Tani T 1999 Rev. Sci. Instrum. 70 2041 doi: 10.1063/1.1149707 [88] Jasny J, Sepiol J, Irngartinger T, Traber M, Renn A, Wild U P 1996 Rev. Sci. Instrum. 67 1425 doi: 10.1063/1.1146868 [89] Fujiyoshi S, Fujiwara M, Kim C, Matsushita M, van Oijen A M, Schmidt J 2007 Appl. Phys. Lett. 91 051125 doi: 10.1063/1.2767778 [90] Vàcha F, Vacha M, Bumba L, Hashizume K, Tani T 2000 Photosynthetica 38 493 doi: 10.1023/A:1012492919852 [91] Vácha F, Sarafis V, Benediktyová Z, Bumba L, Valenta J, Vácha M, Sheue C R, Nedbal L 2007 Micron 38 170 doi: 10.1016/j.micron.2006.07.013 [92] Sugiura K, Itoh S 2012 Plant Cell Physiol. 53 1492 doi: 10.1093/pcp/pcs093 [93] Breunig H G, Tumer F, Konig K 2013 J. Biophotonics 6 622 doi: 10.1002/jbio.201200093 [94] Shibata Y, Katoh W, Tahara Y 2013 Biochim. Biophys. Acta 1827 520 doi: 10.1016/j.bbabio.2013.02.001 [95] Steinbach G, Schubert F, Kana R 2015 J. Photochem. Photobiol. B: Biol. 152 395 doi: 10.1016/j.jphotobiol.2015.10.003 [96] Fujiwara M, Ishii T, Ishida K, Toratani Y, Furubayashi T, Matsushita M, Fujiyoshi S 2019 Appl. Phys. Lett. 115 033701 doi: 10.1063/1.5110546 [97] Yoshimoto M 2000 Jpn. J. Appl. Phys. 39 6105 doi: 10.1143/JJAP.39.6105 [98] Shibata Y, Katoh W, Chiba T, Namie K, Ohnishi N, Minagawa J, Nakanishi H, Noguchi T, Fukumura H 2014 Biochim. Biophys. Acta 1837 880 doi: 10.1016/j.bbabio.2014.03.006 [99] Metzger M, Konrad A, Skandary S, Ashraf I, Meixner A J, Brecht M 2016 Opt. Express 24 13023 doi: 10.1364/OE.24.013023 [100] Wang L, Bateman B, Zanetti-Domingues L C, Moores A N, Astbury S, Spindloe C, Darrow M C, Romano M, Needham S R, Beis K, Rolfe D J, Clarke D T, Martin-Fernandez M L 2019 Commun. Biol. 2 74 doi: 10.1038/s42003-019-0317-6 [101] Huebinger J, Grecco H, Masip M E, Christmann J, Fuhr G R, Bastiaens P I H 2021 Sci. Adv. 7 eabk0882 doi: 10.1126/sciadv.abk0882 [102] Yamanaka M, Tsuji K, Kumamoto Y, Tamura S, Miyamura W, Kubo T, Mizushima K, Kono K, Hirano H, Sugiura K 2023 bioRxiv 2023.08. 01.551103 [103] Hasegawa M, Yoshida T, Yabuta M, Terazima M, Kumazaki S 2011 J. Phys. Chem. B 115 4184 doi: 10.1021/jp111306k [104] van Oijen A M, Ketelaars M, Kohler J, Aartsma T J, Schmidt J 1999 Science 285 400 doi: 10.1126/science.285.5426.400 [105] Jana S, Shibata Y 2020 Biophys. J. 118 36 doi: 10.1016/j.bpj.2019.11.023 [106] Girolomoni L, Cazzaniga S, Pinnola A, Perozeni F, Ballottari M, Bassi R 2019 Proc. Natl. Acad. Sci. U. S. A. 116 4212 doi: 10.1073/pnas.1809812116 [107] Kondo T, Pinnola A, Chen W J, Dall'Osto L, Bassi R, Schlau-Cohen G S 2017 Nat. Chem. 9 772 doi: 10.1038/nchem.2818 [108] Barzda V, de Grauw C J, Vroom J, Kleima F J, van Grondelle R, van Amerongen H, Gerritsen H C 2001 Biophys. J. 81 538 doi: 10.1016/S0006-3495(01)75720-7 [109] Barzda V, Gulbinas V, Kananavicius R, Cervinskas V, Van Amerongen H, Van Grondelle R, Valkunas L 2001 Biophys. J. 80 2409 doi: 10.1016/S0006-3495(01)76210-8 [110] Robert B 2009 Photosynth. Res. 101 147 doi: 10.1007/s11120-009-9440-4 [111] Tamamizu K, Kumazaki S 2019 Biochim. Biophys. Acta 1860 78 doi: 10.1016/j.bbabio.2018.11.012 [112] Tamamizu K, Sakamoto T, Kurashige Y, Nozue S, Kumazaki S 2023 Spectrochimica Acta. Part A 290 122258 doi: 10.1016/j.saa.2022.122258 [113] Wu H W, Volponi J V, Oliver A E, Parikh A N, Simmons B A, Singh S 2011 Proc. Natl. Acad. Sci. U. S. A. 108 3809 doi: 10.1073/pnas.1009043108 [114] Gall A, Pascal A A, Robert B 2015 Biochim. Biophys. Acta 1847 12 doi: 10.1016/j.bbabio.2014.09.009 [115] Mizushima K, Kumamoto Y, Tamura S, Yamanaka M, Mochizuki K, Li M, Egoshi S, Dodo K, Harada Y, Smith N I 2023 bioRxiv 2023.11. 15.567077 [116] Jiang S, Zhang Y, Zhang R, Hu C R, Liao M H, Luo Y, Yang J L, Dong Z C, Hou J G 2015 Nat. Nanotechnol. 10 865 doi: 10.1038/nnano.2015.170 [117] Hasegawa M, Shiina T, Terazima M, Kumazaki S 2010 Plant Cell Physiol. 51 225 doi: 10.1093/pcp/pcp182 [118] Gitelson A A, Buschmann C, Lichtenthaler H K 1998 J. Plant Physiol. 152 283 doi: 10.1016/S0176-1617(98)80143-0 [119] Watkins L P, Yang H 2005 J. Phys. Chem. B 109 617 doi: 10.1021/jp0467548 [120] Schlau-Cohen G S, Wang Q, Southall J, Cogdell R J, Moerner W 2013 Proc. Natl. Acad. Sci. U. S. A. 110 10899 doi: 10.1073/pnas.1310222110 [121] Schlau-Cohen G S, Yang H-Y, Krüger T P, Xu P, Gwizdala M, Van Grondelle R, Croce R, Moerner W 2015 J. Phys. Chem. Lett. 6 860 doi: 10.1021/acs.jpclett.5b00034 [122] Goldsmith R H, Moerner W 2010 Nat. Chem. 2 179 doi: 10.1038/nchem.545 [123] Allorent G, Lefebvre-Legendre L, Chappuis R, Kuntz M, Truong T B, Niyogi K K, Ulm R, Goldschmidt-Clermont M 2016 Proc. Natl. Acad. Sci. U. S. A. 113 14864 doi: 10.1073/pnas.1607695114 [124] Tokutsu R, Fujimura-Kamada K, Yamasaki T, Okajima K, Minagawa J 2021 Plant Physiol. 185 1894 doi: 10.1093/plphys/kiab004 [125] Horton P, Wentworth M, Ruban A 2005 FEBS Lett. 579 4201 doi: 10.1016/j.febslet.2005.07.003 [126] Liguori N, Roy L M, Opacic M, Durand G, Croce R 2013 J. Am. Chem. Soc. 135 18339 doi: 10.1021/ja4107463 [127] Holub O, Seufferheld M J, Gohlke C, Govindjee, Clegg R M 2000 Photosynthetica 38 581 doi: 10.1023/A:1012465508465 [128] Holub O, Seufferheld M, Gohlke C, Govindjee, Heiss G, Clegg R 2007 J. Microsc. 226 90 doi: 10.1111/j.1365-2818.2007.01763.x [129] Kumazaki S, Akari M, Hasegawa M 2013 Plant Physiology 161 1321 doi: 10.1104/pp.112.206680 [130] Nozue S, Mukuno A, Tsuda Y, Shiina T, Terazima M, Kumazaki S 2016 Biochim. Biophys. Acta 1857 46 doi: 10.1016/j.bbabio.2015.10.003 [131] Tamarat P, Maali A, Lounis B, Orrit M 2000 J. Phys. Chem. A 104 1 doi: 10.1021/jp992505l [132] Kaufmann R, Hagen C, Grunewald K 2014 Curr. Opin. Chem. Biol. 20 86 doi: 10.1016/j.cbpa.2014.05.007 [133] Nabhan M A, Silvera Batista C A, Cliffel D E, Jennings G K 2023 ACS Appl. Polym. Mater. 5 3278 doi: 10.1021/acsapm.2c02126 [134] Gust A, Zander A, Gietl A, Holzmeister P, Schulz S, Lalkens B, Tinnefeld P, Grohmann D 2014 Molecules 19 15824 doi: 10.3390/molecules191015824 [135] Paul T, Myong S 2022 STAR Protocols 3 101152 doi: 10.1016/j.xpro.2022.101152 [136] Cohen A E, Moerner W 2005 Appl. Phys. Lett. 86 093109 doi: 10.1063/1.1872220 [137] Brotosudarmo T H P, Wittmann B, Seki S, Fujii R, Köhler J r 2022 J. Phys. Chem. Lett. 13 5226 doi: 10.1021/acs.jpclett.2c01160 [138] Basché T, Moerner W, Orrit M, Talon H 1992 Phys. Rev. Lett. 69 1516 doi: 10.1103/PhysRevLett.69.1516 [139] Hussels M, Brecht M 2011 FEBS Lett. 585 2445 doi: 10.1016/j.febslet.2011.06.018 [140] Zhang X J, Fujita Y, Kaneda N, Tokutsu R, Ye S, Minagawa J, Shibata Y 2021 bioRxiv 2021.10. 27.465227 [141] Moerner W, Fromm D P 2003 Rev. Sci. Instrum. 74 3597 doi: 10.1063/1.1589587 [142] Brecht M, Radics V, Nieder J B, Bittl R 2009 Proc. Natl. Acad. Sci. U. S. A. 106 11857 doi: 10.1073/pnas.0903586106 [143] Ishii K, Tahara T 2013 J. Phys. Chem. B 117 11423 doi: 10.1021/jp406864e [144] Ishii K, Tahara T 2013 J. Phys. Chem. B 117 11414 doi: 10.1021/jp406861u [145] Sarkar B, Ishii K, Tahara T 2024 J. Phys. Chem. B 128 4685 doi: 10.1021/acs.jpcb.4c01224 [146] Wang D H, Fiebig O C, Harris D, Toporik H, Ji Y, Chuang C, Nairat M, Tong A L, Ogren J I, Hart S M 2023 Proc. Natl. Acad. Sci. U. S. A. 120 e2220477120 doi: 10.1073/pnas.2220477120 [147] Kunz R, Timpmann K, Southall J, Cogdell R J, Freiberg A, Kohler J 2014 Biophys. J. 106 2008 doi: 10.1016/j.bpj.2014.03.023 [148] 翁羽翔 2007 物理 36 816 doi: 10.3321/j.issn:0379-4148.2007.11.003 Weng Y X 2007 Physics 36 816 doi: 10.3321/j.issn:0379-4148.2007.11.003 [149] Kunz R, Timpmann K, Southall J, Cogdell R J, Freiberg A, Köhler J r 2012 J. Phys. Chem. B 116 11017 doi: 10.1021/jp3040456 [150] Kunz R, Timpmann K, Southall J, Cogdell R J, Freiberg A, Köhler J 2013 Angew. Chem. 52 8726 doi: 10.1002/anie.201303231 [151] 冷轩, 王专, 翁羽翔 2016 植物生理学报 52 1681 Leng X, Wang Z, Weng Y X 2016 Plant Physiol. J. 52 1681 [152] Hildner R, Brinks D, Nieder J B, Cogdell R J, Van Hulst N F 2013 Science 340 1448 doi: 10.1126/science.1235820 [153] Pålsson L-O, Dekker J P, Schlodder E, Monshouwer R, van Grondelle R 1996 Photosynth. Res. 48 239 doi: 10.1007/BF00041014 [154] Beekman L M, Frese R N, Fowler G J, Picorel R, Cogdell R J, van Stokkum I H, Hunter C N, van Grondelle R 1997 J. Phys. Chem. B 101 7293 doi: 10.1021/jp963447w [155] Schlodder E, Hussels M, Çetin M, Karapetyan N V, Brecht M 2011 Biochim. Biophys. Acta 1807 1423 doi: 10.1016/j.bbabio.2011.06.018 [156] Liu Z F, Yan C H, Wang K B, Kuang T Y, Zhang J P, Gui L L, An X M, Chang W R 2004 Nature 428 287 doi: 10.1038/nature02373 [157] Snellenburg J J, Wlodarczyk L M, Dekker J P, van Grondelle R, van Stokkum I H 2017 Biochim. Biophys. Acta 1858 64 doi: 10.1016/j.bbabio.2016.10.001 [158] Nellaepalli S, Ozawal S I, Kuroda H, Takahashi Y 2018 Nat. Commun. 9 1 doi: 10.1038/s41467-017-02088-w [159] Ozawa S I, Onishi T, Takahashi Y 2010 J. Biol. Chem. 285 20072 doi: 10.1074/jbc.M109.098954 [160] Shao S X 2017 Ph. D. Dissertation (London: Imperial College London [161] Shibata K 1957 J. Biochem. 44 147 doi: 10.1093/oxfordjournals.jbchem.a126741 [162] Schuster G, Ohad I, Martineau B, Taylor W C 1985 J. Biol. Chem. 260 11866 doi: 10.1016/S0021-9258(17)39111-1 [163] Franck F, Eullaffroy P, Popovic R 1997 Photosynth. Res. 51 107 doi: 10.1023/A:1005767526448 [164] Sundqvist C, Dahlin C 1997 Physiol. Plant. 100 748 doi: 10.1111/j.1399-3054.1997.tb00002.x [165] Marchand M, Dewez D, Franck F, Popovic R 2004 J. Photochem. Photobiol. B: Biol. 75 73 doi: 10.1016/j.jphotobiol.2004.05.008 [166] Amirjani M, Sundqvist C 2006 Photosynthetica 44 83 doi: 10.1007/s11099-005-0162-3 [167] Peng L W, Ma J F, Chi W, Guo J K, Zhu S Y, Lu Q T, Lu C M, Zhang L X 2006 Plant Cell 18 955 doi: 10.1105/tpc.105.037689 [168] Kovacevic D, Dewez D, Popovic R 2007 Photosynthetica 45 105 doi: 10.1007/s11099-007-0016-2 [169] Hell S W, Wichmann J 1994 Opt. Lett. 19 780 doi: 10.1364/OL.19.000780 [170] Blom H, Widengren J 2017 Chem. Rev. 117 7377 doi: 10.1021/acs.chemrev.6b00653 [171] Kozawa Y, Nakamura T, Uesugi Y, Sato S 2022 Biomed. Opt. Express 13 1702 doi: 10.1364/BOE.449329 [172] Komura M, Itoh S 2009 Photosynth. Res. 101 119 doi: 10.1007/s11120-009-9463-x [173] Kusumi A, Tsuji A, Murata M, Sako Y, Yoshizawa A C, Kagiwada S, Hayakawa T, Ohnishi S 1991 Biochemistry 30 6517 doi: 10.1021/bi00240a024 [174] Biskup C, Zimmer T, Benndorf K 2004 Nat. Biotechnol. 22 220 doi: 10.1038/nbt935 [175] Liu L X, Li Y H, Sun L G, Li H, Peng X, Qu J L Multiphoton Microscopy in the Biomedical Sciences XIV pp307-311 [176] Yoneda T, Tanimoto Y, Takagi D, Morigaki K 2020 Langmuir 36 5863 doi: 10.1021/acs.langmuir.0c00613 [177] Meredith S A, Yoneda T, Hancock A M, Connell S D, Evans S D, Morigaki K, Adams P G 2021 Small 17 2006608 doi: 10.1002/smll.202006608 [178] Harris D, Toporik H, Schlau-Cohen G S, Mazor Y 2023 Nat. Commun. 14 4650 doi: 10.1038/s41467-023-40146-8 [179] Onoa B, Fukuda S, Iwai M, Bustamante C, Niyogi K K 2020 Biophys. J. 118 1876 doi: 10.1016/j.bpj.2020.02.029 [180] Yang K S, Chan F Y, Watanabe H, Yoshioka S, Inouye Y, Uchihashi T, Ishitobi H, Verma P, Umakoshi T 2024 Nano Lett. 24 2805 doi: 10.1021/acs.nanolett.3c04877 -

 首页
首页 登录
登录 注册
注册


 下载:
下载: