-
DNA 是生物体内携带遗传信息的核心物质, 主要存在于细胞核和线粒体中[1]. DNA通过特定的碱基序列编码遗传指令, 指导蛋白质合成并传递遗传特征. 值得注意的是, DNA稳定性受多种因素的协同调控[2]. 系统解析其作用机制, 可以揭示DNA动态构象在生命活动中的功能切换规律, 也可为生物技术中核酸分子稳定性优化提供理论框架.
DNA的稳定性首先由其碱基序列的化学拓扑特性决定, G≡C碱基对(base pair, bp)较A = T具有更高的热力学稳定性[3,4]. 此外, DNA稳定性受溶液离子强度和温度的影响. 传统观点认为单价阳离子能够通过静电屏蔽作用减弱DNA磷酸骨架间的电荷排斥, 从而增强双螺旋结构的稳定性[5]. 然而, 最新研究表明, 当单价阳离子浓度超过临界值时, 其过电荷效应(overcharging)反而会削弱双链稳定性[6]. 升温则会触发DNA熔解[7].
单链DNA(single-stranded DNA, ssDNA)在温度下降过程中容易自发折叠形成局部互补配对的非功能性DNA发夹(hairpin)二级结构, 干扰转录延伸复合体的进程. DNA发夹由双链茎部(stem, 通过碱基互补配对稳定)和单链环部(loop)构成, 其动态构象转变与双螺旋DNA稳定性有关[8]. 茎部长度与G≡C含量正相关于结构稳定性, 但过长的茎部易引发动力学陷阱, 阻碍构象的快速转换.
冷休克蛋白(cold shock protein, Csp)由65—70个氨基酸组成, 可以与任意序列的ssDNA结合[9], 但对胸腺嘧啶[10]和Y-box核心序列ATTGG[11]具有较强的结合能力. 来自常温枯草芽孢杆菌(bacillus subtilis)、嗜热芽孢杆菌(bacillus caldolyticus)和超嗜热海栖热袍菌(thermotoga maritima)的3种同源冷休克蛋白BsCsp, BcCsp 和 TmCsp不含有二硫键且结构简单, 是研究蛋白质与DNA相互作用的理想模型.
Csp虽被证实通过结合ssDNA发挥转录抗终止功能[12,13], 但其分子机制尚未完全解析. 通过冷休克反应中蛋白质表达量的变化, Csp的具体作用可以理解为: 当外界环境温度剧烈下降时, 细菌体内Csp基因转录上调[14], Csp表达量显著增加[15–17]. 而由于非冷休克基因序列折叠成发夹等二级结构, 导致非冷休克蛋白表达量急剧下降. 作为核酸伴侣, Csp能够与非冷休克基因启动子区域的ssDNA和mRNA结合[18], 使其二级结构被破坏, 帮助转录翻译过程正常进行[19,20], 进而恢复蛋白正常表达[21].
光镊[22,23]、原子力显微镜[24]和磁镊[25]在内的单分子技术[26]通过对单个核酸链或蛋白质链施加拉力, 可以探测到核酸或蛋白质分子的构象转变动力学特征. 早期的研究利用磁镊测量了在不同浓度的ssDNA存在时, Csp力依赖的折叠和去折叠速率. 发现Csp表现出不同的力敏感的去折叠速 率[27,28]以及ssDNA不仅与天然状态的BcCsp结合, 还能够与部分去折叠态的构象结合[29].
由于磁镊具有在小力下长时间稳定测量的优点[30], 先前的研究通过磁镊, 在恒定力下已经测量到了DNA发夹折叠和去折叠来回转变过程[31]. 本工作为了研究具有不同热稳定性Csp同源蛋白的功能保守性, 利用磁镊拉伸DNA发夹, 分别测量了在不同BsCsp, BcCsp和TmCsp蛋白浓度下DNA发夹力依赖的折叠和去折叠速率. 通过贝尔模型拟合, 得到了DNA发夹临界力的变化, 从而探究Csp对DNA发夹稳定性的影响. 研究发现DNA发夹力依赖的折叠速率随着Csp浓度的增大而降低, 而当Csp达到一定浓度后, 发夹的去折叠速率显著增大. 通过对DNA发夹两端施加跳变力, 直接证实了Csp主要与ssDNA而不与dsDNA结合.
-
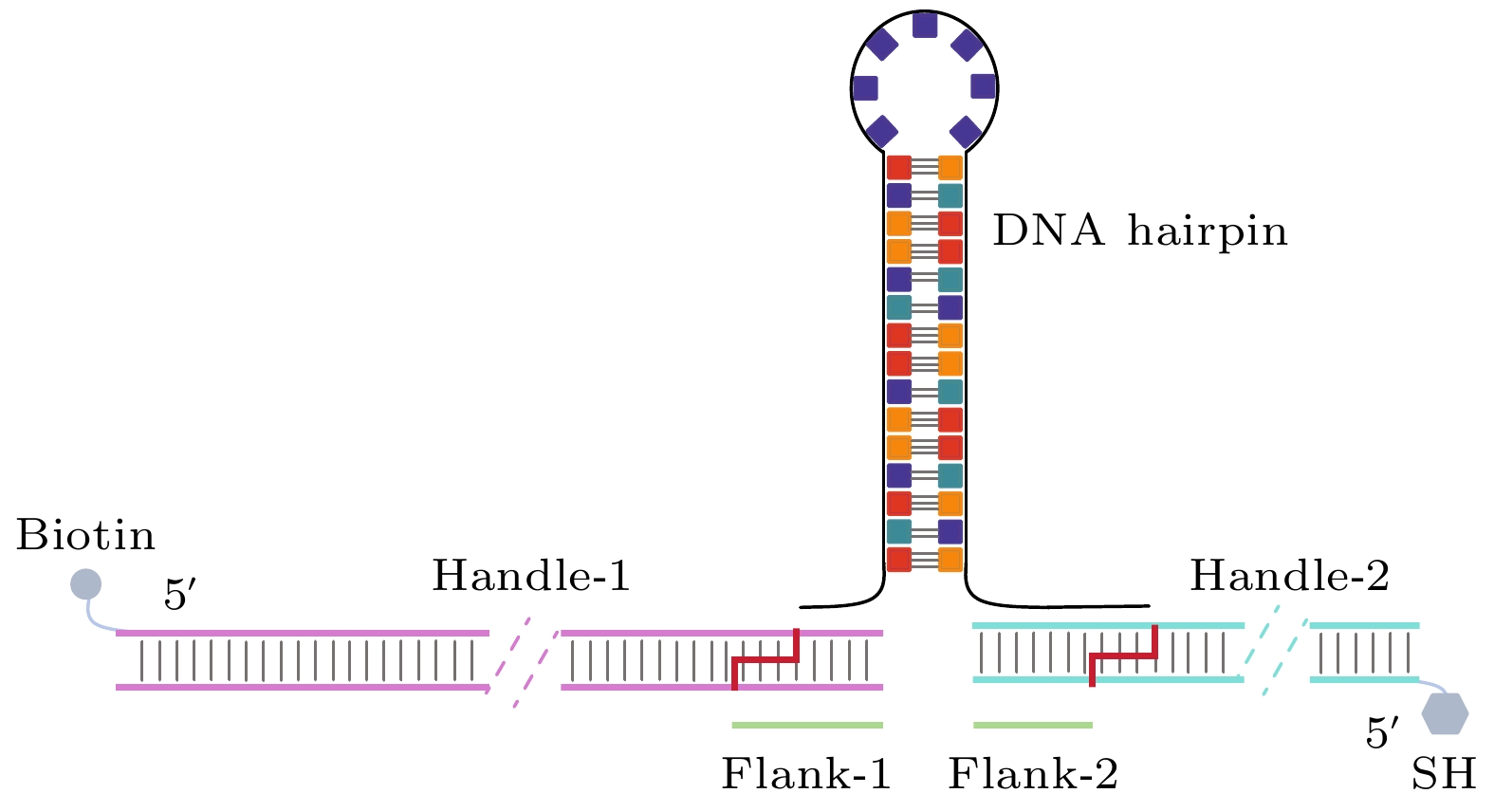
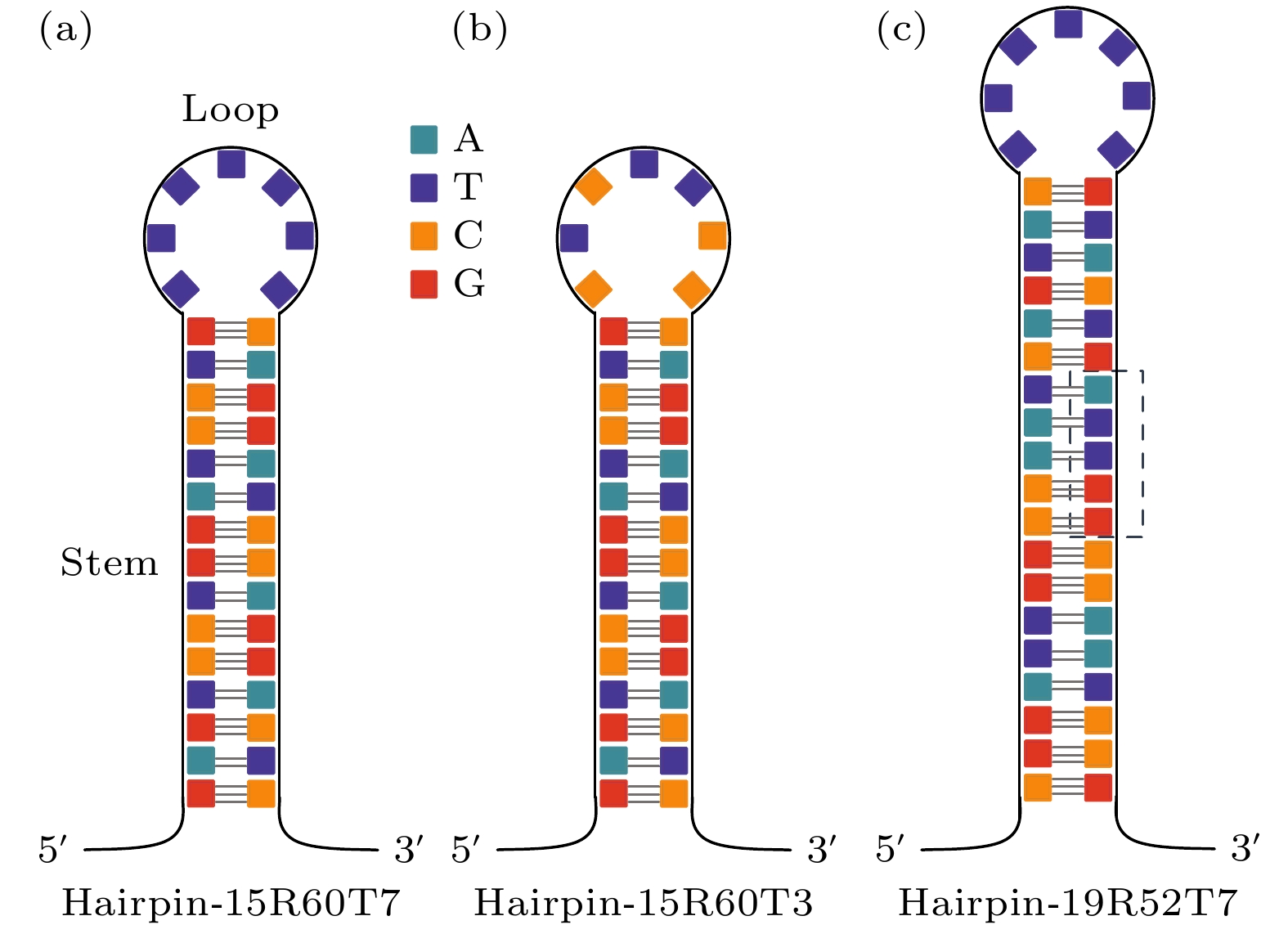
我们设计了3组DNA发夹结构用于磁镊实验. 其中, 发夹-15R60T7 (图1(a))和发夹-15R60T3(图1(b))均含15-bp随机序列茎部(GC含量60%), 环部分别包含7个和3个胸腺嘧啶; 发夹-19R52T7(图1(c))插入ATTGG特征序列构建19-bp茎部(GC含量52%), 环部含7个胸腺嘧啶.
为减少发夹两端与磁球、载玻片表面的非特 异性吸附, 在发夹结构两端引入双链DNA手柄(handle): 加入λ-DNA、dNTPs、高保真DNA聚合酶和引物, 通过聚合酶链式反应扩增制备DNA手柄, 其中Handle-1(372 bp)使用正向引物5'-CCACGACACTAGCTGCTGCATTACCATCTGCCTGGTATCAGGGCAAC-3'和生物素标记的反向引物5'-Biotin-GCTCCGCCGACACGTTATGG-3' 进行制备, 而Handle-2(240 bp)使用正向引物5'-thiol-AAGCGGGGATTTCCGTCGGG-3' 和反向引物5'-GGAGTCTGTGCACAAGTAATACCGCCAGCGAGGTGGCC-3' 在含4 mmol/L DTT的体系中扩增以防止二硫键形成. 随后将扩增出来的DNA手柄分别经过琼脂糖凝胶电泳(95 V, 35 min)验证和试剂盒纯化.
为了让手柄与发夹进行连接, 使用BstXI限制性内切酶处理DNA手柄纯化产物(37 ℃, 2 h)生成黏性末端. 将合成的DNA发夹序列放入95 ℃水浴锅自然冷却至室温完成退火处理, 以保证ssDNA形成发夹结构. 将带有黏性末端的DNA手柄、DNA发夹和ssDNA侧链(flank)等摩尔浓度与T4多核苷酸激酶、T4 DNA连接酶混合, 于37 ℃水浴孵育30 min. 随后转移至16 ℃进行过夜连接反应, 图2展示了最终合成的磁镊拉伸DNA发夹构建物.
-
蛋白构建体HisTag-Csp的构建是将公司合成的3种Csp基因序列分别插入到载体pQE-60实现的. 取2 μL Csp-pQE-60重组质粒通过热激法转化至50 μL BL21表达感受态细胞, 涂布于含有氨苄青霉素的LB固体培养基, 37 ℃过夜培养后挑取单菌落接种至300 mL氨苄抗性的液体LB培养基. 在37 ℃, 200 r/min振荡培养至光密度达0.6(10—12 h), 加入异丙基-β-D-硫代吡喃半乳糖(IPTG)至终浓度0.25 mmol/L, 调整培养条件至25 ℃, 200 r/min继续诱导表达8—10 h. 随后通过4 ℃, 5000 r/min离心收集菌体, 将菌体重悬于裂解缓冲液(50 mmol/L Tris、500 mmol/L氯化钠、10%甘油、20 mmol/L咪唑, pH = 8.0). 经超声破碎后, 目标蛋白使用Ni-NTA Sefinose Resin (生工)和Superdex 200 (通用医疗)进行纯化, 纯化后的蛋白经液氮速冻后保存于–80 ℃超低温冰箱.
-
首先将22×32 mm载玻片与20×20 mm盖玻片依次置于含5% 洗涤剂、去离子水的染色槽中超声清洗, 随后载玻片经等离子清洗后, 浸入1% 3-氨基丙基三乙氧基硅烷(APTES, Sigma)的甲醇溶液. 室温硅烷化50 min后, 用甲醇、去离子水冲洗并放置烘箱烘干. 用细条状的封口膜将载玻片与盖玻片黏合, 并用玻璃胶在两端制作蓄水池. 室温下晾至1 h后, 注入经1×PBS缓冲液稀释的3.0 μm聚苯乙烯微球. 接着冲入1% Sulfo-SMCC并静置20—25 min. 将连接好的DNA发夹构建物经1×PBS缓冲液稀释后加入到流动室中, 静置30 min后, 注入600 μL 1×PBS缓冲液冲走未连接的DNA链, 最后加入1% BSA的PBS缓冲液, 于4 ℃冰箱过夜孵育以屏蔽玻璃表面的非特异性相互作用. 随后, 将链霉亲和素包被的磁球注入流动室, 并孵育5 min, 于是通过生物素-链霉亲和素特异性连接的单分子体系构建成功.
-
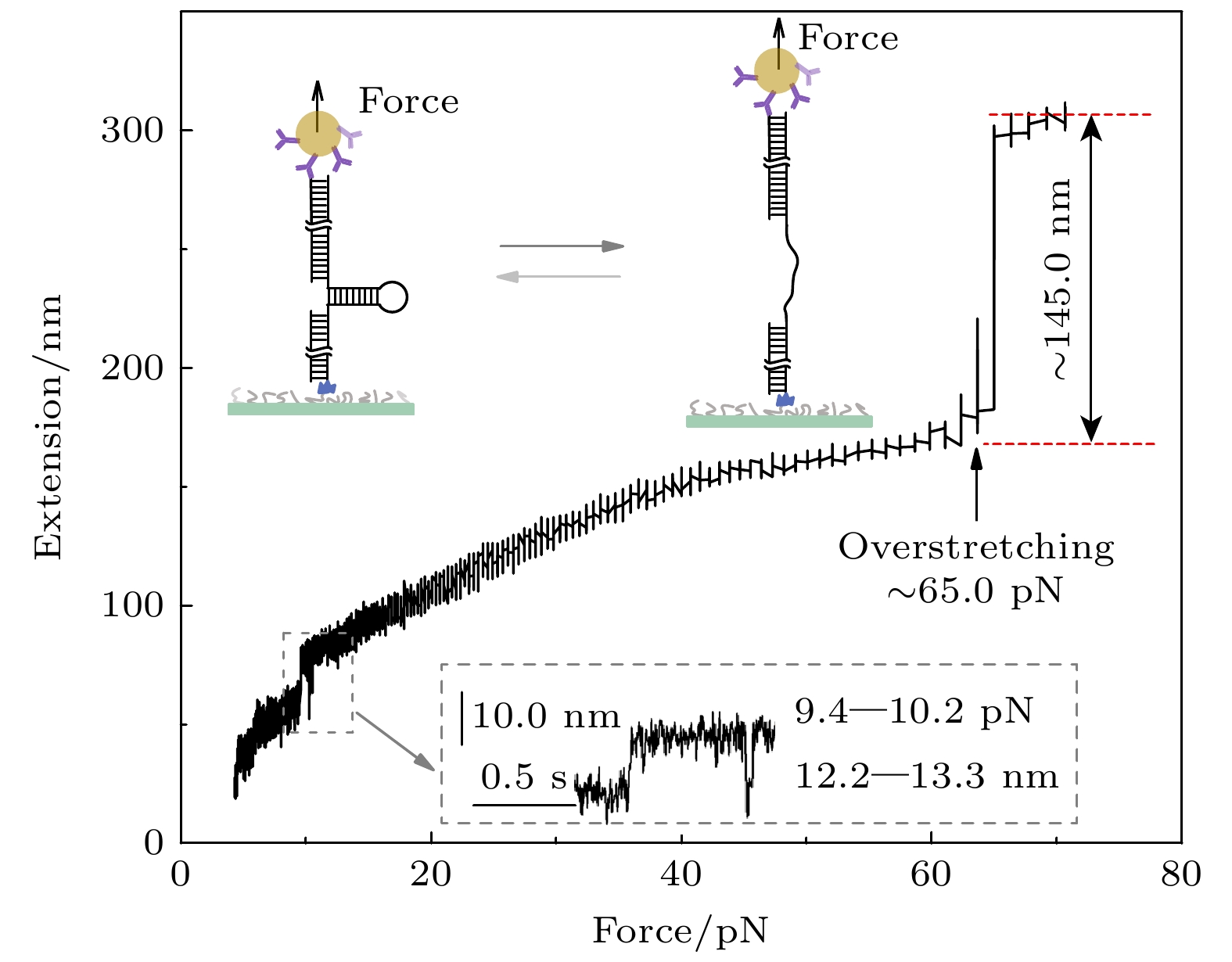
为了确定DNA发夹(15R60T7)的特征性力学响应轨迹, 首先在没有Csp蛋白的缓冲液中对DNA系绳施加3.0—70.0 pN左右的拉力(图3). 当拉力处于9.4 pN和10.2 pN时, 分别出现步长为12.2 nm和13.3 nm的去折叠信号. 在9.4—10.2 pN范围内, DNA发夹发生去折叠和折叠转换, 对应发夹结构中15个bp的双链-单链构象变换. 当拉力加载到65.0 pN左右时, 双链DNA手柄发生B型到S型的构象转变, 其延伸长度骤增70%(612 bp×0.34 nm/bp×70% ≈ 145 nm), 该力学响应特性与理论预测一致, 可以确认所拉到的正是目标DNA分子.
-
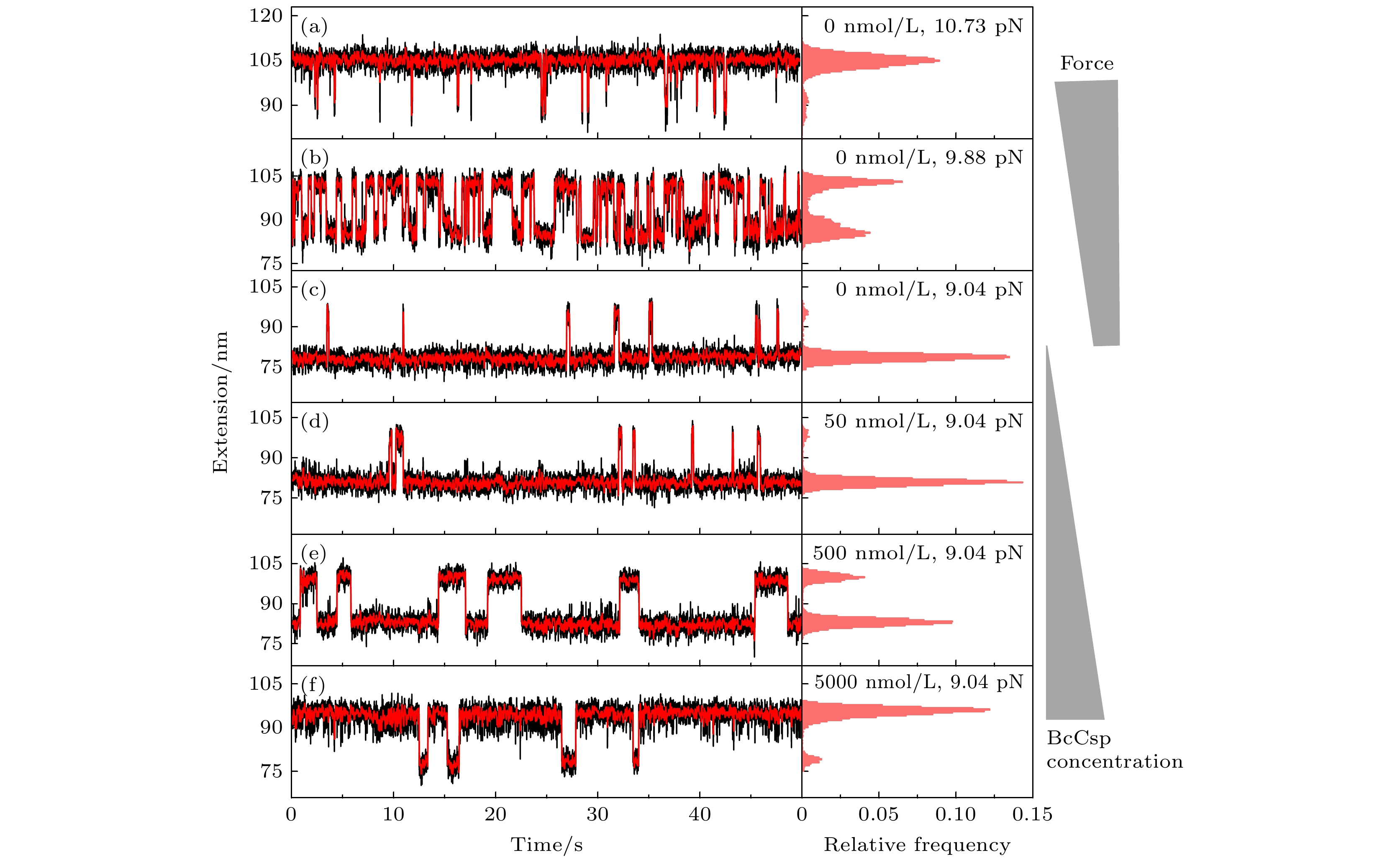
从加载速率拉伸实验中, DNA发夹结构表现出往复的折叠和去折叠动态转换特征. 基于磁镊技术可以长时间恒力测量的优点, 系统探究了Csp蛋白对DNA发夹平衡态动力学的影响. 实验首先在无BcCsp条件下进行恒力平衡态测试, 随后通过缓冲液置换依次注入含50 nmol/L, 500 nmol/L和5000 nmol/L BcCsp的测量体系(每次置换体积200 μL). 图4显示, 经平滑处理后DNA发夹延伸的分布直方图呈现双峰分布, 对应DNA发夹的折叠态与去折叠态.
在无配体条件下, DNA发夹在力增过程中逐渐向去折叠态偏移. 当BcCsp浓度为50 nmol/L时, 平衡态转变没有出现显著变化, 表明该浓度下未形成有效蛋白-核酸复合物. 值得注意的是, 当BcCsp浓度提高至500 nmol/L和5000 nmol/L时, DNA发夹在相同外力作用下表现出明显的去折叠态偏好性, 推测其机制源于BcCsp对DNA发夹去折叠态构象的特异性结合作用. 该现象证实Csp蛋白的结合能显著降低DNA发夹的热力学稳定性.
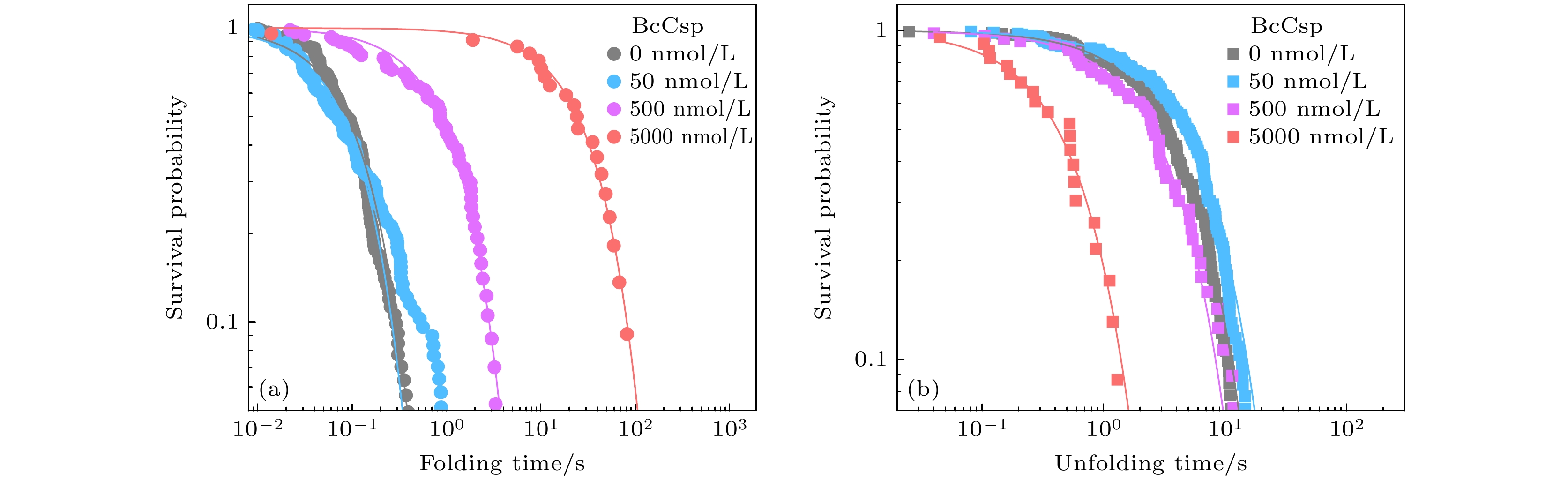
基于隐马尔可夫模型构建的构象态识别框架, 对发夹-15R60T7的时间伸长曲线进行折叠和去折叠态构象的自动识别, 得到了折叠和去折叠状态寿命的累积分布. 采用指数函数拟合了不同BcCsp浓度(0—5000 nmol/L)下该发夹结构去折叠和折叠状态的存活概率(图5), 得到其折叠和去折叠速率. 在9.04 pN恒力条件下, 浓度梯度实验显示: 发夹-15R60T7力依赖的折叠速率随着BcCsp浓度增大而逐渐减小, 当BcCsp浓度达到500 nmol/L时, 折叠速率显著降低, 降幅达两个数量级. 而去折叠速率的显著提升仅发生在BcCsp浓度为5000 nmol/L 时, 这可能是由于发夹初始解链时新暴露的ssDNA区域, 为高浓度的Csp蛋白提供了快速结合位点, 通过结合暴露的ssDNA降低了去折叠能垒.
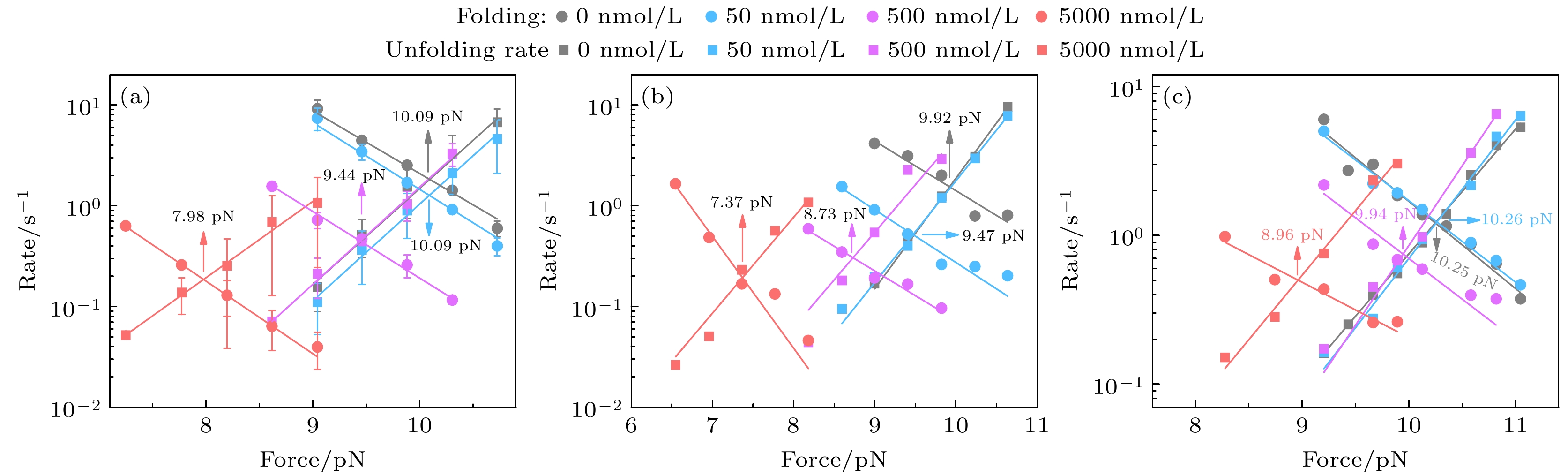
DNA发夹力依赖的折叠速率kf和去折叠速率ku的对数值与力呈线性关系. 在测量缓冲液中分别含有 0 nmol/L (9.04—10.73 pN), 50 nmol/L (9.04—10.73 pN), 500 nmol/L (8.62—10.31 pN)和5000 nmol/L (7.25—9.04 pN) BcCsp时, 力依赖的折叠和去折叠速率可以通过贝尔模型拟合:
$ {k}_{{\mathrm{u}}}\left(f\right)={k}_{{\mathrm{u}}}^{0}{\mathrm{ }}{\mathrm{e}}{\mathrm{x}}{\mathrm{p}}\left(\dfrac{f{x}_{{\mathrm{u}}}{\mathrm{ }}}{{k}_{{\mathrm{B}}}T}\right) $ 和$ {k}_{{\mathrm{f}}}\left(f\right)={k}_{{\mathrm{f}}}^{0}{\mathrm{ }}{\mathrm{e}}{\mathrm{x}}{\mathrm{p}}\left(-\dfrac{f{x}_{{\mathrm{f}}}{\mathrm{ }}}{{k}_{{\mathrm{B}}}T}\right) $ , 其中$ f $ 为DNA发夹所受的拉力; kf和ku为折叠和去折叠速率;$ {{k}_{{\mathrm{f}}}^{0}{\mathrm{和}}k}_{{\mathrm{u}}}^{0} $ 为外推到拉力为零时的折叠和去折叠速率;$ {x}_{{\mathrm{f}}}{\mathrm{和}}{x}_{{\mathrm{u}}} $ 为折叠和去折叠距离, 分别为去折叠态、自然状态与过渡态在拉力方向上伸长的差;$ {k}_{{\mathrm{B}}} $ 为玻尔兹曼因子; T为绝对温度. 折叠速率和去折叠速率拟合线段交点对应的力即为DNA发夹的临界力$ {f}_{{\mathrm{c}}} $ . 实验测得DNA发夹-15R60T7在BcCsp浓度为0 nmol/L, 50 nmol/L, 500 nmol/L和5000 nmol/L时的临界力分别为10.09 pN, 10.09 pN, 9.44 pN和7.98 pN(图6(a)), 表明BcCsp浓度的增大显著降低DNA发夹的机械稳定性, 表现为$ {f}_{{\mathrm{c}}} $ 值随蛋白浓度升高而下降. 当BcCsp浓度从0 nmol/L增至5000 nmol/L时, 在9.04 pN下, DNA发夹-15R60T7的平衡常数$ {k}_{{\mathrm{e}}{\mathrm{q}}} = {k}_{{\mathrm{f}}} / {k}_{{\mathrm{u}}} $ 从58减小到0.04, 降幅超过3个数量级, 这也说明了BcCsp的结合促使平衡向去折叠态偏移.按照相同的方法, 研究了BcCsp对DNA发夹-15R60T3平衡态动力学的影响. 图6(b)展示了发夹-15R60T3在溶液中含有0—5000 nmol/L BcCsp时力依赖的折叠和去折叠速率. 随着BcCsp浓度的增大, 发夹-15R60T3的折叠速率逐渐减小, 去折叠速率在BcCsp浓度大于500 nmol/L时明显增大. DNA 发夹发生平衡态转变的力区间和临界力逐渐向小力方向移动.
为了探究BsCsp蛋白对DNA发夹结构有何影响, 研究了BsCsp对DNA发夹-15R60T3平衡态动力学的影响. 图6(c)展示了发夹-15R60T3在溶液中含有0—3000 nmol/L BsCsp时力依赖的折叠和去折叠速率. 当BsCsp浓度达到300 nmol/L以上时, DNA 发夹折叠速率明显下降. 而只有当BsCsp浓度在3000 nmol/L时, 去折叠速率才显著增大. DNA发夹-15R60T3在0 nmol/L, 30 nmol/L, 300 nmol/L 和3000 nmol/L BsCsp 存在时DNA发夹的临界力分别是 10.25 pN, 10.26 pN, 9.94 pN和8.96 pN. 随着BsCsp浓度的增大, 发夹的临界力也逐渐减小. 在力等于9.20 pN时, DNA发夹-15R60T3的平衡常数
$ {K}_{{\mathrm{e}}{\mathrm{q}}} $ 从0 nmol/L BsCsp时的37减小到3000 nmol/L BsCsp时0.57, 减小了2个数量级左右, 这也说明了BsCsp的结合能够降低 DNA发夹的稳定性.研究揭示, BcCsp和BsCsp通过结合单链DNA, 不仅显著抑制其折叠动力学, 还在高浓度时触发去折叠速率增大. 当浓度超过临界阈值时, Csp分子通过亚毫秒级快速结合动力学占据新生ssDNA区域, 形成空间位阻效应, 并诱导茎部DNA双螺旋发生去折叠转变.
-
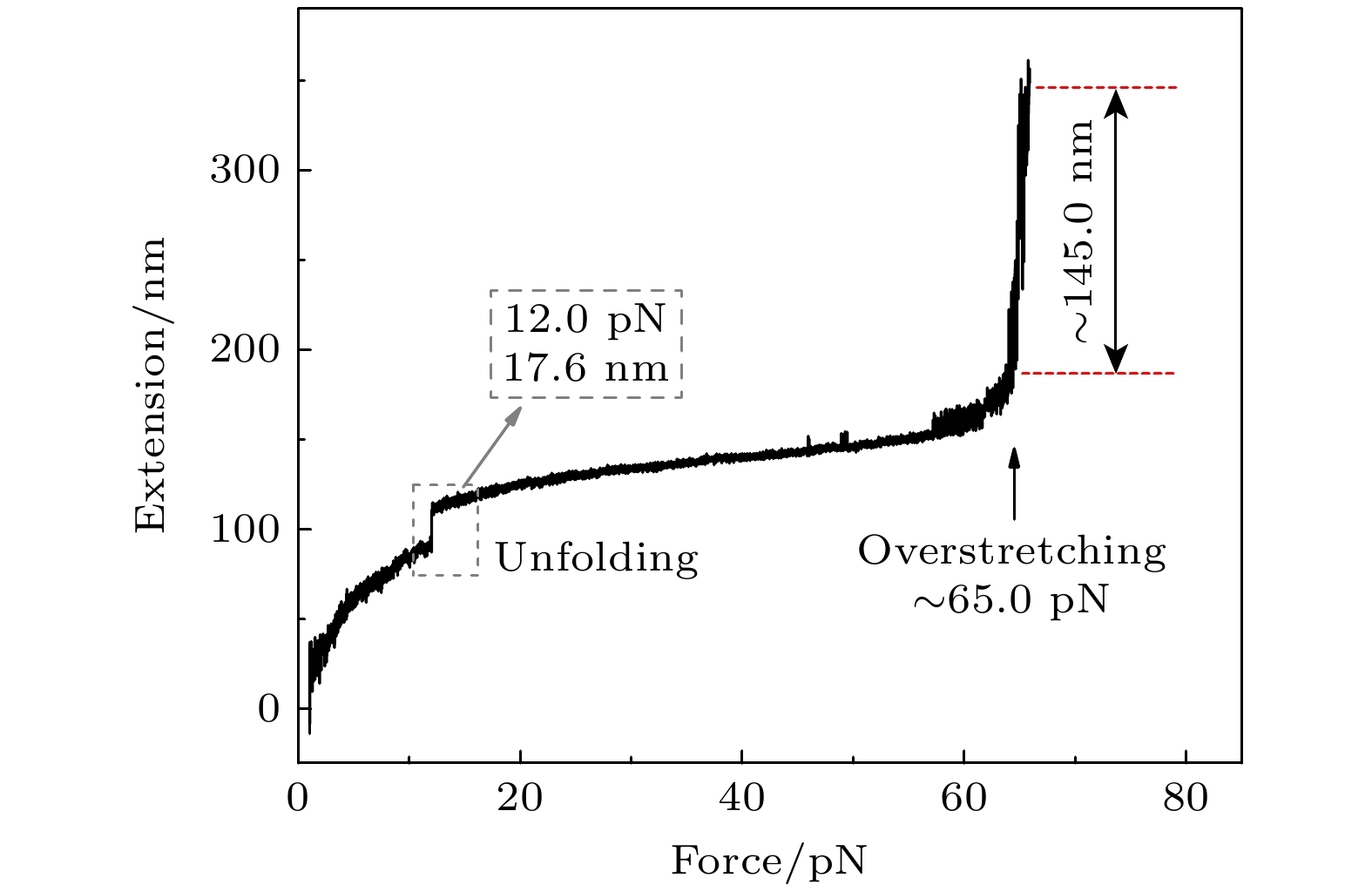
为明确Csp蛋白对单链/双链DNA的选择 性结合特性, 基于Csp对ATTGG序列的高亲 和特性[11], 设计了茎部含有该基序的DNA发夹-19R52T7 (图1(c)), 力从1.0 pN加载到67.0 pN, 发夹在12.0 pN以步长为17.6 nm发生去折叠, 并在65.0 pN处观察到双链DNA手柄的过渡拉伸信号(图7), 确定拉到了正确的DNA系绳.
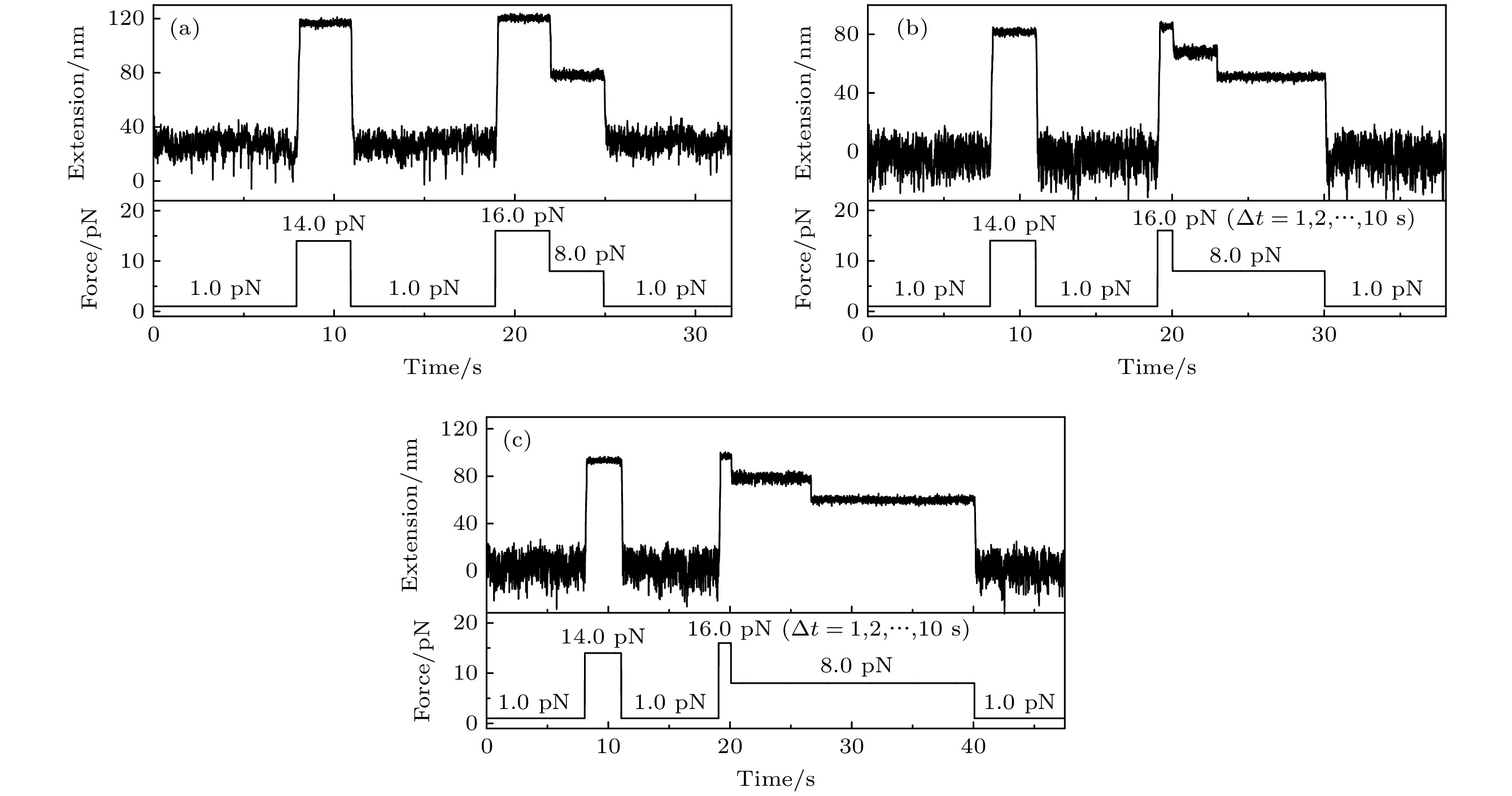
首先对DNA发夹-19R52T7进行力从小到大的力跳变实验, 来验证Csp是否与dsDNA结合. 首先将拉力在1.0 pN停留8 s, 保证DNA发夹处于折叠状态. 随后将力增至14.0 pN并停留3 s, 使DNA发夹发生去折叠. 由于在该力下DNA发夹去折叠速率快于磁镊5 ms时间分辨率, 以至于在0 nmol/L TmCsp的缓冲液中, 没有观察到DNA发夹在去折叠过程中伸长出现延迟跳变的现象(图8(a)). 当TmCsp浓度增大到100 nmol/L和500 nmol/L时, 也没有观察到延迟现象(图8(b), (c)), 表明TmCsp很少与dsDNA结合.
我们又进行力从大到小的力跳变实验, 验证Csp是否与ssDNA结合. 对刚才的DNA发夹施加16.0 pN的拉力并停留3 s, 确保DNA发夹处于去折叠态. 随后将力减小到8.0 pN, DNA发夹回到折叠态. 在0 nmol/L TmCsp测量缓冲液中, DNA发夹的伸长没有出现延迟(图8(a)). 而当TmCsp的浓度增大到100 nmol/L和500 nmol/L 时, DNA发夹在折叠过程中延伸分别出现3 s和7 s的延迟(图8(b), (c)), 说明TmCsp能够与ssDNA结合.
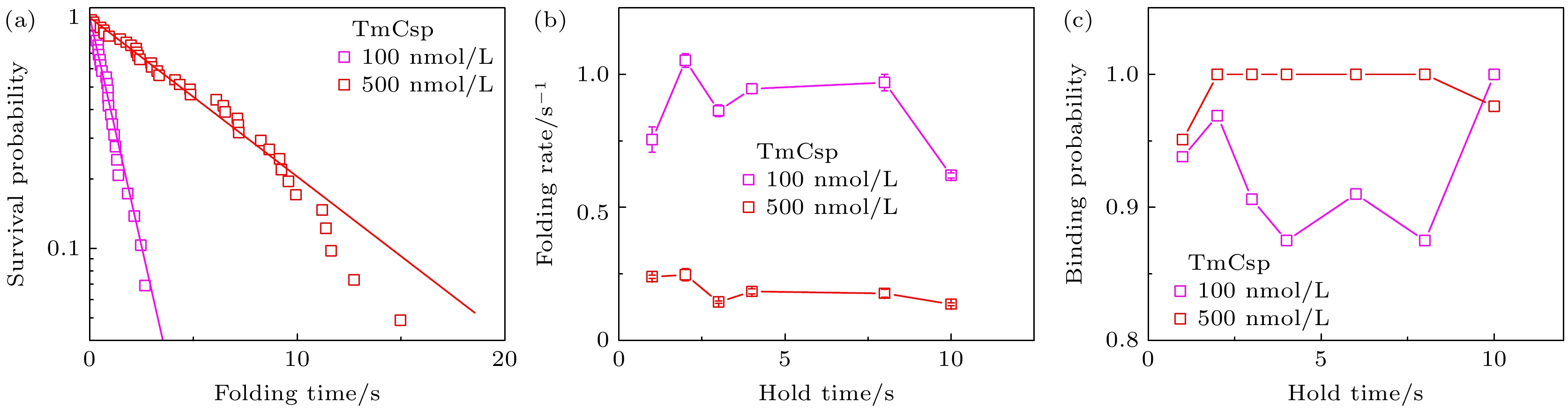
TmCsp与ssDNA的结合快慢直接影响生物体应对冷休克等刺激的生理恢复速率. 我们试图通过调控16.0 pN力的维持时间(即DNA发夹-19R52T7处于ssDNA状态的时长)来得到TmCsp与DNA发夹-ATTTGG的结合动力学特性. 在力从16.0 pN跃迁至8.0 pN的实验中, 测量了DNA发夹完全折叠所需时间. 通过指数拟合去折叠状态的生存概率, 得到DNA发夹的折叠速率(图9(a)). 当TmCsp浓度从100 nmol/L增至500 nmol/L时, DNA 发夹在8.0 pN下的折叠速率显著降低(图9(b)), 但在16.0 pN去折叠状态的停留时间对折叠速率无显著影响. 这说明TmCsp与ssDNA结合可以快速达到平衡状态.
通过统计在力减小的跳变实验中DNA 发夹伸长出现延迟的概率, 来判定TmCsp是否与ssDNA结合. 图9(c)显示, DNA 发夹结合TmCsp的概率与16.0 pN停留时间(1 s及以上)无关, 这一现象也证实了TmCsp与ssDNA是一个快速结合的过程.
-
本研究使用磁镊系统研究BcCsp, BsCsp对DNA发夹-15R60T7和发夹-15R60T3中双螺旋结构稳定性的调控机制及TmCsp与发夹-19R52T7中单双链DNA的结合特性. 测量了BcCsp和BsCsp存在和不存在时, DNA发夹力依赖的折叠和去折叠速率以及研究了DNA发夹处于去折叠态时间长短对其折叠速率和结合TmCsp概率的影响.
首先, 通过测量不同浓度BcCsp和BsCsp条件下DNA发夹-15R60T7和发夹-15R60T3的折叠和去折叠动力学, 发现随着Csp浓度增大, DNA发夹的折叠速率逐渐减小. 这主要是由于Csp结合到去折叠态的发夹ssDNA上, 进一步稳定了DNA发夹的去折叠态, 减缓了发夹的折叠转变. 只有当茎部上的Csp完全解离时, ssDNA才能形成发夹结构. 一个Csp结合位点大概是6—7个核苷酸[10,32,33], 上述两种DNA发夹茎部含有30个核苷酸, 能够结合4—5个Csp. 因此在相同力下, Csp浓度越高, 结合到DNA发夹ssDNA区域上的概率越大, 更难形成发夹结构. 由于Csp几乎不与处于dsDNA态的发夹结合, 当Csp浓度比较低时, Csp不会对DNA发夹的去折叠转变产生影响. 而当Csp浓度增大到阈值时, DNA发夹的去折叠速率显著增大. 这可能是DNA发夹刚被打开, Csp快速结合到发夹新暴露的ssDNA区域, 并阻止其重新折叠, 从而促进发夹加速打开.
实验观察到, 随着BcCsp浓度由0 nmol/L增至5000 nmol/L, DNA发夹-15R60T7的临界力从10.09 pN显著降低至7.98 pN(降幅21%), 而发夹-15R60T3的临界力则由9.92 pN降至7.37 pN(降幅26%). 类似地, 当BsCsp浓度从0 nmol/L升高到3000 nmol/L时, 发夹-15R60T3的临界力从10.26 pN下降至8.96 pN(降幅13%). 这些数据清楚地表明, Csp能够显著降低DNA双螺旋结构的稳定性. 这种结构调控作用在分子水平上解释了Csp如何促进核酸二级结构解链以保障转录等关键生命活动的正常进行.
接着我们设计了DNA发夹的力增大或力减小跳变实验, 通过观察延伸是否出现延迟, 直接观察到Csp与ssDNA而不与dsDNA结合, 且Csp结合ssDNA速率极快, 以至于在实验可调节的等待时间范围内, 几乎看不到DNA发夹折叠速率和结合概率的明显变化. 这种高效的结合特性使其能够及时阻止核酸局部配对结构的形成, 对维持细胞在低温下行使正常功能具有重要作用.
本研究不仅阐明了Csp对DNA结构稳定性的调控规律, 还揭示了其快速结合ssDNA的分子机制. 这些发现为深入理解冷休克蛋白在低温胁迫响应中的功能提供了新的实验证据和理论依据. 特别值得关注的是细胞中Csp的浓度可达几十个 μmol/L[21], Csp通过降低DNA二级结构的稳定性和快速结合ssDNA的双重作用, 形成了一套高效的调控机制, 这可能是生物体适应环境变化的重要策略之一.
冷休克蛋白对DNA发夹稳定性影响及结合特性的单分子磁镊研究
Effect of cold shock protein on DNA hairpin stability and binding characteristics by single molecule magnetic tweezers
-
摘要: 冷休克蛋白是一类高度保守的核酸结合蛋白, 由65—70个氨基酸组成的5条反向平行β链, 形成结构紧凑的β桶状结构. 冷休克蛋白在细菌应对冷刺激过程中起重要作用, 但其具体工作机制尚未完全阐明. 本研究利用磁镊技术系统研究了不同浓度冷休克蛋白对DNA发夹结构折叠和去折叠动力学的影响, 定量测定了相应条件下DNA 发夹的折叠和去折叠速率. 实验结果表明, 在一定浓度范围内, 随着冷休克蛋白浓度增大, DNA发夹的折叠速率显著降低; 而去折叠速率保持不变. 当冷休克蛋白达到一定浓度阈值时, 去折叠速率也呈现明显上升趋势. 进一步研究发现, 冷休克蛋白浓度增大使DNA 发夹的临界力减小, 从而降低了发夹的结构稳定性. 通过力跳变实验, 更直观地表现出冷休克蛋白只与单链DNA结合, 而不与双链DNA相互作用. 这些单分子水平的研究结果揭示了冷休克蛋白通过调控核酸双螺旋结构稳定性来维持细菌低温适应性的分子机制.Abstract: Cold shock proteins (Csps) are a class of highly conserved nucleic acid-binding protein composed of 65−70 amino acids that form a compact β-barrel structure with five antiparallel β-strands. As nucleic acid-binding proteins, Csps play an important role in bacterial response to cold shock, yet their precise working mechanism is still unclear. As is well known, DNA hairpin undergoes folding-unfolding transitions under small constant forces. Magnetic tweezers technique has obvious advantages in this kind of research, especially its capacity for extended-duration constant-force measurements at pico-Newton force level, which makes it very suitable for characterizing the conformational transition dynamics of DNA hairpin at low forces of several pico-Newton. In this study, we first stretch DNA hairpin from its N- and C-termini by using magnetic tweezers. Then, we sequentially introduce Csp buffer solutions with increasing concentrations into the flow chamber and measure the folding and unfolding rates of the DNA hairpin at different Csp concentrations. It is found that within a certain concentration range, increasing Csp concentration can significantly reduce the DNA hairpin folding rate while keeping the unfolding rate almost unchanged. This behavior occurs because Csp only binds to single-stranded DNA (ssDNA), and interacts with the ssDNA region of the unfolded DNA hairpin, thereby hindering the folding process. As Csp does not interact with double-stranded DNA (dsDNA), the above-mentioned effect on the unfolding process is negligible. Furthermore, the critical force of DNA hairpin progressively decreases with the increase of Csp concentration, demonstrating that Csp effectively destabilizes the hairpin structure. When the Csp concentration reaches sufficiently high levels, the DNA hairpin’s unfolding rate increases considerably. This phenomenon may be caused by the rapid binding of Csp to newly exposed ssDNA regions of partially unfolded DNA hairpins, which prevents refolding and accelerates the unfolding pathway. In force-jump experiments using Csp-containing buffers, the binding preference of Csp for either ssDNA or dsDNA can be directly determined by analyzing whether the delayed response of DNA hairpin extension occurs. In force-increasing jump experiments, no extension delay is observed in the DNA hairpin unfolding process. In contrast, force-decreasing jump experiments shows significant extension delay in the folding process. These single-molecule measurements provide direct evidence that Csp only specifically binds to ssDNA, further demonstrating that its binding kinetics occur very rapidly. This study delves into the molecular mechanisms by which Csps maintain normal cellular functions in cold chock conditions.
-
Key words:
- cold shock protein /
- DNA hairpin /
- magnetic tweezers /
- single-stranded DNA .
-
-
图 1 三种DNA发夹结构 (a), (b)茎部为随机序列的发夹-15R60T7 (a)和发夹-15R60T3 (b); (c)茎部含特异性ATTGG基序(虚线框)的发夹-19R52T7
Figure 1. Three DNA Hairpin structures: (a), (b) Hairpin-15R60T7 (a) and Hairpin 15R60T3 (b) with random stem sequences; (c) Hairpin -19R52T7 containing the specific ATTGG motif in the stem (marked by a dashed box).
图 2 DNA发夹构建物示意图. 限制性内切酶特异性切割DNA手柄(红线)后, 通过T4 DNA连接酶与发夹、侧链部分连接. 生物素标记的Handle-1的5’端与磁球表面的链霉亲和素相连, 巯基修饰的Handle-2的5’端与载玻片表面Sulfo-SMCC相连. 斜向虚线表示省略的碱基对, 未按标准比例绘制
Figure 2. Schematic of DNA Hairpin construct. The DNA handles are specifically cleaved by restriction endonuclease (red lines), followed by T4 DNA ligase-mediated junction with the Hairpin and flank segments. The 5’ end of biotin-labeled Handle-1 is conjugated to streptavidin on the surface of magnetic beads, while the 5’-thiol-modified end of Handle-2 is conjugated with Sulfo-SMCC-coated glass substrates. Dashed diagonal lines represent omitted base pairs. Structural dimensions are not proportionally scaled.
图 3 DNA发夹-15R60T7代表性的力-延伸示意图. 当拉力增大时, DNA发夹在大约9.4 pN处以12.2 nm的步长去折叠, 紧接着又折叠回去; 在约10.2 pN处发生第二次去折叠, 步长为13.3 nm, 插图描述了DNA 发夹在力作用下发生折叠-去折叠转变
Figure 3. Representative force-extension curve of DNA Hairpin-15R60T7. As the force increases, the DNA Hairpin unfolds at approximately 9.4 pN with a step size of 12.2 nm, followed by a refolding event. A second unfolding event occurs at around 10.2 pN with a step size of 13.3 nm. The inset illustrates the force-induced folding and unfolding transitions of the DNA Hairpin.
图 4 BcCsp浓度梯度下DNA发夹平衡态动力学表征 (a)—(c) DNA发夹-15R60T7在无BcCsp体系中的时间-延伸曲线, 分别对应10.73 pN, 9.88 pN和9.04 pN的恒力测量条件; (d)—(f)含梯度浓度BcCsp (50, 500和5000 nmol/L)时, 9.04 pN力场下DNA发夹的动力学响应; 右侧面板为对应平滑延伸的相对频率分布, 均呈现双峰分布特征(黑色原始数据采样率200 Hz, 红色曲线经0.1 s 时间窗口平滑处理)
Figure 4. Equilibrium measurement of DNA Hairpin in solutions with different concentration of BcCsp: (a)–(c) Extension time course of DNA Hairpin-15R60T7 in the absence of BcCsp under constant force measurements at 10.73 pN, 9.88 pN, and 9.04 pN; (d)–(f) dynamic responses of the DNA Hairpin at 9.04 pN in solutions with BcCsp concentrations of 50, 500, and 5000 nmol/L. The right panels show relative frequency of the smoothed extensions, exhibiting two peaks. The raw data (black) is recorded at 200 Hz and smoothed over a 0.1 s time window (red).
图 5 在0—5000 nmol/L BcCsp范围内, DNA发夹-15R60T7在 9.04 pN下去折叠态(a)和折叠态(b)的存活概率, 其中实线表示指数拟合以确定kf和ku
Figure 5. Survival probability of folded (a) and unfolded states (b) of DNA Hairpin-15R60T7 at 9.04 pN in solutions with 0–5000 nmol/L BcCsp. The solid curves represent the exponential fitting to determine kf and ku.
图 6 DNA发夹在Csp测量缓冲液中力依赖的折叠和去折叠速率 (a), (b) DNA发夹-15R60T7 (a)和发夹-15R60T3 (b)分别在0—5000 nmol/L BcCsp测量缓冲液中力依赖的折叠和去折叠速率; (c) DNA发夹-15R60T3在0—3000 nmol/L BsCsp测量缓冲液中力依赖的折叠和去折叠速率; 箭头表示的交叉点给出了DNA发夹在不同浓度Csp下的临界力
Figure 6. Force-dependent folding and unfolding rates of DNA Hairpin in solutions with different concentration of Csp: (a), (b) Force-dependent folding and unfolding rates of DNA Hairpin-15R60T7 (a) and Hairpin-15R60T3 (b) in solutions with 0–5000 nmol/L BcCsp; (c) force-dependent folding and unfolding rates of DNA Hairpin-15R60T3 in solutions with 0–3000 nmol/L BsCsp. The intersection points indicated by the arrows give the critical forces of the DNA Hairpin at different concentrations of Csp.
图 7 DNA发夹-19R52T7典型的力-延伸示意图. DNA发夹在大约12.0 pN处以17.6 nm的步长发生去折叠, 当力加载到65 pN时, DNA双链手柄发生过渡拉伸转变
Figure 7. Representative force-extension curve of DNA Hairpin-19R52T7. The DNA Hairpin unfolds at approximately 12.0 pN with a step size of 17.6 nm, and the DNA double-stranded handles overstretch when force is 65 pN.
图 8 力跳跃实验研究TmCsp与单双链DNA结合动力学. (a) 0 nmol/L, (b) 100 nmol/L和(c) 500 nmol/L TmCsp 存在时, DNA发夹-19R52T7 去折叠和折叠的典型力跳变测量; (a)—(c)左侧的第1个力跳变验证TmCsp是否与dsDNA结合, 左侧的第2个力跳变验证TmCsp是否与ssDNA相互作用
Figure 8. Force-jump experiments to investigate the binding kinetics of TmCsp to ssDNA and dsDNA. Representative force-jump measurements of DNA Hairpin-19R52T7 in the presence of (a) 0 nmol/L, (b) 100 nmol/L, and (c) 500 nmol/L TmCsp. In panels (a)–(c), the first force jump (left) determines whether TmCsp binds to dsDNA, while the second force jump (right) assesses its interaction with ssDNA.
图 9 DNA发夹-19R52T7与TmCsp结合动力学的蛋白浓度和去折叠时间依赖性分析 (a) 100 nmol/L和500 nmol/L TmCsp溶液中, DNA发夹-19R52T7在8.0 pN下的去折叠态的存活概率, 其中实线为指数拟合, 用于确定折叠速率; (b), (c) 16.0 pN拉力条件下, 去折叠态停留时间对DNA发夹在8.0 pN下的折叠速率(b)和结合概率(c)的影响
Figure 9. Analysis of protein concentration and unfolding time dependence in the binding kinetics of TmCsp with DNA Hairpin-19R52T7. (a) Survival probability of the unfolded state for DNA Hairpin-19R52T7 at 8.0 pN in the presence of 100 nmol/L and 500 nmol/L TmCsp. The solid lines represent exponential fitting to determine the folding rates. (b), (c) Effect of unfolded state dwell time of DNA Hairpin at 16.0 pN on the folding rate at 8.0 pN (b) and the binding probability (c).
-
[1] Watson J D, Crick F H C 1953 Nature 171 737 doi: 10.1038/171737a0 [2] Travers A, Muskhelishvili G 2015 FEBS. J. 282 2279 doi: 10.1111/febs.13307 [3] Bailly C, Waring M J, Travers A A 1995 J. Mol. Biol. 253 1 doi: 10.1006/jmbi.1995.0530 [4] Virstedt J, Berge T, Henderson R M, Waring M J, Travers A A 2004 J. Struct. Biol. 148 66 doi: 10.1016/j.jsb.2004.03.007 [5] Dessinges M N, Maier B, Zhang Y, Peliti M, Bensimon D, Croquette V 2002 Phys. Rev. Lett. 89 248102 doi: 10.1103/PhysRevLett.89.248102 [6] Zhang C, Tian F J, Zuo H W, et al. 2025 Nat. Commun. 16 113 doi: 10.1038/s41467-024-55404-6 [7] Hunter C A 1993 J. Mol. Biol. 230 1025 doi: 10.1006/jmbi.1993.1217 [8] Bosco A, Camunas-Soler J, Ritort F 2014 Nucleic Acids Res. 42 2064 doi: 10.1093/nar/gkt1089 [9] Budkina K S, Zlobin N E, Kononova S V, Ovchinnikov L P, Babakov A V 2020 Biochemistry (Mosc.) 85 1 doi: 10.1134/S0006297920140011 [10] Lopez M M, Yutani K, Makhatadze G I 1999 J. Biol. Chem. 274 33601 doi: 10.1074/jbc.274.47.33601 [11] Graumann P, Marahiel M A 1994 FEBS Lett. 338 157 doi: 10.1016/0014-5793(94)80355-2 [12] Bae W, Xia B, Inouye M, Severinov K 2000 Proc. Natl. Acad. Sci. 97 7784 doi: 10.1073/pnas.97.14.7784 [13] Phadtare S, Inouye M, Severinov K 2002 J. Biol. Chem. 277 7239 doi: 10.1074/jbc.M111496200 [14] Jiang W, Jones P, Inouye M 1993 J. Bacteriol. 175 5824 doi: 10.1128/jb.175.18.5824-5828.1993 [15] Brandi A, Pietroni P, Gualerzi C O, Pon C L 1996 Mol. Microbiol. 19 231 doi: 10.1046/j.1365-2958.1996.362897.x [16] Goldenberg D, Azar I, Oppenheim A B 1996 Mol. Microbiol. 19 241 doi: 10.1046/j.1365-2958.1996.363898.x [17] Jones P G, Inouye M 1994 Mol. Microbiol. 11 811 doi: 10.1111/j.1365-2958.1994.tb00359.x [18] Mani A, Gupta D K 2015 J. Biomol. Struct. Dyn. 33 861 doi: 10.1080/07391102.2014.907747 [19] Caballero C J, Menendez-Gil P, Catalan-Moreno A, et al. 2018 Nucleic Acids Res. 46 1345 doi: 10.1093/nar/gkx1284 [20] Zhang Y, Burkhardt D H, Rouskin S, Li G W, Weissman J S, Gross C A 2018 Mol. Cell 70 274 doi: 10.1016/j.molcel.2018.02.035 [21] Horn G, Hofweber R, Kremer W, Kalbitzer H R 2007 Cell. Mol. Life Sci. 64 1457 doi: 10.1007/s00018-007-6388-4 [22] Bustamante C, Alexander L, Maciuba K, Kaiser C M 2020 Annu. Rev. Biochem. 89 443 doi: 10.1146/annurev-biochem-013118-111442 [23] Ashkin A, Dziedzic J M, Bjorkholm J E, Chu S 1986 Opt. Lett. 11 288 doi: 10.1364/OL.11.000288 [24] Zlatanova J, Lindsay S M, Leuba S H 2000 Prog. Biophys. Mol. Bio. 74 37 doi: 10.1016/S0079-6107(00)00014-6 [25] Smith S B, Finzi L, Bustamante C 1992 Science 258 1122 doi: 10.1126/science.1439819 [26] Stirnemann G, Giganti D, Fernandez J M, Berne B J 2013 Proc. Natl. Acad. Sci. 110 3847 doi: 10.1073/pnas.1300596110 [27] Xue Z Y, Sun H, Hong H Y, Zhang Z W, Zhang Y H, Guo Z L, Le S M, Chen H 2024 Phys. Rev. Res. 6 023170 doi: 10.1103/PhysRevResearch.6.023170 [28] Hong H Y, Guo Z L, Sun H, Yu P, Su H H, Ma X N, Chen H 2021 Commun. Chem. 4 156 doi: 10.1038/s42004-021-00592-1 [29] Xue Z Y, Yu P, Zhang Y H, Zhang Z W, Sun H, Hou Z Q, Hong H Y, Le S M, Chen H 2025 Phys. Rev. E 111 014413 doi: 10.1103/PhysRevE.111.014413 [30] Petrosyan R, Narayan A, Woodside M T 2021 J. Mol. Biol. 433 167207 doi: 10.1016/j.jmb.2021.167207 [31] Liang T, Yang C, Song X Y, Feng Y Y, Liu Y H, Chen H 2023 Phys. Rev. E 108 014406 doi: 10.1103/PhysRevE.108.014406 [32] Zeeb M, Balbach J 2003 Protein Sci. 12 112 doi: 10.1110/ps.0219703 [33] Lopez M M, Yutani K, Makhatadze G I 2001 J. Biol. Chem. 276 15511 doi: 10.1074/jbc.M010474200 -

 首页
首页 登录
登录 注册
注册


 下载:
下载: