-
萎缩芽孢杆菌(ATCC-9372)是一株重要的芽孢杆菌属菌株,广泛应用于农业、科研和卫生等领域[1-2]。在不良环境下,萎缩芽孢杆菌会变为具有抗逆性的芽孢,其对热、紫外线、电离辐射和某些化学物质具有较强的抗性,可用于灭菌效果研究,以及芽孢的形成、萌发机制和耐热机理研究等[3]。利用单颗粒质谱技术识别和区分营养细胞和芽孢的独特生化标志物,对理解其生物学特性和代谢途径具有重要意义。同时,ATCC-9372芽孢与一些对公共卫生安全有潜在威胁的细菌(如炭疽杆菌)有着相似的生化成分和传播方式,了解萎缩芽孢杆菌的生化特征有助于了解与其相似的致病菌,并可对其进行及时检测和预警。
单颗粒质谱技术是一种可以同时实时分析单个气溶胶颗粒的大小、数量浓度和化学成分的工具,已被应用于环境检测、生物医药等领域[4-9]。基于质谱技术分析萎缩芽孢杆菌营养细胞和芽孢的生化和形态特性的相关研究主要集中在20年前[10-14],如Tobias等[10]利用生物气溶胶质谱仪(BAMS)跟踪1组萎缩芽孢杆菌营养细胞在产孢过程中的生化和形态变化,发现在由营养细胞转变为芽孢的过程中,质谱特征逐渐丰富。近些年,单颗粒质谱技术取得了很大进展,但是,随着质谱数据处理算法的不断丰富[15-18],还未见联合先进的深度学习算法与单颗粒质谱技术区分不同状态萎缩芽孢杆菌的报道。
卷积神经网络(CNN)是深度学习的代表算法之一[19],它能够通过多层结构提取数据的层次化特征,其中更高级别的特征用于表示更抽象的数据语义[20],具有表征学习的能力。近年来,CNN已应用于质谱数据的分类领域[21-23],并取得了较好的结果。为了进一步提取分析MALDI-TOF质谱数据中的关键特征,Wang等[24]采用Score-CAM神经网络可视化方法成功提取了MALDI-TOF质谱数据中对分类贡献最大的前1%特征,并证明了该算法用于质谱关键分类特征提取结果的可信度。
本研究利用深度学习算法和分类模型可视化方法区分萎缩芽孢杆菌的营养细胞和芽孢,并从质谱离子特征角度进行分析。同时,建立萎缩芽孢杆菌质谱数据库,从生化角度分析不同状态下的萎缩芽孢杆菌,对比它们在粒径、质谱特征以及成分上的差异,并利用Score-CAM算法对一维卷积神经网络(1D-CNN)分类过程进行可视化分析,提取出1D-CNN分类过程中所依据的关键特征离子。旨为实现对萎缩芽孢杆菌营养细胞及其芽孢的准确识别与区分,为质谱数据的处理分析提供思路和方法。
-
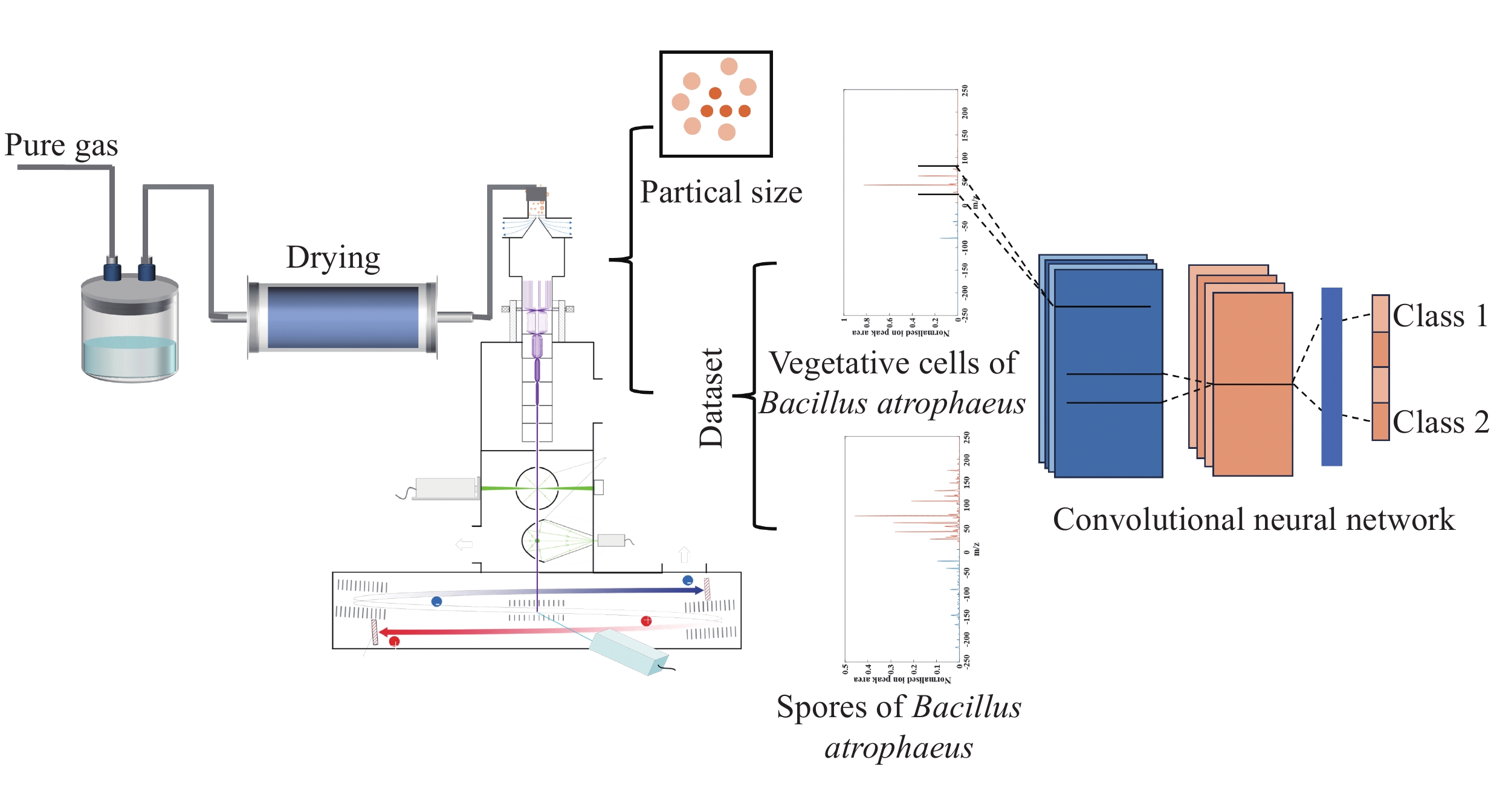
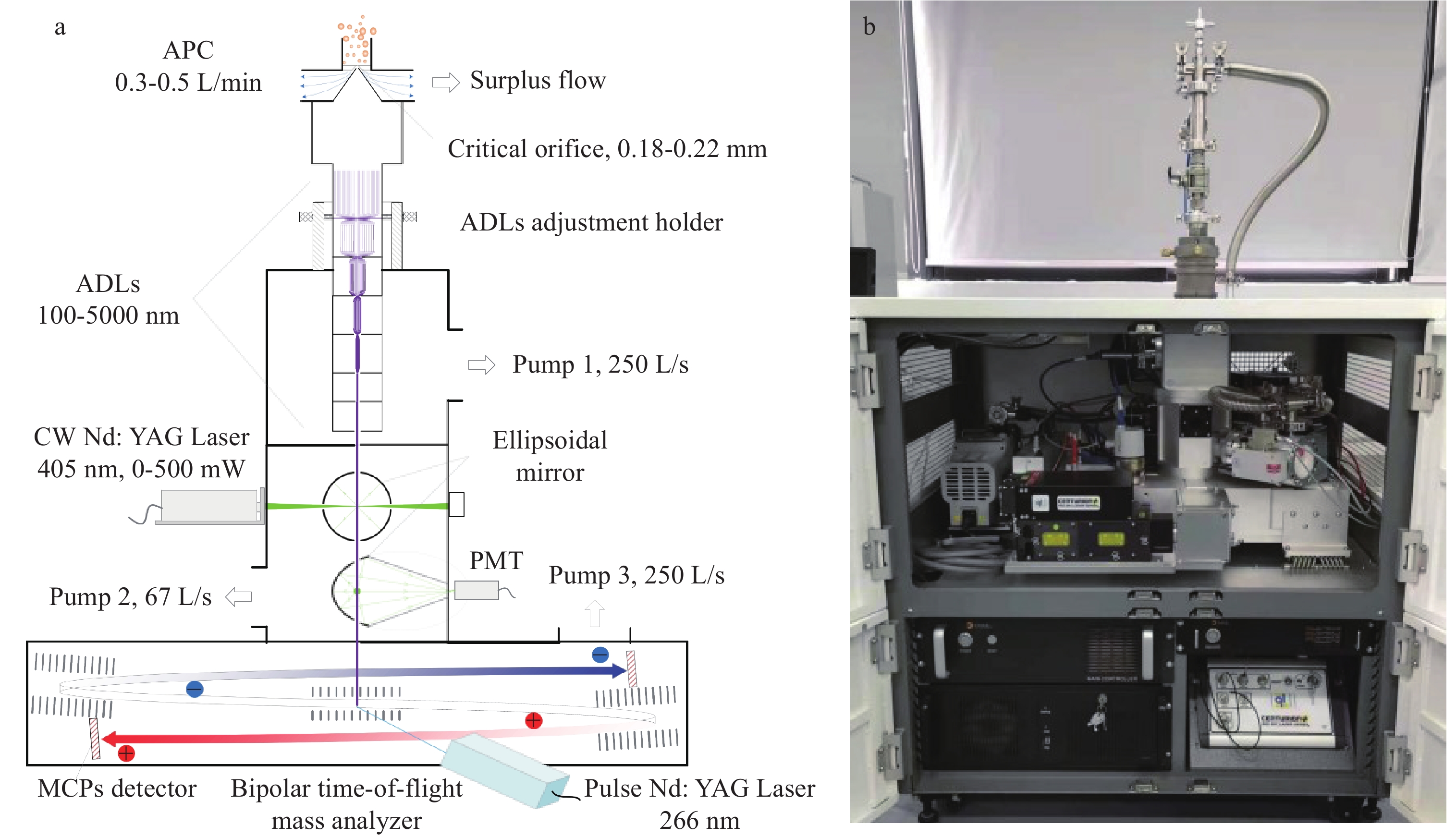
本实验采用广东禾信仪器有限公司生产的高性能-单颗粒气溶胶质谱仪(HP-SPAMS),该仪器的进样系统和实际结构示意图示于图1,具体原理参见文献[25],本文只进行简要介绍。
进样:气溶胶颗粒通过进样孔进入真空系统,多余的气体由前置泵从分离锥中抽出;在临界孔后超音速气流的加速作用下,颗粒从多余的气体中分离出来进入分离锥,然后进入缓冲腔;在缓冲腔内,颗粒的速率逐渐降低并进入下面的多级空气动力透镜组,该透镜组可以有效地将颗粒聚焦到透镜轴线上,从透镜组射出后会通过加速喷嘴再次加速。测径:颗粒离开空气动力学透镜组后进入测径区,在测径区先后经过2束连续激光器(Nd:YAG激光)发射的激光束(波长405 nm),产生的光信号分别被聚焦到光电倍增管(PMT)上得以检测;时序电路根据2个PMT信号的时间间隔计算颗粒的飞行速率,进而换算出颗粒的空气动力学直径。电离:颗粒在电离系统中被脉冲激光烧蚀和电离,产生相应的正负离子碎片。质谱分析:在电场力的作用下,正负离子碎片信号被双极飞行时间质谱仪记录,生成每个被击中颗粒的正负离子质谱图。
-
萎缩芽孢杆菌的营养细胞和芽孢悬液均购自中国工业微生物菌种保藏管理中心。萎缩芽孢杆菌营养细胞样品的制作流程如下:1)培养。用甘油菌和液体培养基以1:1 250比例配制菌液,置于摇床中培养(37 ℃,120 r/min),在15、18、21 h(位于细菌培养的对数期与稳定期)3个时间点分别取样30 mL至离心管中。2)离心。将样品以4 000 r/min转速离心3次以沉淀细菌,每次5 min,最后得到的溶剂为去离子水的细菌溶液。3)发生和干燥。将细菌溶液摇匀后倒入洁净的TSI-9302气溶胶发生器中,潮湿的气溶胶会影响质谱仪的打击效率,因此需要在发生器与质谱仪之间添加干燥管,同时为了避免细菌交叉污染,每次正式发生之前,都会用去离子水进行发生,直至质谱仪检测不到颗粒。4)测试。细菌气溶胶以0.3 L/min流速进入质谱仪中,开始粒径和质谱数据的收集。芽孢样品是将购买的芽孢悬液稀释至108 CFU/mL后进行气溶胶发生与测试。
-
基于以上实验流程建立用于训练和测试的数据集。其中,营养细胞和芽孢的数据集分别包括7 000、5 000张质谱数据,将数据集按照7:3比例分为训练集和测试集。为了评价模型的分类稳定性和实验的可重复性,采用相同的实验方法建立各3 000张营养细胞和芽孢的验证集。
数据集建立方法如下:不同质谱图对应的向量长度不同,为满足1D-CNN的输入要求(输入数据的长度应一致),本实验采用分箱法,即将m/z以间隔1划分,在每个获得的区间内取峰面积之和作为该m/z所对应的峰面积,这样就可以将每个粒子的正质谱和负质谱映射到1个500维的向量X上,每个向量Xj(j=1,2,3……500)对应不同的质荷比(m/z −250,−249,−248……+250);在映射前,根据式(1)对峰高进行数值归一化处理,其中Sij表示第i个质谱图的第j个质荷比的峰高,Sij*表示归一化后的峰高,N表示质谱图的个数(代表数据集的大小)。
对整理好的数据集添加数字标签,营养细胞和芽孢对应的数字标签分别为1和2,在分析分类模型的分类结果时依据数字标签对应不同的细菌。在进行模型训练时,将数据集按照7:3比例划分为训练集和测试集,并将数据全部打乱。
-
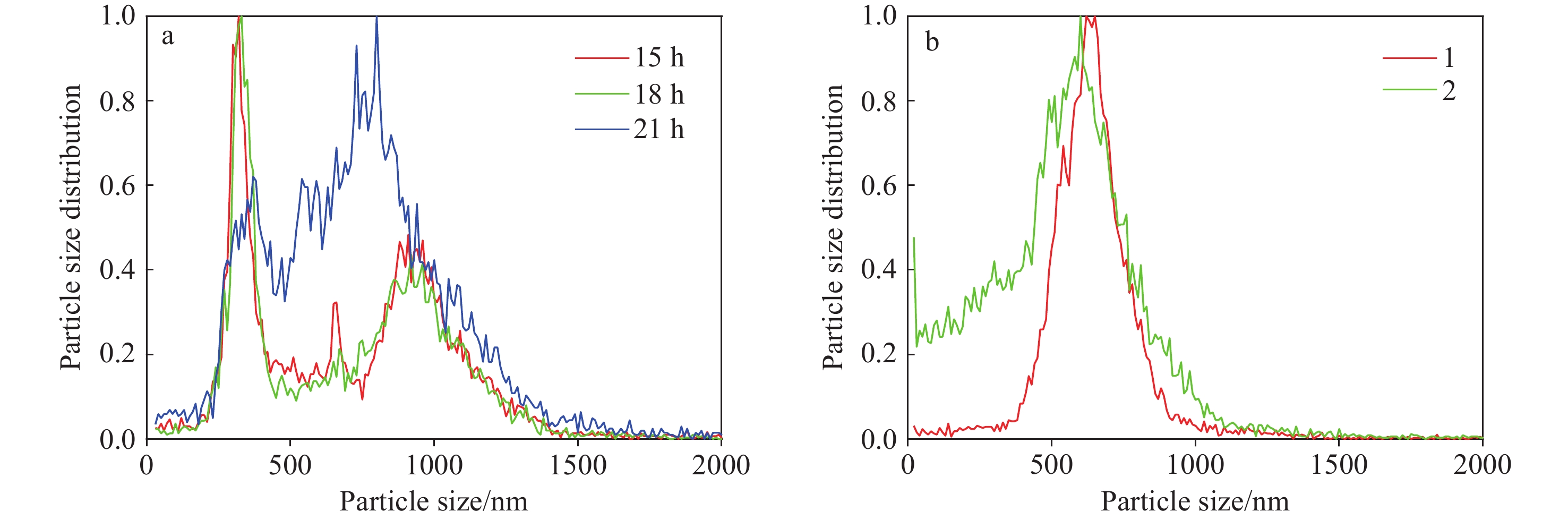
本实验利用单颗粒质谱仪收集的是单个气溶胶颗粒的空气动力学粒径,3个时间点采样的营养细胞和2次实验收集的芽孢的粒径分布示于图2。可以看出,营养细胞宽度为~0.33 μm,长度为~0.9 μm,且不同采样时间点的粒径大小基本一致;芽孢粒径主要集中在0.62 μm左右,2次针对芽孢的测量结果基本一致。而实际的营养细胞为棒状,宽0.5~1.0 μm、长2.0~4.0 μm,粒径大小会随着生长条件变化而略有不同[3];芽孢为椭圆形,在干燥状态下的宽度为~0.7 μm,长度为~1.8 μm[26]。整体上,萎缩芽孢杆菌的芽孢粒径小于营养细胞,且芽孢的粒径分布比营养细胞更集中,该特点与文献[10]的研究结果较为一致。从粒径的区别可以推断出,萎缩芽孢杆菌在由营养细胞态向芽孢态的转变过程中会发生细胞壁增厚、细胞内容物被压缩等生物学以及形态学变化。质谱仪测得的粒径比实际情况偏小,主要是因为空气动力学透镜对大颗粒的传输效率较低,在运输过程中易产生惯性撞击损失;同时,粒径结果还受进样小孔处的进样压强影响,当进样压强低于标准值范围时,测得的粒径偏小[25]。
-
经过前期对比,本实验选择包含6个卷积单元的1D-CNN进行分类模型训练。1D-CNN包括输入层、卷积层、归一化层、激励层、全连接层、softmax层和输出层,其中卷积层、归一化层、激励层构成1个卷积单元,卷积单元的个数等于卷积层数。该神经网络的结构参数如下:采用“Adam”算法作为分类模型的优化算法,输入数据的维度对应每张质谱图的向量维度,为500×1×1;卷积单元中,卷积核的大小为3×1,批归一化层中的批大小为128,选用RuLU函数作为激活层的激活函数;初始化学习率为0.001,经过100轮训练后,学习率下降为0.000 1;每次迭代开始前将数据集打乱以提高模型的泛化能力。对模型的训练轮数共计200,利用训练集对分类模型的训练时间约为11 min,利用训练好的分类模型进行识别,在不包括数据导入时间的前提下,分类模型在0.4 s左右即可得出3 000张质谱数据的识别结果,该算法具有较高的识别效率,极大降低了对质谱数据分类的时间成本。
-
1D-CNN在测试集和验证集上的分类结果列于表1,其中,测试集包含的质谱数据量分别占各自数据集的3/10。可以看出,针对在实验室环境下收集的质谱数据,该模型的识别准确率均在99%以上,模型的分类效果和泛化性能较好。准确的识别结果证明CNN可以有效学习不同种类细菌的质谱离子分布及其强度特征,并进行准确地识别。
-
Score-CAM算法是Wang等[27]提出的一种CNN可视化方法,其生成的可视化结果在视觉效果和定位性能上均有较好的效果,常被用于图像分类领域。本实验基于Matlab R2022b平台将该算法应用于质谱分析领域,参照Wang等[15]的特征提取方法,检验不同离子作为信息特征的重要性,判断1D-CNN模型准确识别2种状态萎缩芽孢杆菌的分类依据。
-
因为同种细胞不同个体之间的组成或其他特性存在微小差异,且受激光能量波动等外界因素的影响,单次测量单个微生物细胞的质谱图很难包含该种微生物的所有典型的特征离子,所以,本实验基于分类模型对验证集的分类结果,对分类正确的质谱数据进行求平均运算,得到2种实验对象的平均质谱图,并对质谱图中的典型特征离子进行标注。平均质谱图可以充分代表2种样品的质谱特征,利用分类模型对平均质谱图进行识别,均得到准确的分类结果。
萎缩芽孢杆菌营养细胞和芽孢平均质谱图中的典型特征离子及其成分来源列于表2。本实验将文献中已有明确成分来源的离子准确性标为准确,对来源尚未确定、仅限于推测或者还未提及过的离子标为尚未确定。值得一提的是,本实验采用的单颗粒质谱仪的分辨率在500~2 500之间,此分辨率下难以进行同分异构体的区分,另外,质量轴可能存在不稳定的问题,导致离子指认的错误发现率(FDR)提高。但是,文献[12,28-29]中介绍的离子指认方法具有较高的可靠性:利用在未标记、13C标记和15N标记的生长介质中生长的孢子的质谱来确定与正离子和负离子模式质谱中观察到的每个质谱峰相关的碳和氮原子的数量,进而确定相应离子的化学式;通过独立确定几种化学标准品的裂解模式来确定与碎片离子峰相关的母离子结构;将液相色谱与质谱联用进行代谢组学分析等。以上方法在一定程度上解决了由于同分异构体的存在而导致离子指认错误发现率提高的问题,为今后进一步确定更多离子的化学表达式及其成分来源提供了思路。
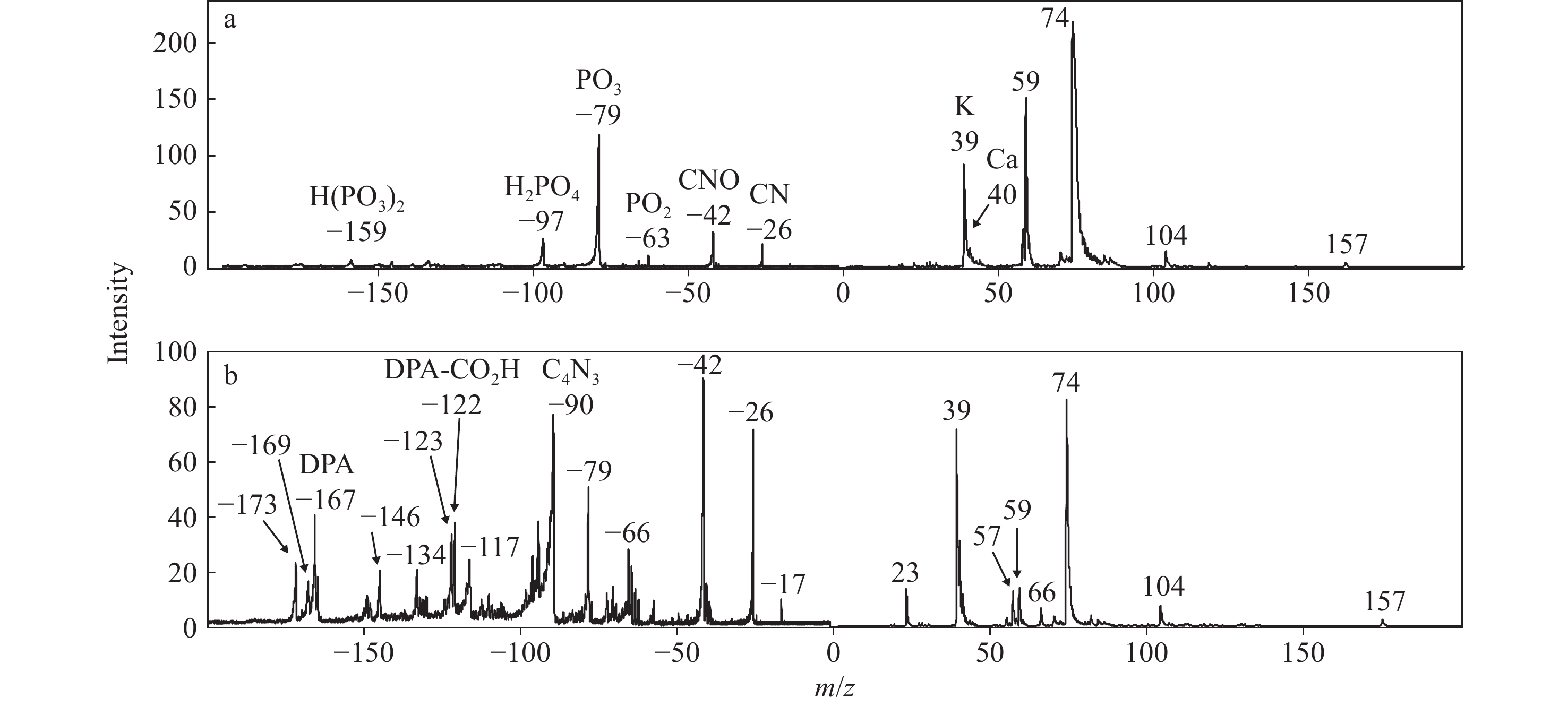
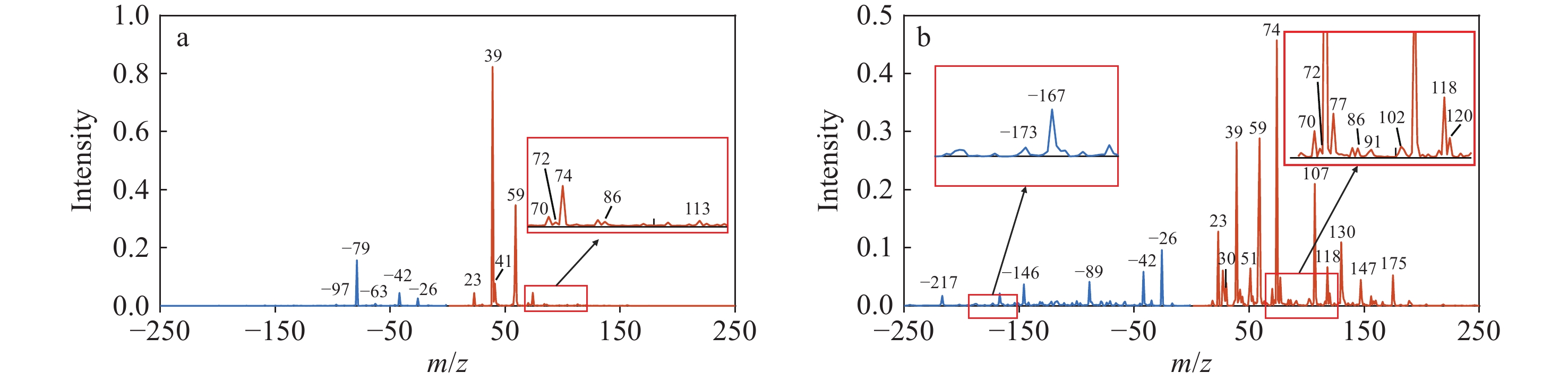
Tobias等[10]收集的萎缩芽孢杆菌营养细胞及其产生的芽孢的质谱图示于图3。本实验结果中营养细胞的质谱特征与Tobias等[10]利用BAMS得到的实验结果高度相似,负离子模式谱图中主要包括97HPO4−、79PO3−、63PO2−、42CNO−、26CN−等离子,来自含磷和氮的化合物,其中磷酸根主要来自细菌的核酸、三磷酸腺苷以及细胞膜等成分,有机氮离子主要来自细菌内的蛋白质成分(尤其是具有额外官能团的氨基酸,如谷氨酸、精氨酸)以及一些细菌代谢产物等[14,25]。图3、4中营养细胞的正离子模式谱图中包含的离子成分较为一致,但是离子的强度分布有差异。本实验结果中,信号最强的4个离子依次是39K+、59C3NH9+、41K+和23Na+,其中,39K+和23Na+是细菌细胞中最常见的金属离子[25,30],在孢子萌发的早期阶段,孢子内部会释放出80%的39K+和23Na+[32],除此之外,还有一些强度较弱的氨基酸去羧基离子峰,如70[Proline−COOH]+、72[Valine−COOH]+、74[Threonine−COOH]+、86[Leucine−COOH]+[30],这些离子在芽孢态的萎缩芽孢中也存在,但信号强度较弱。这表明,孢子中存在少量的脯氨酸、缬氨酸、苏氨酸、亮氨酸等氨基酸,该结果与Srivastava等[12]关于萎缩芽孢杆菌孢子的研究结果较为一致。但是,Srivastava 团队通过同位素标记实验和化学标准品实验确定了m/z +74对应的离子为74N(CH3)4+,来自孢子中的甜菜碱成分,并未提到m/z 74是74[Threonine−COOH]+的可能。而图3a正离子谱图中信号强度最强的4个离子依次是74[Threonine−COOH]+、59C3NH9+、39K+和40Ca+。虽然质谱仪的工作原理、电离激光的波长和实验对象的种类相同,但实验样品的状态不同,样品中特定分子的浓度会有差异,浓度越高,离子强度越高。此外,本研究使用单颗粒质谱仪的一些参数(如检测器的灵敏度和进样结构等)与Tobias等[10]使用的质谱仪相关参数存在差异,以上原因都会造成质谱图中离子信号强度分布不同。
通过分析图3b、4b可以发现,与营养细胞相比,芽孢的质谱特征更丰富,这主要是因为芽孢为了在极端条件生存会在形成过程中积累特定的生化物质,如二吡啶酸(DPA,化学式为C7H5NO4)、钙离子、一些特殊的蛋白质和多糖以及包括小分子酸可溶性蛋白在内的保护性分子等,这些物质在营养细胞中不存在或含量较低;此外,芽孢是代谢几乎完全停止的休眠细胞,代谢状态的不同也会导致质谱特征的差异。
图4b中,与营养细胞相比,芽孢的负离子谱图中增多的典型离子主要包括m/z −89、146[glutamate−H]−、173[arginine−H]−、167[DPA]−、m/z −217等。有研究[12]表明,在芽孢态的萎缩芽孢杆菌中,DPA占其干重的5%~15%,且L-谷氨酸和L-精氨酸的含量较高,可达0.1 mol/L。但是,磷酸根离子的信号强度明显降低,可能是因为其主要存在于孢子内核酸、腺苷二磷酸和三磷酸以及细胞膜中[11],而孢子的最外层主要是由蛋白质构成的孢子外壳[33],质谱图中来自孢子外壳特征离子的信号强度会相对较强。图4b与图4a相比,芽孢的正离子谱图中,74[Threonine−COOH]+的信号强度明显增强,新出现的典型离子主 要 在 m/z +74~+175 范围内,包括m/z +107、+118、+130、+147、+175。图4b与图3b相比,芽孢负离子谱图中的90C4N3−信号强度较低,正离子谱图中没有明显的含钙化合物离子,如57CaOH+、66CaCN+和82CaCNO+,而是更多较高m/z的离子,目前还未见这些离子的相关文献报道。根据Driks[31]对枯草芽孢杆菌外壳生化成分的研究分析可知,芽孢外壳除含有少量的碳水化合物和脂质等物质外,主要是由蛋白质组成的。因此,推测m/z +74~+175中的高信号强度离子可能主要来自芽孢外壳的蛋白质,准确来源还需进一步研究。
-
Wang等[27,33]已详细介绍过Score-CAM算法的原理和可靠性,在原理不变的基础上,本工作对该算法做出了针对质谱分析的适应性改进。首先,数据输入,将若干个数据维度为500×1×1的质谱向量输入到训练好的1D-CNN模型中,利用activations 函数获取指定卷积层输出的激活图和不同离子的得分(Score),激活图也叫特征图,能够反映输入数据的不同区域对卷积核的激活程度,本实验选择卷积层的最后一层来获取激活图;然后,将N个激活作为掩码与最初的输入相乘,乘积代入模型,得到N个新的输出分数Score’;Score’与Score作差,得到置信度增量CIC,CIC归一化后的结果作为卷积层输出激活图的权重;将权重与激活图相乘,乘积在通道维度上求和,再对求和结果进行归一化处理,即可得到不同m/z离子的得分。
-
基于以上Score-CAM算法流程,随机选择100条质谱数据导入到训练好的二分类1D-CNN模型中,得到每条质谱数据所有m/z得分,将不同m/z离子按照得分从高到低排序,得到1个500×100的矩阵向量,其中,500是每条质谱数据的向量长度,每一列的500个数分别对应每一质谱图中不同m/z离子根据得分由高到低排列,100代表质谱数据的个数。本工作选取矩阵的前10行,计算其中不同m/z离子的出现次数,按照出现次数的大小对不同离子进行排列,得到的结果是综合100条质谱数据不同m/z离子对分类贡献程度的排序;选取前20个贡献大的离子进行分析,并从中剔除在实际质谱图中强度接近或者等于0的m/z信号,从而实现对重要特征离子的选择。
特征离子的选择结果列于表3。对比发现,对营养细胞和芽孢分类贡献大的高分m/z离子大多不重叠,且均为2种状态细胞质谱图中差异性较强的特征离子,只有26CN−和23Na+在2种细胞中均取得了较高分数。首先,从营养细胞的Score-CAM结果中选出8个特征离子,包括正离子和负离子各4个,除m/z +113对应的离子外,其余7个离子均在文献[10]中被报道过,并且它们在平均质谱图中的信号峰均比较明显。从芽孢的Score-CAM结果中选出15个特征离子,4个高分负离子在芽孢平均质谱图中的信号强度均较强,但高分正离子并非在质谱图中均具有较强的信号强度,如m/z +91、+102;同时,芽孢质谱图中有些信号较强离子的得分不在前20,如59C3NH9+和74[Threonine−COOH]+,它们均存在于萎缩芽孢杆菌的营养细胞和芽孢中,且均属于高强度信号。因此,本工作认为不同m/z的Score-CAM得分与信号强度没有必然联系,而与离子的特殊性相关。
从高分特征离子追溯到细胞对应的成分有助于判别萎缩芽孢杆菌由营养细胞状态转化为芽孢态后发生的成分变化。如在4.1节提到的167[DPA]−离子就来源于芽孢中典型的区别于营养细胞的成分二吡啶酸,它是萎缩芽孢杆菌芽孢中的主要组成部分,也是芽孢抗性的重要标志物,与Ca2+形成的复合物能够吸收水分并降低芽孢内部的水分活性,有助于提高芽孢的热稳定性和抗干燥能力[32];芽孢中的146[glutamate−H]−和173[arginine−H]−分别来自芽孢中的精氨酸和谷氨酸,有研究[12]表明,在DPA存在时,精氨酸和谷氨酸的有效电离效率增加了1个数量级,它们在孢子中位置很近,可能与DPA一起处于核心位置,有利于离子的形成;此外,与营养细胞相比,芽孢质谱图中磷酸根离子峰的信号强度较弱,这主要是因为营养细胞的代谢活动比较旺盛,而磷酸盐在所有代谢过程中都很重要,且代谢活跃的细胞中存在更多形式的磷酸盐[29]。以上这些可成分溯源且信号强度不同的离子均是在Score-CAM结果中得分高的离子,验证了该算法对质谱关键分类特征提取的准确性。
-
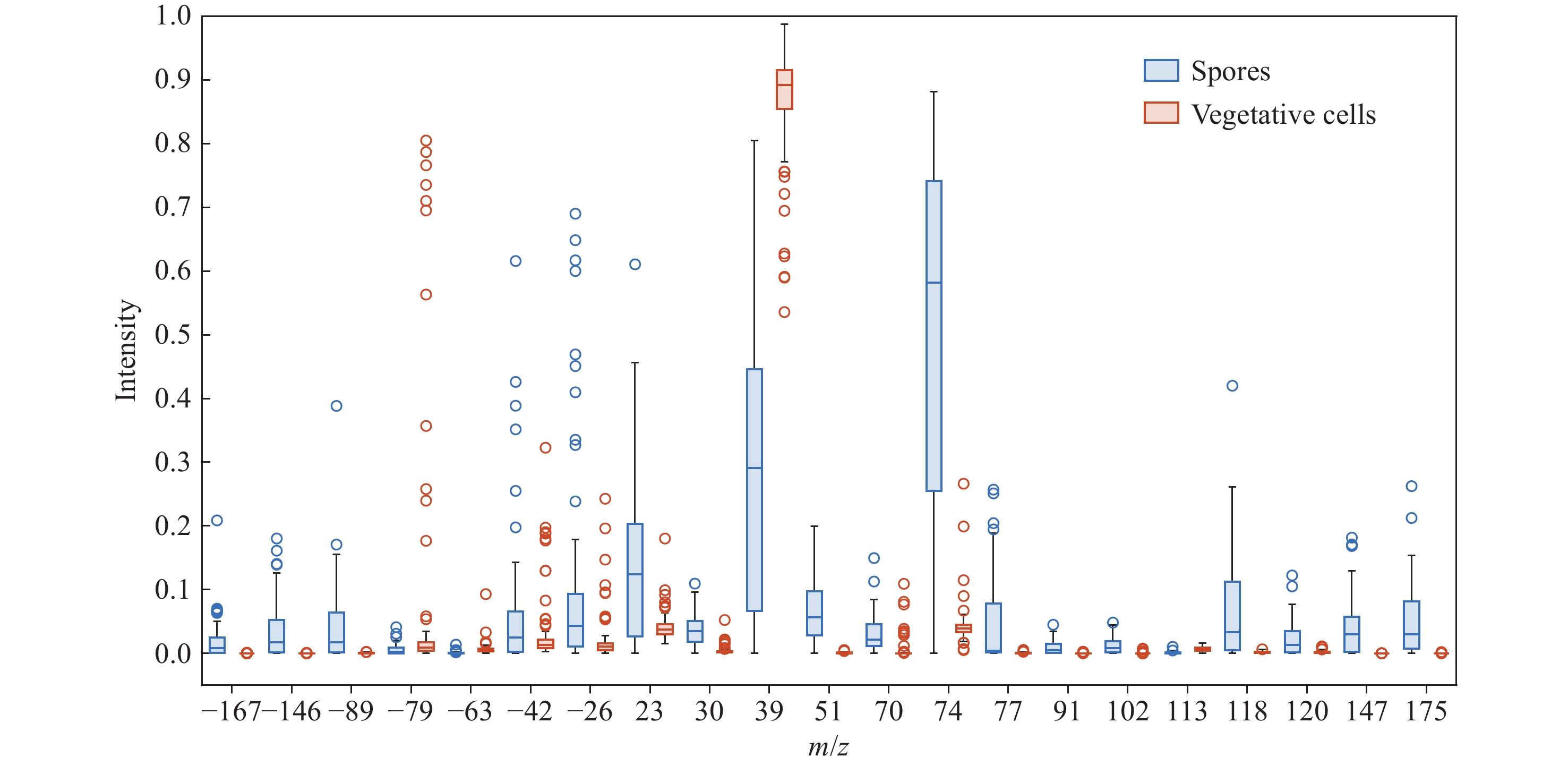
为了判断Score-CAM算法特征离子选择结果的可靠性,更好地观察以上高分离子在2种细胞之间的差异性,随机选择被1D-CNN模型正确识别的100张质谱图,从中提取出这些离子的信号强度并绘制箱型图,纵坐标代表每种离子信号归一化之后的强度,示于图5。可以看出,这些高分离子的信号强度分布存在明显差异,芽孢离子的信号强度波动程度大于营养细胞;有些离子的信号强度有较多的异常值,这些信号强度的变化是正常的,可能与激光能量的波动和生物成分的变化有关[10]。
现有文献还无法确定m/z −89、+51、+77、+91、+102、+113、+147 这 7 个离子的准确化学式,为了判定这几个新观察到的高分离子是否有可能成为种类标志离子,还需对它们的稳定性和可重复性进行判断。本工作再次分别采集了培养16 h的萎缩芽孢杆菌营养细胞和芽孢的3 000张质谱数据并进行分类模型训练。训练好的模型对营养细胞和芽孢的识别准确率分别为97.9%、99.77%。本工作将以上7个离子归一化后的信号强度与图4中的信号强度进行对比,结果列于表4。可以发现,2次测试的离子信号强度基本相同,因此,以上7个高分离子有着良好的稳定性和重复性,具有成为种类标志物的潜力。
相对于传统的通过肉眼观察来提取质谱图中特异性离子的方法,在单颗粒质谱分析领域,Score-CAM算法帮助我们打开了CNN这个“黑匣子”。首先,解释CNN模型有助于建立人们对分类模型的信心,促进深度学习算法在质谱识别领域的应用,对探索芽孢与营养细胞之间的区别具有重要作用,本实验结果表明CNN模型能够自动检测到质谱图中的重要特征。其次,该算法可以帮助研究人员判断能够区分营养细胞与芽孢的有效特征离子,发现以前被忽略的、可靠的重要标志离子,如m/z −89、+51、+77、+91、+102、+113、+147。在现有文献中,这些离子没有明确的化学式参考,但是在营养细胞或芽孢中的信号强度均较强。因此,可以采用同位素标记等方法确定这些离子的化学式,从而确定它们的成分来源,并与生物标志物进行关联。若将该方法应用于病原体鉴定、代谢途径观察以及疾病诊断等领域,可有效帮助提升鉴定和诊断的准确率。
-
本研究采用单颗粒质谱技术,利用1D-CNN算法区分萎缩芽孢杆菌的营养细胞和芽孢,分析它们的粒径和质谱特征,并利用Score-CAM算法分析不同m/z离子的分类贡献。结果发现,该模型在测试集和验证集上的识别准确率均在99%以上;芽孢的质谱特征更加丰富,这与其成分的变化直接相关;最后,利用箱型图展示Score-CAM结果中高分特征离子的信号强度差异,并验证了7种高分离子成为种类标志物的潜在可能。值得注意的是,本实验没有在大气环境中进行萎缩芽孢杆菌的检测与识别,在后续实验中将利用质谱仪在空气背景下检测萎缩芽孢杆菌,并对单颗粒质谱仪的检测灵敏度进行测试。
本研究将CNN应用于萎缩芽孢杆菌营养细胞及其芽孢的分类识别,并将Score-CAM算法应用于提取其营养细胞和芽孢的一些关键特征。发现不同种类细菌的质谱特征非常相似,难以直接找出它们的特征离子,因此,在后续研究中,还可以将该方法应用于不同种细菌的区分。本研究为质谱数据的处理分析提供了新的思路与方法,同时,鉴于萎缩芽孢杆菌与一些致病菌(如炭疽杆菌)的相似性,通过快速识别和区分营养细胞与芽孢,可以提高对高危致病菌的检测和响应能力。
联合单颗粒质谱与Score-CAM算法判定分析萎缩芽孢杆菌营养细胞和芽孢
Combined Single Particle Mass Spectrometry and Score-CAM Algorithm for Differentiation and Analysis of Vegetative Cells and Spores of Bacillus atrophaeus
-
摘要: 萎缩芽孢杆菌(ATCC-9372)是一株重要的芽孢杆菌属菌株,利用单颗粒质谱技术区分萎缩芽孢杆菌营养细胞和芽孢的独特生化标志物,对理解其生物学特性和代谢途径具有重要意义。近年来,国内外单颗粒质谱技术取得了很大进展,但是,随着质谱数据处理算法的不断丰富,还未见联合先进的深度学习算法与单颗粒质谱技术区分不同状态萎缩芽孢杆菌的报道。本研究利用深度学习算法和分类模型可视化方法区分萎缩芽孢杆菌的营养细胞和芽孢,并从粒径和质谱离子特征角度进行分析。通过对比粒径发现,营养细胞的粒径大于芽孢,不同采样时间点的营养细胞的粒径大小基本一致。另外,采用相同的方法建立用于训练及测试分类模型的数据集和用于评价模型分类稳定性的验证集,发现模型在测试集和验证集上的识别准确率均在99%以上;对Score-CAM结果中得分高的特征离子进行成分溯源分析,通过箱型图展现了这些特征离子信号强度的分布差异。本研究从生化角度对不同状态下的萎缩芽孢杆菌进行深入分析,可为质谱数据的处理分析提供思路和方法。Abstract: Bacillus atrophaeus (ATCC-9372) is an important strain of the Bacillus genus. The use of single particle mass spectrometry to distinguish unique biochemical markers of vegetative cells and spores of Bacillus atrophicus is important for understanding their biological properties. The main objective of this study is to distinguish vegetative cells and spores of Bacillus atrophaeus by analyzing the diameter and characteristic mass spectrometry ions of Bacillus atrophaeus by combined using of deep learning algorithms and classification model visualization methods. Firstly, the samples were prepared by collecting and centrifuging Bacillus atrophaeus that has been cultured for a certain period, and the spore samples of Bacillus atrophaeus were diluted. Then, single particle mass spectrometry was used to collect particle size and mass spectrometry data for the above two samples and to construct mass spectrometry datasets for the two objects. Following this, the particle sizes of the two samples were compared, and the datasets were divided. Based on the Matlab platform, a Convolutional Neural Network (CNN) classification model was trained to analyze the experimental results. Lastly, the typical ion characteristics of each were analyzed according to the average mass spectra, and the CNN classification process was visually analyzed using the Score-CAM algorithm. The differential ion characteristics between the vegetative cells and spores of Bacillus atrophaeus were extracted and analyzed. It was found that the particle size of vegetative cells is larger than that of spores, and the particle size of vegetative cells is essentially consistent at different sampling times. The CNN classification model achieves an accuracy of over 99% on both the test set and the validation set, indicating that the CNN model can fully learn and analyze the mass spectrometry characteristics. Their respective typical ion characteristics were analyzed by comparing the average mass spectra, which led to the introduction of their compositional differences, but not all typical ions could be accurately identified. Finally, a source analysis was performed on the ions with high scores in the Score-CAM results, and box plots demonstrated significant differences in the signal intensity of these high-scoring characteristic ions between the two states of Bacillus atrophaeus. Repeated experiments showed that the discovered high-scoring characteristic ions in the vegetative cells and spores of Bacillus atrophaeus have good stability and repeatability, suggesting their potential as species markers. This study performs an in-depth analysis of Bacillus atrophaeus in different states from a biochemical point of view, providing new insights into and methods for the processing and analysis of mass spectrometry data.
-
Key words:
- single particle mass spectrometry /
- Bacillus atrophaeus /
- vegetative cells /
- spores /
- 1D-CNN /
- Score-CAM .
-
-
表 1 1D-CNN在测试集和验证集上的分类结果
Table 1. Classification results of 1D-CNN on test and validation sets
分类对象
Classified object识别对象总数目
Number of identified object分类结果
Classification result识别准确率
Recognition accuracy/%营养细胞
Vegetative cell芽孢
Spore测试集 营养细胞 2100 2098 2 99.90 芽孢 1500 0 1500 100.00 验证集 营养细胞 3000 2999 1 99.97 芽孢 3000 3 2997 99.86 表 2 萎缩芽孢杆菌营养细胞和芽孢平均质谱图中的典型特征离子及其成分来源
Table 2. Typical ionic features in the average mass spectra of vegetative cells and spores of Bacillus atrophaeus and the origin of their components
萎缩芽孢杆菌
Bacillus atrophaeus离子化学式
Ionic chemical formula成分来源
Source of ingredient来源准确性
Accuracy of source参考文献
Reference营养细胞中典型正离子 23Na+、 39K+、41K+ 金属离子/无机盐离子 准确 [12] 59C3NH9+ 含氮有机碎片 准确 [25,30] 70[Proline−COOH]+、72[Valine−COOH]+、86[Leucine−COOH]+ 氨基酸残基 准确 [12,14,25,29] 74[Threonine−COOH]+或74N(CH3)4+ 氨基酸残基/甜菜碱 尚未确定 [12,29] 营养细胞中典型负离子 97HPO4−、79PO3−、63PO2−、 核酸、腺苷二磷酸和三磷酸
细胞膜准确 [14,25] 42CNO−、26CN− 蛋白质、细菌代谢产物 准确 [12,14,25,29] 芽孢中典型正离子 23Na+、 39K+ 金属离子/无机盐离子 准确 [12] 59C3NH9+ 含氮有机碎片 准确 [25,30] m/z +51、m/z +91、
m/z +102未知 尚未确定 118N(CH3)2CH2COOH+ 质子化甜菜碱 尚未确定 [12] 120[Phenylalanine−COOH]+ 氨基酸残基 准确 [25] m/z +107、m/z +130、
m/z +147、m/z +175芽孢外壳 尚未确定 [31] 70[Proline−COOH]+、72[Valine−COOH]+、86[Leucine−COOH]+ 氨基酸残基 准确 [12,14,25,29] 74[Threonine−COOH]+或74N(CH3)4+ 氨基酸残基/甜菜碱 尚未确定 [12,29] 芽孢中典型负离子 42CNO−、26CN− 蛋白质、细菌代谢产物 准确 [12,14,25,29] 146[glutamate−H]−、173[arginine−H]−、 去质子化氨基酸 准确 [10,12] 167[DPA]− 二吡啶酸 准确 [10,11,29] m/z −89、m/z −217 未知 尚未确定 表 3 特征选择结果
Table 3. Feature selection results
分析对象
Object of analysis负离子
Negative ion正离子
Positive ion营养细胞 79PO3−、63PO2−、
42CNO−、26CN−23Na+、70[proline−COOH]+
74[Threonine−COOH]+、m/z +113芽孢 26CN−、m/z −89、
146[glutamate−H]−、167[DPA]−23Na+、30[Glycine−COOH]+、39K+、m/z +51、m/z +77、m/z +91、m/z +102、118N(CH3)2CH2COOH+、120[Phenylalanine−COOH]+、m/z +147、175[C6H14N4O2+H]+ 表 4 2次测试结果中新发现的7种高分离子特征信号强度对比
Table 4. Comparison of the intensity of the seven newly identified characteristic signals of high-fraction ions in the results of the two tests
质荷比
m/z归一化后的信号强度-1
Normalised signal strength-1归一化后的信号强度-2
Normalised signal strength-2−89 0.0416 0.0444 +51 0.0647 0.0652 +77 0.0489 0.0492 +91 0.0088 0.0089 +102 0.0123 0.0126 +113 0.0059 0.0071 +147 0.0450 0.0476 -
[1] LEWIS D L, ARENS M. Resistance of microorganisms to disinfection in dental and medical devices[J]. Nature Medicine, 1995, 1(9): 956 -958 . doi: 10.1038/nm0995-956[2] DANCER S J D. Importance of the environment in meticillin-resistant staphylococcus aureus acquisition: the case for hospital cleaning[J]. The Lancet Infectious Diseases, 2008, 8(2): 101 -113 . doi: 10.1016/S1473-3099(07)70241-4[3] 刘波, 李红, 姚粟, 李金霞, 程池. 枯草芽孢杆菌黑色变种ATCC 9372的特性及其应用[J]. 中国消毒学杂志, 2009, 26(2): 236 -237, 241 . LIU Bo, LI Hong, YAO Su, LI Jinxia, CHENG Chi. Characteristics and application of Bacillus subtilis black variety ATCC 9372[J]. Chinese Journal of Disinfection, 2009, 26(2):236 -237, 241 (in Chinese).[4] 李磊. 单颗粒气溶胶质谱仪的改进及环境应用[D]. 上海: 上海大学, 2014. [5] SULTANA C M, AL-MASHAT H, PRATHER K A. Expanding single particle mass spectrometer analyses for the identification of microbe signatures in sea spray aerosol[J]. Analytical Chemistry, 2017, 89(19): 10 162-10 170. [6] ZHANG Y, PEI C, ZHANG J, CHENG C, LIAN X, CHEN M, HUANG B, FU Z, ZHOU Z, LI M. Detection of polycyclic aromatic hydrocarbons using a high performance-single particle aerosol mass spectrometer[J]. Journal of Environmental Sciences, 2023, 124: 806 -822 . doi: 10.1016/j.jes.2022.02.003[7] YANG J, MA S, GAO B, LI X, ZHANG Y, CAI J, LI M, YAO L A, HUANG B, ZHENG M. Single particle mass spectral signatures from vehicle exhaust particles and the source apportionment of on-line PM2.5 by single particle aerosol mass spectrometry[J]. Science of the Total Environment, 2017, 593: 310 -318 .[8] ZAWADOWICZ M A, FROYD K D, MURPHY D M, CZICZO D J. Improved identification of primary biological aerosol particles using single-particle mass spectrometry[J]. Atmospheric Chemistry and Physics, 2017, 17(11): 7 193-7 212. [9] TONER S M, SHIELDS L G, SODEMAN D A, PRATHER K A. Using mass spectral source signatures to apportion exhaust particles from gasoline and diesel powered vehicles in a freeway study using UF-ATOFMS[J]. Atmospheric Environment, 2008, 42(3): 568 -581 . doi: 10.1016/j.atmosenv.2007.08.005[10] TOBIAS H J, PITESKY M E, FERGENSON D P, STEELE P T, HORN J, FRANK M, GARD E E. Following the biochemical and morphological changes of Bacillus atrophaeus cells during the sporulation process using bioaerosol mass spectrometry[J]. Journal of Microbiological Methods, 2006, 67(1): 56 -63 . doi: 10.1016/j.mimet.2006.03.001[11] FERGENSON D P, PITESKY M E, TOBIAS H J, STEELE P T, CZERWIENIEC G A, RUSSELL S C, LEBRILLA C B, HORN J M, COFFEE K R, SRIVASTAVA A, PILLAI S P, SHIH M P, HALL H L, RAMPONI A J, CHANG J T, LANGLOIS R G, ESTACIO P L, HADLEY R T, FRANK M, GARD E E. Reagentless detection and classification of individual bioaerosol particles in seconds[J]. Analytical Chemistry, 2004, 76(2): 373 -378 . doi: 10.1021/ac034467e[12] SRIVASTAVA A, PITESKY M E, STEELE P T, TOBIAS H J, FERGENSON D P, HORN J M, RUSSELL S C, CZERWIENIEC G A, LEBRILLA C B, GARD E E, FRANK M. Comprehensive assignment of mass spectral signatures from individual Bacillus atrophaeus spores in matrix-free laser desorption/ionization bioaerosol mass spectrometry[J]. Analytical Chemistry, 2005, 77(10): 3 315-3 323. [13] STEELE P T, TOBIAS H J, FERGENSON D P, PITESKY M E, HORN J M, CZERWIENIEC G A, RUSSELL S C, LEBRILLA C B, GARD E E, FRANK M. Laser power dependence of mass spectral signatures from individual bacterial spores in bioaerosol mass spectrometry[J]. Analytical Chemistry, 2003, 75(20): 5 480-5 487. [14] TOBIAS H J, SCHAFER M P, PITESKY M, FERGENSON D P, HORN J, FRANK M, GARD E E. Bioaerosol mass spectrometry for rapid detection of individual airborne Mycobacterium tuberculosis H37Ra particles[J]. Applied and Environmental Microbiology, 2005, 71(10): 6 086-6 095. [15] WANG H Y, HSIEH T T, CHUNG C R, CHANG H C, HORNG J T, LU J J, HUANG J H. Efficiently predicting vancomycin resistance of Enterococcus faecium from MALDI-TOF MS spectra using a deep learning-based approach[J]. Frontiers in Microbiology, 2022, 13: 821 233. [16] PAPAGIANNOPOULOU C, PARCHEN R, RUBBENS P, WAEGEMAN W. Fast pathogen identification using single-cell matrix-assisted laser desorption/ionization-aerosol time-of-flight mass spectrometry data and deep learning methods[J]. Analytical Chemistry, 2020, 92(11): 7 523-7 531. [17] BEHRMANN J, ETMANN C, BOSKAMP T, CASADONTE R, KRIEGSMANN J, MAAΒ P. Deep learning for tumor classification in imaging mass spectrometry[J]. Bioinformatics, 2018, 34(7): 1 215-1 223. [18] BOSKAMP T, LACHMUND D, OETJEN J, CORDERO HERNANDEZ Y, TREDE D, MAASS P, CASADONTE R, KRIEGSMANN J, WARTH A, DIENEMANN H, WEICHERT W, KRIEGSMANN M. A new classification method for MALDI imaging mass spectrometry data acquired on formalin-fixed paraffin-embedded tissue samples[J]. Biochimica et Biophysica Acta (BBA)-Proteins and Proteomics, 2017, 1 865(7): 916-926. [19] GU J, WANG Z, KUEN J, MA L, SHAHROUDY A, SHUAI B, LIU T, WANG X, WANG G, CAI J, CHEN T. Recent advances in convolutional neural networks[J]. Pattern Recognition, 2018, 77: 354 -377 . doi: 10.1016/j.patcog.2017.10.013[20] ZHANG C Y, PHILIP CHEN C L, CHEN D, KIN TEK N. MapReduce based distributed learning algorithm for restricted Boltzmann machine[J]. Neurocomputing, 2016, 198: 4 -11 . doi: 10.1016/j.neucom.2015.09.129[21] MORTIER T, WIEME A D, VANDAMME P, WAEGEMAN W. Bacterial species identification using MALDI-TOF mass spectrometry and machine learning techniques: a large-scale benchmarking study[J]. Computational and Structural Biotechnology Journal, 2021, 19: 6 157-6 168. [22] WANG G, RUSER H, SCHADE J, PASSIG J, ADAM T, DOLLINGER G, ZIMMERMANN R. 1D-CNN network based real-time aerosol particle classification with single-particle mass spectrometry[J]. IEEE Sensors Letters, 2023, 7(11): 6 007 904. [23] HU F, ZHOU M, YAN P, LI D, LAI W, BIAN K, DAI R. Identification of mine water inrush using laser-induced fluorescence spectroscopy combined with one-dimensional convolutional neural network[J]. RSC Advances, 2019, 9(14): 7 673-7 679. [24] WANG H Y, CHUNG C R, CHEN C J, LU K P, TSENG Y J, CHANG T H, WU M H, HUANG W T, LIN T W, LIU T P, LEE T Y, HORNG J T, LU J J. Clinically applicable system for rapidly predicting Enterococcus faecium susceptibility to vancomycin[J]. Microbiology Spectrum, 2021, 9(3): e0091321. doi: 10.1128/Spectrum.00913-21 [25] LI X, LI L, ZHUO Z, ZHANG G, DU X B, LI X, HUANG Z, ZHOU Z, CHENG Z. Bioaerosol identification by wide particle size range single particle mass spectrometry[J]. Atmosphere, 2023, 14(6): 1 017. [26] PLOMP M, LEIGHTON T J, WHEELER K E, MALKIN A J. The high-resolution architecture and structural dynamics of bacillus spores[J]. Biophysical Journal, 2005, 88(1): 603 -608 . doi: 10.1529/biophysj.104.049312[27] WANG H, WANG Z, DU M, YANG F, ZHANG Z, DING S, MARDZIEL P, HU X. Score-CAM: score-weighted visual explanations for convolutional neural networks[C]//2020 IEEE/CVF Conference on Computer Vision and Pattern Recognition Workshops (CVPRW). June 14-19, 2020. Seattle, WA, USA. IEEE, 2020: 111-119. [28] 牛向凤, 张晓清, 于馨恒, 苏营雪, 陈磊, 张卫文. 液相质谱联用的代谢组方法优化及其在蓝细菌分析中的应用[J]. 生物工程学报, 2015, 31(4): 577 -590 . NIU Xiangfeng, ZHANG Xiaoqing, YU Xinheng, SU Yingxue, CHEN Lei, ZHANG Weiwen. Optimization and application of targeted LC-MS metabolomic analyses in photosynthetic cyanobacteria[J]. Chinese Journal of Biotechnology, 2015, 31(4):577 -590 (in Chinese).[29] CZERWIENIEC G A, RUSSELL S C, TOBIAS H J, PITESKY M E, FERGENSON D P, STEELE P, SRIVASTAVA A, HORN J M, FRANK M, GARD E E, LEBRILLA C B. Stable isotope labeling of entire Bacillus atrophaeus spores and vegetative cells using bioaerosol mass spectrometry[J]. Analytical Chemistry, 2005, 77(4): 1 081-1 087. [30] 曾真, 喻佳俊, 刘平, 黄福桂, 陈颖, 黄正旭, 高伟, 李梅, 周振, 李磊. 利用单颗粒气溶胶质谱仪分析细菌气溶胶颗粒[J]. 分析化学, 2019, 47(9): 1 344-1 351. ZENG Zhen, YU Jiajun, LIU Ping, HUANG Fugui, CHEN Ying, HUANG Zhengxu, GAO Wei, LI Mei, ZHOU Zhen, LI Lei. Investigation of characters of bioaerosols on single particle aerosol mass spectrometer[J]. Chinese Journal of Analytical Chemistry, 2019, 47(9): 1 344-1 351(in Chinese). [31] DRIKS A. Bacillus subtilis spore coat[J]. Microbiology and Molecular Biology Reviews, 1999, 63(1): 1 -20 . doi: 10.1128/MMBR.63.1.1-20.1999[32] MOIR A. Spore germination receptors-a new paradigm[J]. Trends in Microbiology, 2023, 31(8): 767 -768 . doi: 10.1016/j.tim.2023.05.012[33] WANG H, NAIDU R, MICHAEL J, SNIGDHA KUNDU S. SS-CAM: smoothed score-CAM for sharper visual feature localization[J]. ArXiv e-Prints, doi: org/10.48550/arXiv.2006.14255. -

 首页
首页 登录
登录 注册
注册


 下载:
下载: