-
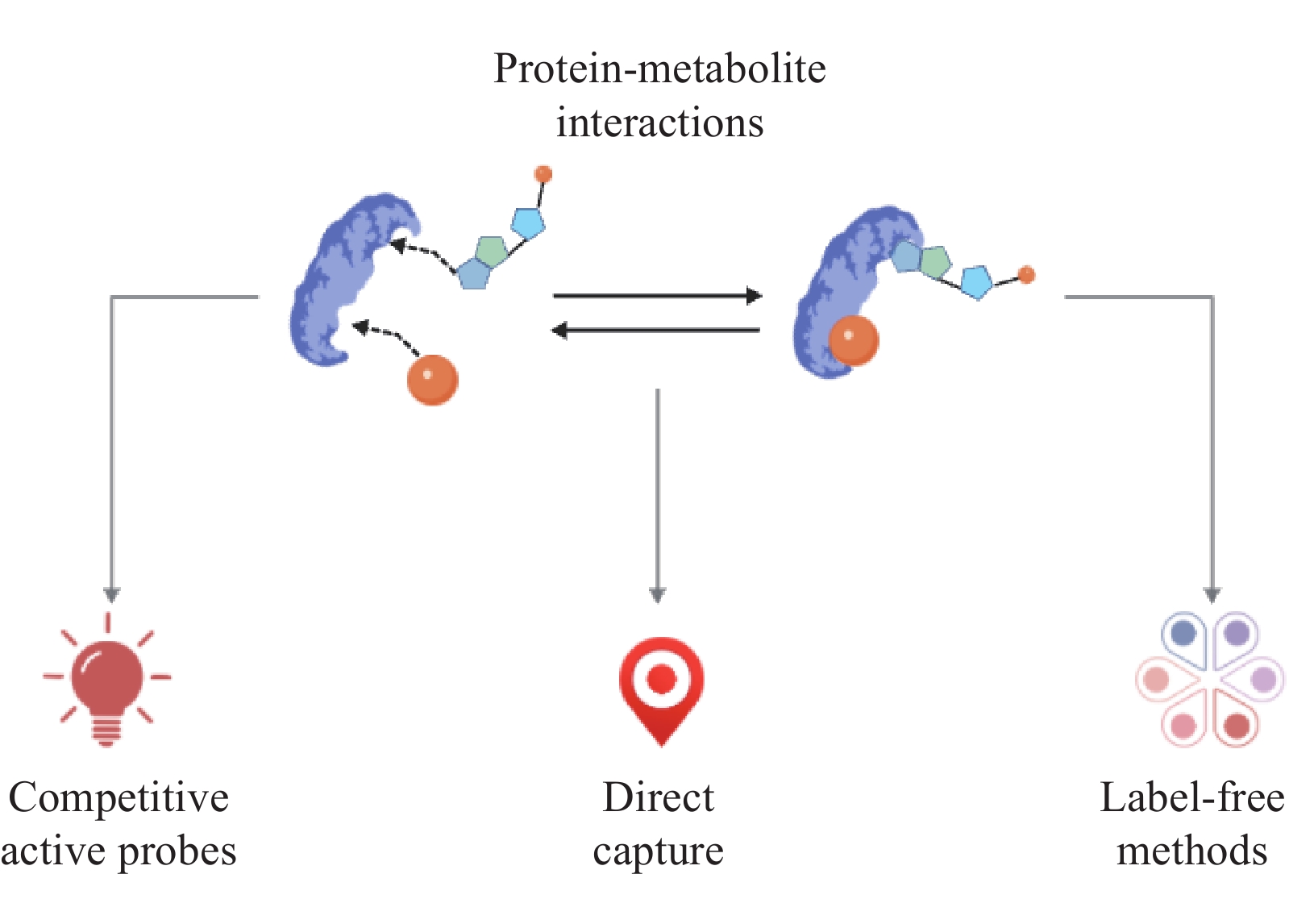
在人体中,代谢物除作为新陈代谢转化的中间体外,还充当信号分子直接或间接参与体内各种适应性反应。小分子代谢物与蛋白质之间可通过共价或非共价结合产生相互作用,从而调控蛋白质的结构、活性和功能,目前已报道了代谢物与蛋白质之间存在不同类型的相互作用[1]。蛋白质-代谢物相互作用在调节蛋白质功能和协调细胞反应中具有重要作用[2-4],例如,代谢酶、转录因子、转运蛋白和膜受体的活性均可由蛋白质-代谢物相互作用介导。因此,研究蛋白质-代谢物相互作用可以更深入地解析细胞代谢、遗传/表观遗传调控、环境感知和信号转导等多种生物过程。
蛋白质-代谢物相互作用主要分为共价修饰和非共价修饰两大类。其中,非共价修饰是通过弱相互作用,如氢键、离子键、疏水相互作用等实现代谢物与蛋白质的结合[5-6],通常是动态的、可逆的,例如,代谢物与酶的结合、激素和受体之间的结合,此类蛋白质-代谢物相互作用能够灵活地响应细胞内外环境的变化;共价修饰是指代谢物通过形成共价键修饰蛋白质的特定位点,进而改变蛋白质的功能[7],很多翻译后修饰属于这一类型,例如,乙酰化[8]、磷酸化[9]和羰基化[10]等。
近年来,基于质谱的蛋白质组学技术发展迅速,推动了蛋白质-代谢物相互作用的系统研究与分析。随着质谱[11]、化学探针[12-14]、无标记检测[15-17]等技术的成熟,研究者能够更高效、更精准地捕捉代谢物与蛋白质之间的相互作用。例如,化学探针技术通过模拟代谢物的结构特征,特异性地捕获与代谢物结合的蛋白质;质谱技术通过提供更大规模、更高通量且更快速的分析手段,助力深入研究蛋白质-代谢物相互作用。以上技术的结合不仅提高了蛋白质-代谢物相互作用的检测灵敏度和特异性,而且有助于在更接近生理条件下探讨相互作用的动态过程[16,18-19]。
随着蛋白质组学技术的发展,蛋白质-代谢物相互作用的研究方法逐渐多样化,这为深入理解代谢调控机制提供了新思路。本文将介绍近年来蛋白质组学技术在探究蛋白质-代谢物相互作用中的进展,并讨论未来方法开发的前景。
-
近年来,利用与目标代谢物“匹配”的竞争性活性探针进行蛋白质-代谢物相互作用的研究策略受到国内外学者的广泛关注。该方法的核心是基于目标代谢物的结构特征,设计带有生物正交等标签的化学探针,通过与靶标蛋白的活性位点或结合位点竞争,特异性捕获与代谢物相互作用的蛋白质,实现对蛋白质-代谢物相互作用的精准解析,示于图1。
研究[25]表明,促炎刺激下的巨噬细胞会发生代谢重编程,影响免疫响应过程。同时,巨噬细胞在炎症刺激后会抑制三羧酸循环水平,其中部分三羧酸循环的中间代谢物参与免疫调控进程,如富马酸[3,26]、衣康酸[27-28]、甲基乙二醛[29]。近期,竞争性活性探针技术被用于代谢物富马酸敏感的新型蛋白质半胱氨酸研究[24]。研究者设计了一种带有生物正交基团的富马酸炔烃探针,通过竞争性的同位素活性蛋白质分析(isoTOP-ABPP)平台,研究富马酸剂量依赖的半胱氨酸变化,共定量分析1 170个半胱氨酸残基,绘制了富马酸的化学蛋白质组图,发现了遗传性癌症综合征遗传性平滑肌瘤病和肾细胞癌中对富马酸水合酶突变敏感的新半胱氨酸活性位点。同时,该研究还揭示了富马酸调节的半胱氨酸与半胱氨酸反应活性呈负相关,这为富马酸调控机制研究提供了新思路。与此同时,北京大学王初团队[23]发展了一种生物正交基团衍生的新型衣康酸炔基探针,可以有效进入细胞中并实现活细胞内衣康酸修饰蛋白的直接标记。该类探针具有与衣康酸相当的半胱氨酸反应活性,可以被衣康酸有效地竞争,同时还表现出与衣康酸类似的抗炎活性,能够抑制糖酵解以及激活Nrf2通路等生物学效应。利用该探针共鉴定到1 926个衣康酸修饰蛋白,覆盖了此前报道的大部分衣康酸修饰蛋白。另外,研究者采用具有时间分辨率的化学蛋白质组学策略,进一步从上述衣康酸修饰蛋白中筛选出199个具有高反应活性的修饰蛋白,包括此前已知的衣康酸功能性底物蛋白ALDOA和KEAP1等,为衣康酸调控炎症等免疫代谢研究提供了新思路。2023年,芝加哥大学的Moellering教授团队[21]使用半胱氨酸特异性探针碘乙酰胺炔烃与甲基乙二醛修饰竞争标记,从全蛋白层次检测半胱氨酸残基甲基乙二醛修饰,在细胞裂解物水平检测了甲基乙二醛在半胱氨酸上的修饰水平,筛选了86个强竞争性半胱氨酸修饰位点,这些修饰位点大多与代谢酶有关。在活细胞水平,该团队采用同样的标记方法进行了“体外”和“原位”实验,发现在HeLa细胞中乙酰转移酶ACAT1上的半胱氨酸显示了多个位点的高竞争性,且这些高竞争性位点都处于酶活中心附近,为其反应活性的功能调控提供了新思路。
酰基辅酶A是脂质生物合成的基本细胞组成部分,也是通过三羧酸循环产生能量的中间体,以及可逆蛋白质乙酰化的必需前体。上述生物学功能的发挥都依赖于酰基辅酶A/蛋白质的相互作用,但是,酰基辅酶A与蛋白质的作用范围和选择性尚未清晰。美国国立卫生研究院国家癌症研究所Meier团队[22]应用树脂固定的辅酶A类似物作为亲和基质,将蛋白质组与酰基辅酶A代谢物预孵育,直接从生物样本中竞争性捕获利用酰基辅酶A的催化酶,并评估它们与目标酶的相对结合亲和力。在此基础上,还开发了化学蛋白质组学平台CATNIP(CoA/AcetylTraNsferase Interaction Profiling),用于分析酰基辅酶A/蛋白质相互作用[30]。通过对CATNIP数据的系统分析,揭示人类蛋白质组中存在不同的酰基辅酶A/蛋白质相互作用特征;通过比较CATNIP和已发表的赖氨酸酰化数据集,进行酰基辅酶A的酶促和非酶促调控功能的注释。同时,该研究说明了如何整合化学蛋白质组学和系统生物学来了解酰基辅酶A代谢在生物学和疾病中的作用。
此外,北京大学王初团队[31]开发了一种有效的无标记和位点特异性化学蛋白质组学策略,该方法将数据非依赖性采集(DIA)质谱与基于活性的蛋白质分析(ABPP)相结合,在分析功能性半胱氨酸组方面展现出更全面的数据覆盖率和更高的定量准确性,包括剂量依赖性定量半胱氨酸对反应性代谢物的敏感性、使用共价片段库筛选可配体的半胱氨酸,以及分析昼夜节律周期中的半胱氨酸组波动。但是,DIA-ABPP技术检测耗时相对较长,未来仍需进一步优化。
上述列举的新型代谢物竞争性活性探针具有较高的反应活性,拓宽了对代谢物调控网络的认识范围。对于此类探针所鉴定到的相互作用蛋白质,鉴定的准确性与这些蛋白质的功能还需结合结构生物学、生物化学等方法进一步探究。
-
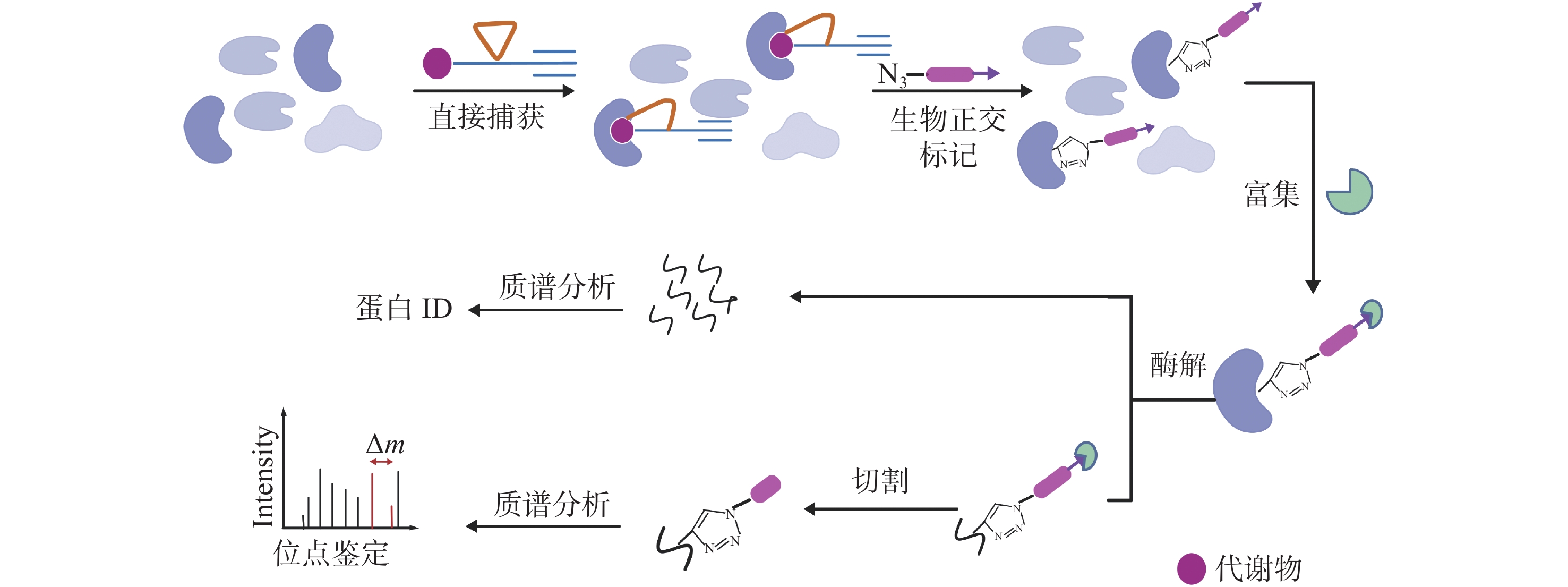
基于化学反应的直接捕获方法的主要原理是利用代谢物与蛋白质之间的共价反应或非共价反应,通过捕获化学反应后的稳定复合物,再结合质谱等分析手段,准确识别与代谢物结合的靶标蛋白。该方法能够在复杂的生物样本中特异地、灵敏地捕捉蛋白质-代谢物相互作用,尤其适用于难以用传统技术检测的瞬时和弱相互作用,其流程图示于图2。
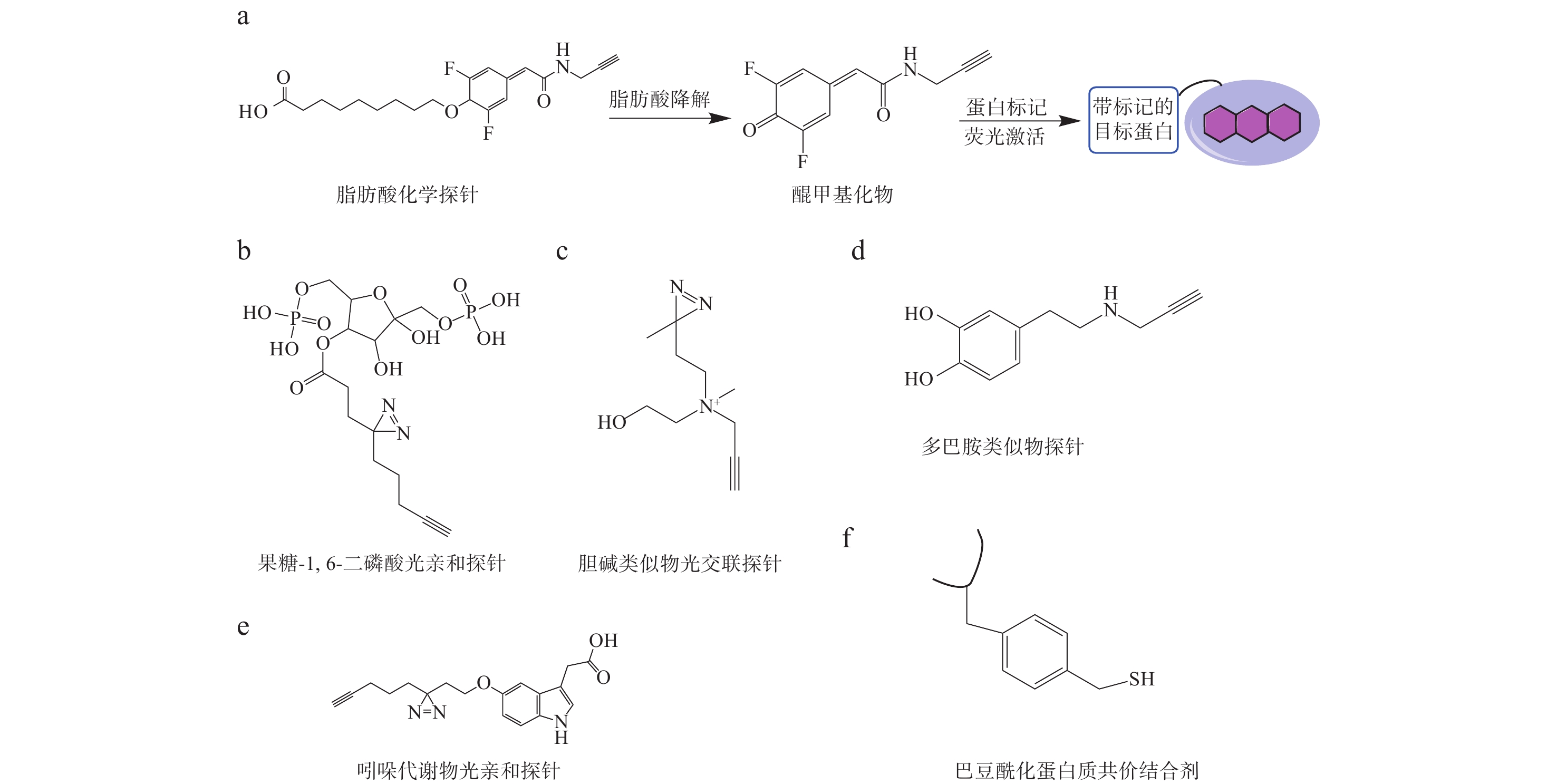
脂质代谢物与蛋白质的相互作用是基于化学反应的直接捕获方法研究的热点。日本九州大学Ojida教授团队[32]于2023年报道了一种脂肪酸探针,可以检测活细胞中脂肪酸β-氧化的活性。该探针是具有简单分子结构的醌甲基,在脂肪酸单元被脂肪酸β-氧化酶切断,释放活性醌甲基,随后被细胞内蛋白质共价捕获,并与其烷基反应柄上的荧光团进行生物正交反应,可以进行不同类型的成像可视化分析。通过化学蛋白质组学技术,在细胞中鉴定出883个醌甲基反应性蛋白质,其中包括数种参与脂肪酸β-氧化途径的线粒体酶,示于图3。日本名古屋大学Taki团队[33]报道了另一种新型的荧光脂肪酸探针,该探针带有一种极性环境敏感的荧光团3a-azapyren-4-one,会根据运输到的细胞器微环境极性改变其吸收和发射特性。基于这种荧光脂肪酸探针的环境敏感性,可以用不同颜色显示其在非极性脂滴、中等极性膜和极性区域中的分布。作者观测了脂肪酸代谢物在不同条件下的细胞内空间分布,并验证了在营养匮乏的肝细胞中,脂滴中三酰甘油的降解主要通过脂肪分解而不是脂肪吞噬。2023年,北京大学陈鹏和季雄团队[19]联合报道了一种单点分辨多组学(SiTomics)策略,包含修饰位点解析(Site-profiling)、位点特异性组蛋白标记的联合蛋白质组学(Site-link)和位点特异性基因组(Site-seq)分析,可用于研究活细胞生理染色质上的组蛋白酰化和相互作用组。通过合成一系列光亲和非天然氨基酸,利用基因密码子拓展技术将其定点引入到特定位点,解析短链脂肪酸处理后位点特异性组蛋白修饰的动态变化。通过SiTomics分析HEK293T细胞在短链脂肪酸刺激下的组蛋白酰化,验证了先前已知的修饰位点,还发现了K36、K37、K64、K116新修饰位点。此外,该研究报道了在H3K56处短链脂肪酸衍生代谢物刺激后的不同酰化,揭示了巴豆酰化和β-羟基丁酰化相互作用的蛋白质组与基因组之间存在信息关联。
同时,研究者们越来越关注三羧酸循环的中间代谢物。例如,腺嘌呤核苷三磷酸(ATP)在细胞能量代谢和信号传导中扮演着重要角色,捕获这些代谢物与蛋白质的相互作用,对于揭示能量代谢调控机制至关重要。北京大学李毓龙实验室[34]开发了一种可用于实时测量细胞外的ATP探针,该探针是基于基因编码的G蛋白偶联受体活化,具有高灵敏度、高选择性和高时空分辨率。与此同时,韩国研究团队[35]报道了通过可逆结合选择性检测核ATP的小分子探针,对各种生物物种具有出色的选择性。使用该探针可以观察到癌细胞系和肿瘤组织中的核ATP水平与正常细胞系和组织的显著差异,在研究核ATP相关生物学方面具有巨大潜力。另一方面,与细胞内源性糖代谢产物果糖-1,6-二磷酸(FBP)相互作用的蛋白质组也有新进展。中国医学科学院药物研究所耿轶群课题组[36]设计了一种光亲和探针,该探针保留了FBP的糖磷酸骨架,引入双吖丙啶和炔烃,通过紫外线诱导的交联与靶蛋白由非共价转为共价结合。作者通过定量化学蛋白质组学策略鉴定了52个具有高置信度的FBP互作蛋白,并验证了其中6种新鉴定的FBP互作蛋白。结果表明,这些蛋白均能被成功捕获,且可被过量的FBP竞争性抑制。该研究还解析了由FBP及其靶蛋白线粒体代谢酶—乙醛脱氢酶 2(ALDH2)介导的糖代谢信号感知机制,发现线粒体代谢酶ALDH2是FBP的关键靶标。
近年来,胆碱类和多巴胺类代谢物作用蛋白的捕获技术取得了重要进展,示于图3。2022年,有研究[37]报道了一种光交联胆碱类似物探针,通过化学蛋白质组学方法捕获到902种蛋白质,其中674种是高富集程度的高可信蛋白质,且有部分蛋白质是已知的抗癌药物靶标。同时,该研究还证明了磷酸胆碱抑制其中一种已鉴定的蛋白质抗癌靶标p32与其内源性配体。美国明尼苏达大学团队[38]开发了一种含有生物正交官能团的多巴胺探针,该探针是一种新型多巴胺类似物,含有炔基官能团,具有与多巴胺相似的反应活性,可以在全蛋白质组层次鉴定并富集多巴胺修饰的蛋白。作者对神经元SH-SY5Y细胞中多巴胺修饰的蛋白进行鉴定,发现其广泛存在于细胞中,并有38种蛋白质显著富集,包括参与内质网应激反应的PDIA3。同时还发现,当PDIA3蛋白上存在多巴胺修饰时,其功能被显著抑制,表明失调的细胞多巴胺代谢加剧了内质网应激反应,进而诱导神经退行性疾病的发生。
微生物群产生多种代谢物来调节宿主的生理和疾病,但它们的蛋白质靶点和作用机制尚未完全阐明。Hang教授团队[39]设计了一种含有双吖丙啶交联基团的光亲和探针(x-alk-IAA),利用化学蛋白质组学方法探索微生物群衍生的吲哚代谢物。在HT-29细胞中鉴定到孤儿受体GPCRs(GPRC5A、GPR107和GPR108)与吲哚-3-乙酸(IAA)相互作用,同时确认了许多潜在的吲哚代谢物相互作用蛋白,包括代谢酶、转运蛋白和免疫传感器,但只有芳香单胺能够结合孤儿受体GPRC5A并刺激β-arrestin募集。另外,研究者通过微生物功能分析揭示了产生GPRC5A激动剂的特定细菌种类和酶,发现7-氟色胺是GPRC5A的有效激动剂。
针对近年来发现的新型赖氨酸翻译后修饰,如巴豆酰化、2-羟基异丁酰化、β-羟基丁酰化和肉豆蔻酰化,相应化学捕获技术也有新进展。CobB是一种烟酰胺腺嘌呤二核苷酸(NAD+)依赖性sirtuin家族的细菌蛋白脱乙酰酶,南开大学团队[40]基于邻近诱导交联将CobB在巴豆酰化相互作用位点用含硫醇的非天然氨基酸进行改造,随后能够以NAD+依赖性方式与巴豆酰化位点发生反应,该共价结合剂具有高选择性和优异的效率,可以弥补巴豆酰化抗体的不足。与此同时,天津医科大学张锴团队[41]利用CobB发展了一种新型自组装多价光交联肽探针,证明CobB可以在体内和体外催化赖氨酸脱-2-羟基异丁酰化,同时使用定量蛋白质组学方法鉴定出99种CobB脱2-羟基异丁酰化作用靶向的内源性底物。此外,最近研究[42]提出了一种新型多价光亲和探针,用于捕获与赖氨酸β-羟基丁酰化反应相结合的蛋白质,通过设计包含模拟组蛋白β-羟基丁酰化修饰的肽段和连接光交联剂的PEG链的β-羟基丁酰化探针,利用交联剂的间隔基团和标记,成功在细胞裂解液中发现了43个与探针相互作用的蛋白质,其中包含多个已知的H3K4me3阅读器。同时,还首次将染色质结合蛋白ENL确定为这一新标记的潜在靶标。
-
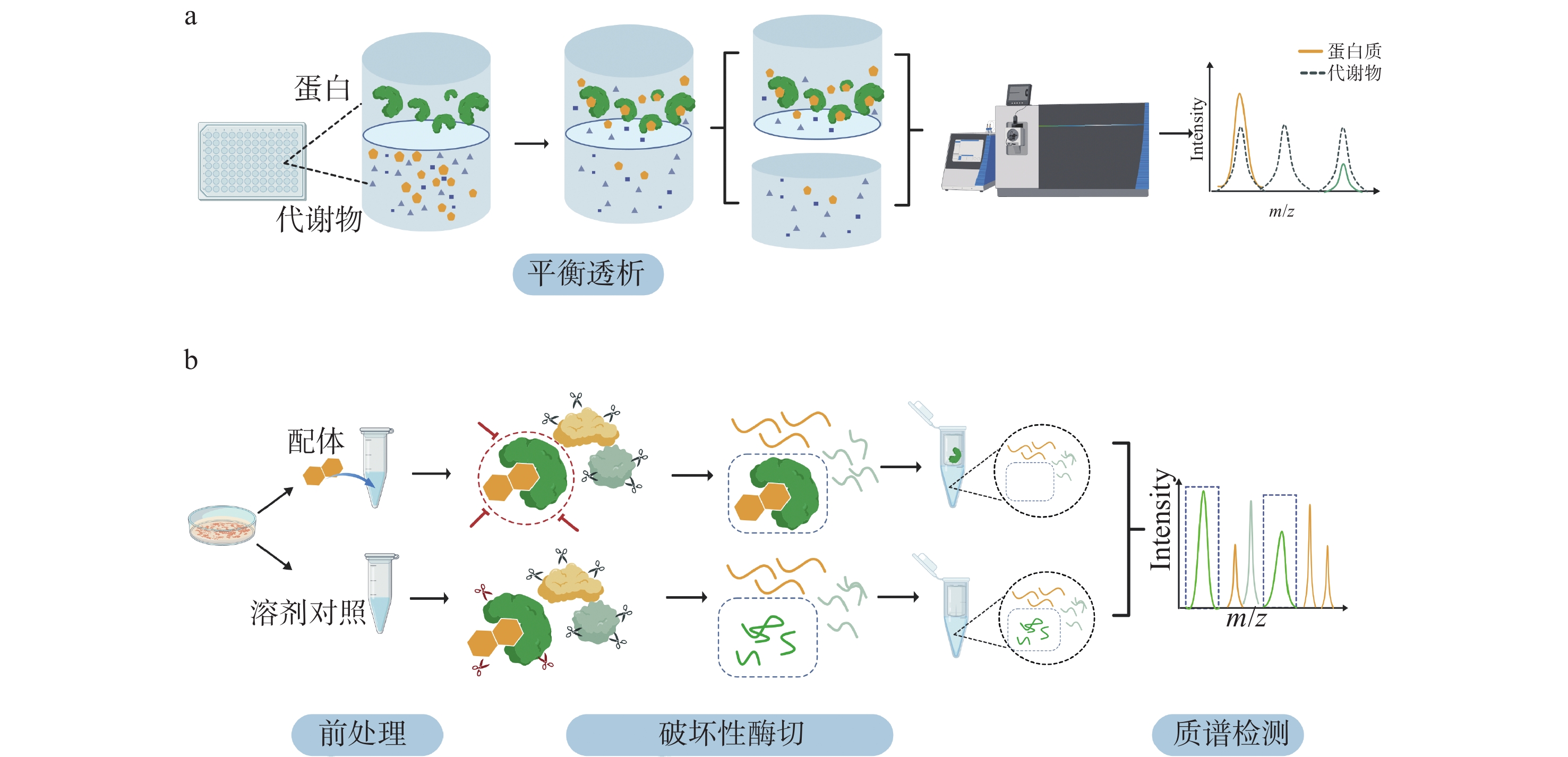
蛋白质-代谢物相互作用研究已从传统的标记方法逐渐转向免修饰标记策略,避免了对代谢物或蛋白质进行化学修饰或引入外源标签,有助于更真实地反映生物体系中的生理状态,在近5年中得到广泛应用,示于图4。
Rutter研究小组[43]通过优化前期开发的结合平衡透析的质谱法(MIDAS),全面系统地发现了蛋白质-代谢物相互作用,包括低亲和力和高亲和力相互作用。该方法的核心为透析法,利用半透性透析膜允许代谢物扩散并阻断蛋白质扩散,将纯化的蛋白质与确定的代谢物库分离。当系统达到相对平衡后,蛋白质室和代谢物室中的游离代谢物(即不相互作用的代谢物)浓度相似。此时,在包含与代谢物相互作用蛋白质的一侧,代谢物总浓度(包括游离的和与蛋白质结合的)增加。最后,使用液相色谱或气相色谱-质谱联用技术定量分析含蛋白质室和对照室中不同代谢物的相对浓度。利用该技术对来自人类碳水化合物代谢的33种酶进行分析,确定了830种蛋白质-代谢物相互作用,包括已知的调节剂、底物和产物以及之前未报道的相互作用。
近期,本课题组开发了一种“以肽段为中心的蛋白局部稳定性探测方法(peptide-centric local stability assay, PELSA)”[44-45]。该方法使用深度酶切将未经变性的蛋白质直接酶切为小肽段,富集肽段后比较加药组和对照组小肽段产生的差异来确定药物的靶蛋白和结合区域。在叶酸、亮氨酸、琥珀酸、富马酸、乳酸、α-酮戊二酸和肿瘤代谢物R2HG等多种代谢物相互作用蛋白的鉴定中,PELSA相较于常用的热蛋白质组技术[46]和mTSA技术[47]展现出更高的鉴定灵敏度,还能够提供丰富的代谢物与蛋白作用区域信息,同时可鉴定到多个可信的未知作用蛋白。此外,PELSA法在药物、金属离子、翻译后修饰肽段及抗体等多种配体的结合蛋白鉴定时,展现出高灵敏的靶蛋白鉴定性能和精准的结合区域定位能力,证明了该方法可以作为一个通用的分析平台研究各种配体与蛋白的相互作用,在代谢物、药物及污染物的识别及作用机制解析等领域具有广泛的应用价值。
虽然质谱等技术在免修饰标记策略中显示出巨大的潜力,但仍存在一定的局限性。例如,MIDAS平台简单易操作,但需要合成大量的纯化蛋白质;PELSA方法仅适用于细胞裂解液样品,而不适用于活细胞样品。因此,从适用性、分析精度、检测灵敏度及数据解读等方面如何优化这些技术,使其能够更加适应动态复杂的细胞环境,是未来研究的重要方向。
-
蛋白质-代谢物相互作用研究是揭示细胞代谢调控机制和疾病发生发展机理的关键途径。本文综述了近5年报道的基于目标代谢物“匹配”的竞争性活性探针、基于化学反应的直接捕获方法和无标记策略,研究者们通过不断创新技术手段,在代谢物与蛋白质相互作用的识别和解析方面取得了一系列突破。这些研究不仅丰富了蛋白质-代谢物相互作用的内涵,还揭示了其在能量代谢、脂质代谢、氧化还原调控等多条代谢通路中的重要作用,为深入探究疾病的分子机制以及开发相关的靶向药物提供了有力支持。竞争性探针能够精确定位特定的代谢物-蛋白质相互作用,而直接捕获方法具有强大的化学特异性。相比之下,免修饰标记策略提供了生物兼容性更优的系统用于相互作用研究。
尽管蛋白质-代谢物相互作用研究取得了显著进展,但仍存在一些关键科学问题亟待解决。首先,目前许多技术手段主要集中于强相互作用的捕获,如何有效地检测瞬时或弱相互作用则面临挑战。此外,代谢物种类繁多、化学性质各异,针对不同代谢物特异性捕获和解析技术仍需进一步发展。同时,需要创建系统的、标准化的报告和整合实验的程序。如本文所述,检测蛋白质-代谢物相互作用的方法很多,但要求和功能差别很大,系统地比较和整合多种方法的数据对于准确使用相互作用组学实验数据至关重要,这一点已在蛋白质-蛋白质相互作用组学领域得到证明[48]。另一方面,大多数高通量方法可以检测到相互作用分子之间的物理关联,但缺乏描述生物学作用的方法[49],这对于相互作用组学和整个生物学领域来说更为根本。
现有研究大多集中于特定代谢物与蛋白质的相互作用网络,全面解析整个代谢网络中不同代谢物与蛋白质的交互关系是一个尚未解决的难题。多组学数据的整合分析将为蛋白质-代谢物相互作用研究提供更全面的视角,通过整合转录组、蛋白质组、代谢组等多维数据,能够更全面地解析代谢物在细胞中的功能及其对蛋白质网络的调控作用。例如,SiTomics策略可广泛应用于除酰化以及组蛋白外的蛋白质的多组学分析和功能分析[19]。其次,随着单细胞技术的发展[50],研究蛋白质-代谢物相互作用的空间和时间分辨率有望进一步提高。这将帮助研究者揭示代谢物在特定细胞类型或亚细胞结构中的动态变化及其与蛋白质的特异性作用。此外,人工智能和机器学习技术在大数据分析中的应用将显著加速蛋白质-代谢物相互作用网络的构建和预测,为靶向药物的开发提供更精准的理论依据。
总的来说,蛋白质-代谢物相互作用的研究正在迅猛发展,基于化学生物学和质谱方法的进步会推动鉴定和识别新型蛋白质-代谢物相互作用。未来结合多学科技术,有望在基础研究和应用研究中取得更加丰硕的成果。
用于解析蛋白质-代谢物相互作用的蛋白质组学技术
Proteomic Techniques for Profiling of Protein-Metabolite Interactions
-
摘要: 代谢物可通过共价或非共价方式与多种蛋白质相互作用,进而调控蛋白质功能。然而,许多此类相互作用非常短暂且亲和力低,并且存在高度动态性,其相互作用网络的复杂性导致蛋白质-代谢物相互作用的系统性鉴定面临极大挑战。近年来,基于质谱的蛋白质组学技术发展迅速,可无偏倚地系统描绘蛋白质-代谢物相互作用。本文综述了近5年用于解析蛋白质-代谢物相互作用的蛋白质组学技术进展,包括与目标代谢物“匹配”的竞争性活性探针、基于化学反应的直接捕获方法等,并深入讨论这些方法的优势、局限性及应用场景,以期为蛋白质-代谢物相互作用的研究提供参考。
-
关键词:
- 蛋白质-代谢物相互作用 /
- 翻译后修饰 /
- 非共价结合 /
- 蛋白质组学
Abstract: Metabolites are small-molecule intermediates in metabolic pathways, which can interact with proteins and regulate protein functions in covalent or non-covalent ways. The interactions between metabolites and proteins play a crucial role in regulating cellular metabolism, influencing various biological processes, and contributing to disease development. Understanding these interactions is vital for elucidating metabolic pathways and designing therapeutic strategies for metabolic disorders and other diseases. However, studying metabolite-protein interactions presents several challenges due to the transient and dynamic nature of these interactions, as well as the vast diversity of metabolites and proteins in biological systems. Despite these obstacles, significant advances have been made in the past decade, particularly with the development of proteomics techniques, which have provided powerful tools to investigate these interactions with unprecedented depth and resolution. This review summarized the progress made in proteomics techniques for resolving protein-metabolite interactions over the past five years, including competitive active probes matched to target metabolites, direct capture methods based on chemical reactions, and label-free strategies for detecting endogenous protein-metabolite interactions. It further discussed the advantages, limitations, and application scenarios of these methods, aiming to provide a reference for studying protein-metabolite interactions. The competitive profiling strategy can covalently capture different types of metabolite-induced modifications with high selectivity and sensitivity. Significantly, this method has also been applied to profile modifications by reactive natural products or covalent drugs. On the other hand, direct capture methods also use chemoselective probes, but these probes are mostly applied in cell lysates due to their cell penetration and toxicity. Label-free strategies are able to detect non-covalent protein-metabolite interactions and record protein structural information in vitro, providing a more native, unmodified system for studying interactions of metabolites with proteins. However, extremely high sample reproducibility and extensive proteomic runs may not be feasible for regular labs. As a result, it is still challenging to uncover the complex nature of protein-metabolite interactions by label-free strategie. The development in analytical and bioinformatic approaches, as well as the advances in omics applications, offer new opportunities for this field. It is promising that a global landscape of the interaction proteins for the metabolites will be depicted in the future. This review provides insights into understanding the development of proteomic techniques for profiling protein-metabolite interactions. -
-
-
[1] CHUBUKOV V, GEROSA L, KOCHANOWSKI K, SAUER U. Coordination of microbial metabolism[J]. Nature Reviews Microbiology, 2014, 12(5): 327 -340 . doi: 10.1038/nrmicro3238[2] FIGLIA G, WILLNOW P, TELEMAN A A. Metabolites regulate cell signaling and growth via covalent modification of proteins[J]. Developmental Cell, 2020, 54(2): 156 -170 .[3] DISKIN C, RYAN T A J, O’NEILL L A J. Modification of proteins by metabolites in immunity[J]. Immunity, 2021, 54(1): 19 -31 .[4] ISHIHAMA A. Prokaryotic genome regulation: multifactor promoters, multitarget regulators and hierarchic networks[J]. FEMS Microbiology Reviews, 2010, 34(5): 628 -645 .[5] DU X, LI Y, XIA Y L, AI S M, LIANG J, SANG P, JI X L, LIU S Q. Insights into protein-ligand interactions: mechanisms, models, and methods[J]. International Journal of Molecular Sciences, 2016, 17(2): 144 .[6] GEROSA L, SAUER U. Regulation and control of metabolic fluxes in microbes[J]. Current Opinion in Biotechnology, 2011, 22(4): 566 -575 .[7] HARMEL R, FIEDLER D. Features and regulation of non-enzymatic post-translational modifications[J]. Nature Chemical Biology, 2018, 14(3): 244 -252 .[8] FANG Z, QIN H, MAO J, WANG Z, ZHANG N, WANG Y, LIU L, NIE Y, DONG M, YE M. Glyco-decipher enables glycan database-independent peptide matching and in-depth characterization of site-specific N-glycosylation[J]. Nature Communications, 2022, 13(1): 1 900. [9] ZHANG H, OU J, YAO Y, WANG H, LIU Z, WEI Y, YE M. Facile preparation of titanium(IV)-immobilized hierarchically porous hybrid monoliths[J]. Analytical Chemistry, 2017, 89(8): 4 655-4 662. [10] HAUCK A K, HUANG Y, HERTZEL A V, BERNLOHR D A. Adipose oxidative stress and protein carbonylation[J]. Journal of Biological Chemistry, 2019, 294(4): 1 083-1 088. [11] SCHLOSSAREK D, ZHANG Y, SOKOLOWSKA E M, FERNIE A R, LUZAROWSKI M, SKIRYCZ A. Don’t let go: co-fractionation mass spectrometry for untargeted mapping of protein-metabolite interactomes[J]. The Plant Journal, 2023, 113(5): 904 -914 .[12] HABERKANT P, STEIN F, HÖGLINGER D, GERL M J, BRÜGGER B, van VELDHOVEN P P, KRIJGSVELD J, GAVIN A C, SCHULTZ C. Bifunctional sphingosine for cell-based analysis of protein-sphingolipid interactions[J]. ACS Chemical Biology, 2016, 11(1): 222 -230 .[13] NIPHAKIS M J, LUM K M, COGNETTA A B, CORREIA B E, ICHU T A, OLUCHA J, BROWN S J, KUNDU S, PISCITELLI F, ROSEN H, CRAVATT B F. A global map of lipid-binding proteins and their ligandability in cells[J]. Cell, 2015, 161(7): 1 668-1 680. [14] KALLEMEIJN W W, LUEG G A, FARONATO M, HADAVIZADEH K, GROCIN A G, SONG O R, HOWELL M, CALADO D P, TATE E W. Validation and invalidation of chemical probes for the human myristoyltransferases[J]. Cell Chemical Biology, 2019, 26(6): 892 .[15] PIAZZA I, KOCHANOWSKI K, CAPPELLETTI V, FUHRER T, NOOR E, SAUER U, PICOTTI P. A map of protein-metabolite interactions reveals principles of chemical communication[J]. Cell, 2018, 172(1/2): 358 -372 .[16] SCHOPPER S, KAHRAMAN A, LEUENBERGER P, FENG Y, PIAZZA I, MÜLLER O, BOERSEMA P J, PICOTTI P. Measuring protein structural changes on a proteome-wide scale using limited proteolysis-coupled mass spectrometry[J]. Nature Protocols, 2017, 12(11): 2 391-2 410. [17] SRIDHARAN S, KURZAWA N, WERNER T, GÜNTHNER I, HELM D, HUBER W, BANTSCHEFF M, SAVITSKI M M. Proteome-wide solubility and thermal stability profiling reveals distinct regulatory roles for ATP[J]. Nature Communications, 2019, 10(1): 1 155. [18] WRIGHT M H, SIEBER S A. Chemical proteomics approaches for identifying the cellular targets of natural products[J]. Natural Product Reports, 2016, 33(5): 681 -708 . doi: 10.1039/C6NP00001K[19] QIN F, LI B, WANG H, MA S, LI J, LIU S, KONG L, ZHENG H, ZHU R, HAN Y, YANG M, LI K, JI X, CHEN P R. Linking chromatin acylation mark-defined proteome and genome in living cells[J]. Cell, 2023, 186(5): 1 066-1 085. [20] JIANG S, LI H, ZHANG L, MU W, ZHANG Y, CHEN T, WU J, TANG H, ZHENG S, LIU Y, WU Y, LUO X, XIE Y, REN J. Generic diagramming platform (GDP): a comprehensive database of high-quality biomedical graphics[J]. Nucleic Acids Research, 2025, 53(D1): D1670 -D1676 . doi: 10.1093/nar/gkae973[21] COUKOS J S, LEE C W, PILLAI K S, LIU K J, MOELLERING R E. Widespread, reversible cysteine modification by methylglyoxal regulates metabolic enzyme function[J]. ACS Chemical Biology, 2023, 18(1): 91 -101 . doi: 10.1021/acschembio.2c00727[22] MONTGOMERY D C, SORUM A W, MEIER J L. Chemoproteomic profiling of lysine acetyltransferases highlights an expanded landscape of catalytic acetylation[J]. Journal of the American Chemical Society, 2014, 136(24): 8 669-8 676. [23] QIN W, ZHANG Y, TANG H, LIU D, CHEN Y, LIU Y, WANG C. Chemoproteomic profiling of itaconation by bioorthogonal probes in inflammatory macrophages[J]. Journal of the American Chemical Society, 2020, 142(25): 10 894-10 898. [24] KULKARNI R A, BAK D W, WEI D, BERGHOLTZ S E, BRINEY C A, SHRIMP J H, ALPSOY A, THORPE A L, BAVARI A E, CROOKS D R, LEVY M, FLORENS L, WASHBURN M P, FRIZZELL N, DYKHUIZEN E C, WEERAPANA E, MARSTON LINEHAN W, MEIER J L. A chemoproteomic portrait of the oncometabolite fumarate[J]. Nature Chemical Biology, 2019, 15(4): 391 -400 . doi: 10.1038/s41589-018-0217-y[25] KELLY B, O’NEILL L A J. Metabolic reprogramming in macrophages and dendritic cells in innate immunity[J]. Cell Research, 2015, 25(7): 771 -784 . doi: 10.1038/cr.2015.68[26] ARTS R J W, NOVAKOVIC B, TER HORST R, CARVALHO A, BEKKERING S, LACHMANDAS E, RODRIGUES F, SILVESTRE R, CHENG S C, WANG S Y, HABIBI E, GONÇALVES L G, MESQUITA I, CUNHA C, van LAARHOVEN A, van de VEERDONK F L, WILLIAMS D L, van der MEER J W M, LOGIE C, O’NEILL L A, DINARELLO C A, RIKSEN N P, van CREVEL R, CLISH C, NOTEBAART R A, JOOSTEN L A B, STUNNENBERG H G, XAVIER R J, NETEA M G. Glutaminolysis and fumarate accumulation integrate immunometabolic and epigenetic programs in trained immunity[J]. Cell Metabolism, 2016, 24(6): 807 -819 . doi: 10.1016/j.cmet.2016.10.008[27] LAMPROPOULOU V, SERGUSHICHEV A, BAMBOUSKOVA M, NAIR S, VINCENT E E, LOGINICHEVA E, CERVANTES-BARRAGAN L, MA X, HUANG S C, GRISS T, WEINHEIMER C J, KHADER S, RANDOLPH G J, PEARCE E J, JONES R G, DIWAN A, DIAMOND M S, ARTYOMOV M N. Itaconate links inhibition of succinate dehydrogenase with macrophage metabolic remodeling and regulation of inflammation[J]. Cell Metabolism, 2016, 24(1): 158 -166 . doi: 10.1016/j.cmet.2016.06.004[28] O’NEILL L A J, ARTYOMOV M N. Itaconate: the poster child of metabolic reprogramming in macrophage function[J]. Nature Reviews Immunology, 2019, 19(5): 273 -281 . doi: 10.1038/s41577-019-0128-5[29] ZHANG X, SCHALKWIJK C G, WOUTERS K. Immunometabolism and the modulation of immune responses and host defense: a role for methylglyoxal?[J]. Biochimica et Biophysica Acta (BBA)-Molecular Basis of Disease, 2022, 1 868(8): 166 425. [30] LEVY M J, MONTGOMERY D C, SARDIU M E, MONTANO J L, BERGHOLTZ S E, NANCE K D, THORPE A L, FOX S D, LIN Q, ANDRESSON T, FLORENS L, WASHBURN M P, MEIER J L. A systems chemoproteomic analysis of acyl-CoA/protein interaction networks[J]. Cell Chemical Biology, 2020, 27(3): 322 -333 . doi: 10.1016/j.chembiol.2019.11.011[31] YANG F, JIA G, GUO J, LIU Y, WANG C. Quantitative chemoproteomic profiling with data-independent acquisition-based mass spectrometry[J]. Journal of the American Chemical Society, 2022, 144(2): 901 -911 . doi: 10.1021/jacs.1c11053[32] UCHINOMIYA S, NAGAURA T, WEBER M, MATSUO Y, ZENMYO N, YOSHIDA Y, TSURUTA A, KOYANAGI S, OHDO S, MATSUNAGA N, OJIDA A. Fluorescence-based detection of fatty acid β-oxidation in cells and tissues using quinone methide-releasing probes[J]. Journal of the American Chemical Society, 2023, 145(14): 8 248-8 260. [33] KAJIWARA K, OSAKI H, GRESSIES S, KUWATA K, KIM J H, GENSCH T, SATO Y, GLORIUS F, YAMAGUCHI S, TAKI M. A negative-solvatochromic fluorescent probe for visualizing intracellular distributions of fatty acid metabolites[J]. Nature Communications, 2022, 13(1): 2 533. [34] WU Z, HE K, CHEN Y, LI H, PAN S, LI B, LIU T, XI F, DENG F, WANG H, DU J, JING M, LI Y. A sensitive GRAB sensor for detecting extracellular ATP in vitro and in vivo[J]. Neuron, 2022, 110(5): 770 -782 . doi: 10.1016/j.neuron.2021.11.027[35] TAMIMA D U, SARKAR D S, ISLAM D M R, SHIL A, KIM D K H, REO D Y J, JUN D Y W, BANNA H, LEE S, AHN P K H. A small-molecule fluorescence probe for nuclear ATP[J]. Angewandte Chemie International Edition, 2023, 62(15): e202300580. doi: 10.1002/anie.202300580 [36] LI T, WANG A, ZHANG Y, CHEN W, GUO Y, YUAN X, LIU Y, GENG Y. Chemoproteomic profiling of signaling metabolite fructose-1,6-bisphosphate interacting proteins in living cells[J]. Journal of the American Chemical Society, 2024, 146(22): 15 155-15 166. [37] DIXIT A, JOSE G P, SHANBHAG C, TAGAD N, KALIA J. Metabolic labeling-based chemoproteomics establishes choline metabolites as protein function modulators[J]. ACS Chemical Biology, 2022, 17(8): 2 272-2 283. [38] HURBEN A K, ERBER L N, TRETYAKOVA N Y, DORAN T M. Proteome-wide profiling of cellular targets modified by dopamine metabolites using a bio-orthogonally functionalized catecholamine[J]. ACS Chemical Biology, 2021, 16(11): 2 581-2 594. [39] ZHAO X, STEIN K R, CHEN V, GRIFFIN M E, LAIRSON L L, HANG H. Chemoproteomics reveals microbiota-derived aromatic monoamine agonists for GPRC5A[J]. Nature Chemical Biology, 2023, 19(10): 1 205-1 214. [40] JI Y, SUN L, CHEN Y, QIN P, XUAN P. Sirtuin-derived covalent binder for the selective recognition of protein crotonylation[J]. Angewandte Chemie International Edition, 2022, 61(31): e202205522. doi: 10.1002/anie.202205522 [41] DONG H, ZHAI G, CHEN C, BAI X, TIAN S, HU D, FAN E, ZHANG K. Protein lysine de-2-hydroxyisobutyrylation by CobB in prokaryotes[J]. Science Advances, 2019, 5(7): eaaw6703. doi: 10.1126/sciadv.aaw6703 [42] CHEN C, CHEN C, WANG A, JIANG Z, ZHAO F, LI Y, HAN Y, NIU Z, TIAN S, BAI X, ZHANG K, ZHAI G. ENL reads histone beta-hydroxybutyrylation to modulate gene transcription [J]. Nucleic Acids Res, 2024, 52(17): 10 029-10 039. [43] HICKS K G, CLUNTUN A A, SCHUBERT H L, HACKETT S R, BERG J A, LEONARD P G, AJALLA ALEIXO M A, ZHOU Y, BOTT A J, SALVATORE S R, CHANG F, BLEVINS A, BARTA P, TILLEY S, LEIFER A, GUZMAN A, AROK A, FOGARTY S, WINTER J M, AHN H C, ALLEN K N, BLOCK S, CARDOSO I A, DING J, DREVENY I, GASPER W C, HO Q, MATSUURA A, PALLADINO M J, PRAJAPATI S, SUN P, TITTMANN K, TOLAN D R, UNTERLASS J, van DEMARK A P, VANDER HEIDEN M G, WEBB B A, YUN C H, ZHAO P, WANG B, SCHOPFER F J, HILL C P, NONATO M C, MULLER F L, COX J E, RUTTER J. Protein-metabolite interactomics of carbohydrate metabolism reveal regulation of lactate dehydrogenase[J]. Science, 2023, 379(6 636): 996-1 003. [44] 叶明亮, 李柯佳. 探测能量状态发生变化的蛋白或配体与蛋白亲和力的方法: 中国, CN117665082A[P]. 2024-03-08. [45] LI K, CHEN S, WANG K, WANG Y, XUE L, YE Y, FANG Z, LYU J, ZHU H, LI Y, YU T, YANG F, ZHANG X, GUO S, RUAN C, ZHOU J, WANG Q, DONG M, LUO C, YE M. A peptide-centric local stability assay enables proteome-scale identification of the protein targets and binding regions of diverse ligands[J]. Nature Methods, 2025, 22(2): 278 -282 . doi: 10.1038/s41592-024-02553-7[46] RUAN C, WANG Y, ZHANG X, LYU J, ZHANG N, MA Y, SHI C, QU G, YE M. Matrix thermal shift assay for fast construction of multidimensional ligand-target space[J]. Analytical Chemistry, 2022, 94(17): 6 482-6 490. [47] PANTOLIANO M W, PETRELLA E C, KWASNOSKI J D, LOBANOV V S, MYSLIK J, GRAF E, CARVER T, ASEL E, SPRINGER B A, LANE P, SALEMME F R. High-density miniaturized thermal shift assays as a general strategy for drug discovery[J]. SLAS Discovery, 2001, 6(6): 429 -440 . doi: 10.1177/108705710100600609[48] LUCK K, SHEYNKMAN G M, ZHANG I, VIDAL M. Proteome-scale human interactomics[J]. Trends in Biochemical Sciences, 2017, 42(5): 342 -354 . doi: 10.1016/j.tibs.2017.02.006[49] SOWMYA G, BREEN E J, RANGANATHAN S. Linking structural features of protein complexes and biological function[J]. Protein Science, 2015, 24(9): 1 486-1 494. [50] VANDEREYKEN K, SIFRIM A, THIENPONT B, VOET T. Methods and applications for single-cell and spatial multi-omics[J]. Nature Reviews Genetics, 2023, 24(8): 494 -515 . doi: 10.1038/s41576-023-00580-2 -

 首页
首页 登录
登录 注册
注册
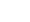


 下载:
下载: